Archei
Archei
I rapporti filogenetici tra i diversi tipi di procarioti (gli organismi composti da cellule semplici senza nucleo, risalenti a circa 3,5 miliardi di anni fa) e tra questi e gli eucarioti (organismi composti da cellule con il nucleo, datati a circa 1,5 miliardi di anni fa) hanno costituito un enigma insolubile prima che fossero messe a punto le tecniche per il sequenziamento del DNA. Nella percezione generale, i procarioti erano considerati un gruppo indivisibile, mentre gli eucarioti sembravano essere una filiazione tardiva di uno dei diversi phyla batterici noti, derivati forse dai micoplasmi. Questi ultimi, essendo privi di parete cellulare, avrebbero infatti più facilmente acquisito (per endocitosi) altri procarioti precursori dei mitocondri e cloroplasti dando origine alle cellule 'internamente compartimentate' ‒ con nucleo e organelli ‒ tipiche degli eucarioti.
Questa rappresentazione della storia e della diversità dei viventi ha subito una svolta imprevista nella seconda metà degli anni Settanta del Novecento in seguito allo sviluppo di tecnologie per la determinazione delle sequenze nucleotidiche dei geni (e implicitamente delle sequenze amminoacidiche delle proteine corrispondenti), e all'idea, dovuta a Linus Pauling ed Emil Zuckerkandl, che le macromolecole informazionali (DNA, RNA, proteine) sono documenti 'storici' la cui consultazione permette di percorrere a ritroso la storia naturale dei viventi. In sostanza, geni (o proteine) omologhi di specie diverse hanno sequenze simili ma non identiche. Le identità (condivisioni di uno stesso tipo di monomero in posizioni corrispondenti di due o più sequenze omologhe) riflettono la sostanziale inviolabilità di 'motivi' di sequenza essenziali per la funzionalità del prodotto genico. Le diversità riflettono la fissazione e il progressivo accumulo delle mutazioni selettivamente neutrali che modificano, nel tempo, le sequenze geniche. Per il carattere additivo delle mutazioni neutrali, sequenze omologhe di due qualsiasi specie sono tanto più dissimili quanto maggiore è l'intervallo di tempo trascorso dalla loro separazione da una sequenza ancestrale appartenuta a un più recente antenato comune delle specie in esame, e la quantificazione delle diversità di sequenza fornisce una chiave per la ricostruzione delle relazioni genealogiche tra le specie alle quali le sequenze appartengono.
Un corollario implicito è che sequenze altamente conservate di geni (o proteine) universali permettono di risalire a divergenze primigenie e, in prospettiva, di ricostruire quell'albero della vita preconizzato da Charles Darwin nella sua opera On the origin of species (1859). In quest'ambito di studi, le sequenze, evolutivamente molto stabili, dei geni che codificano per gli RNA della sottounità minore del ribosoma (Ssu-RNA, una stringa informazionale di 1500-2000 nucleotidi) hanno rappresentato la 'stele di Rosetta' dell'evoluzione cellulare: lo strumento che ha vanificato il dogma dell'indivisibilità dei procarioti e dell'origine degli eucarioti dai batteri conosciuti.
Albero filogenetico degli archei
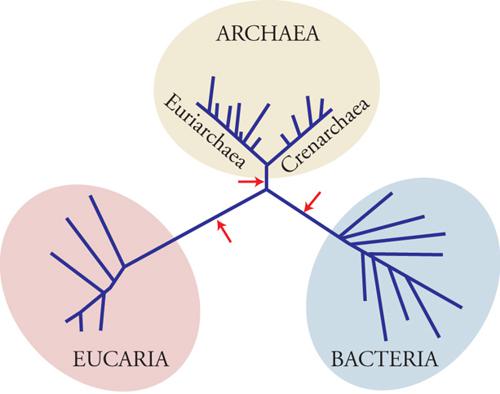
In uno studio conciso e memorabile, Carl Woese e George Fox dell'Università di Urbana (Illinois) dimostravano ‒ nel 1977 ‒ che le sequenze degli Ssu-RNA (Small subunit ribosomal RNA) dei procarioti metanogeni (un gruppo di microrganismi anaerobi obbligati, cioè che crescono solo in assenza di ossigeno) erano tanto 'distanti' da quelle degli altri batteri quanto queste lo erano da quelle degli eucarioti. Ciò implicava che il mondo apparentemente unitario dei procarioti comprendeva due domini filogeneticamente distinti, e che tutti i viventi appartenevano all'una o all'altra di tre linee di discendenza rigidamente segregate (tre 'cladi' o domini primari): gli eucarioti (dominio Eucaria), i batteri tradizionali (dominio Bacteria) e un terzo dominio di vita cellulare costituito dagli organismi inizialmente denominati 'archeobatteri' e ‒ tredici anni più tardi ‒ 'archei' (dominio Archaea) esemplificati, nello studio iniziale, dai soli metanogeni. In base alle similarità di sequenza degli Ssu-RNA il termine 'procariote' risultava quindi destituito di qualsiasi connotazione filogenetica e definisce, attualmente, solo un modello di organizzazione cellulare diverso dal modello eucariotico.
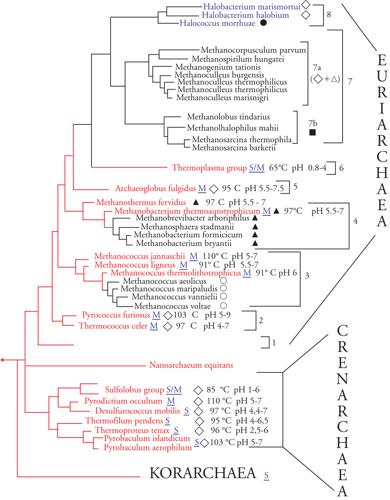
Nel breve giro di tre anni dalla pubblicazione dello studio originale una pletora di microrganismi prevalentemente chemioautotrofi e chemiolitoautotrofi, naturalmente adattati a condizioni estreme di temperatura, pH e salinità, confluiva nel gruppo iniziale dei metanogeni configurando gli archei come un dominio variegato (fig. 2 e Tav. I) che include: (a) i metanogeni (sia mesofili che ipertermofili) che usano strategie energetiche basate su varianti della reazione generale 4H2+CO2 →CH4+2H2O, che a partire da idrogeno e anidride carbonica producono metano e acqua; (b) un gruppo di procarioti aerobi ‒ gli alofili estremi ‒ che prosperano in ambienti ipersalini (saline solari, Mar Morto, Great Salt Lake nello Utah) incompatibili con la sopravvivenza di qualsiasi altro organismo terrestre, utilizzando la classica reazione di riduzione dell'ossigeno molecolare (2[H]+1/2O2→H2O); (c) i 'termofili estremi zolfo-dipendenti', un gruppo di procarioti prevalentemente, ma non esclusivamente, anaerobi che vivono a temperature comprese tra 85 e 113 °C nelle acque solfatariche di aree vulcaniche, in sorgenti termali terrestri o sottomarine e sulle pareti dei 'fumatori neri' distribuiti nella profondità dei fondali oceanici, sfruttando strategie energetiche basate sull'ossidazione dello zolfo elementare
S0+3/2O2+H2O→H2SO4
oppure sulla riduzione dello zolfo ad acido solfidrico mediante l'una o l'altra di tre reazioni principali
2[H]+S0→H2S; H2+S0→H2S;
4H2+S2O32‒+2H+2H2S+3H2O.
In base alle similarità di sequenza degli Ssu-RNA gli archei 'coltivabili' in laboratorio appartenevano all'una o all'altra di due divisioni (o regni) principali: 'crenarcheoti' ed 'euriarcheoti'. I crenarcheoti costituiscono un gruppo omogeneo di archei termofili, termoacidofili o ipertermofili suddiviso negli ordini Sulfolobales, Thermoproteales e Pyrodictiales, che comprendono principalmente anaerobi obbligati zolfo-dipendenti e un numero limitato di aerobi obbligati (il genere Aeropyrum) o facoltativi (diversi membri del genere Pyrobaculum).
Gli euriarcheoti costituiscono un gruppo eterogeneo di specie sia mesofile che ipertermofile che comprende: (a) gli alofili estremi (suddivisi negli ordini Halococcales e Halobacteriales); (b) tutti i metanogeni ‒ sia mesofili che ipertermofili ‒ rappresentati dal genere Methanopyrus (temperatura ottimale di crescita 113-115 °C) e dagli ordini Methanococcales, Methanobacteriales, Methanomicrobiales (questi ultimi suddivisi nelle famiglie Methanosarcinaceae e Methanospirillaceae); (c) diversi tipi di micoplasmi zolfo-dipendenti sia aerobi che anaerobi confinati in ambienti caldo-acidi (pH 1,0-1,5) che formano l'ordine Thermoplasmales; (d) l'ordine Thermococcales, che comprende un gruppo di anaerobi obbligati ipertermofili e zolfo-dipendenti (il genere Thermoccoccus e diversi membri del genere Pyrococcus); (e) l'ordine Archaeoglobales rappresentato finora dal solo ipertermofilo anaerobio zolfo-riducente Archaeoglobus fulgidus. Il genere Methanopyrus costituisce la radiazione più profonda del regno mentre gli ordini Methanomicrobiales e Halobacteriales/Halococcales rappresentano le divergenze più recenti suggerendo, implicitamente, un'origine degli alofili da un precursore metanogenico.
La struttura tripartita dell'albero filogenetico universale e l'ordine di divergenza dei phyla nel dominio Archaea sono stati confermati dall'analisi delle sequenze amminoacidiche di singole proteine, dal confronto delle sequenze 'concatenate' di un notevole numero di proteine universali (circa 5000 posizioni amminoacidiche) e da confronti dei contenuti genici dei genomi interamente sequenziati. Inoltre, molto recentemente il dominio degli archei si è arricchito di un nuovo phylum di collocazione filogenetica ancora incerta (i 'nanoarcheoti') rappresentativo di 'minicellule' (circa 400 nm di diametro) provviste di un genoma minimo (meno di 0,5 ∙ 106 coppie di basi) che conducono un'esistenza endosimbiontica come parassiti obbligati di archei anaerobi ipertermofili.
A tutt'oggi i genomi di ventisei archei rappresentativi sono stati completamente sequenziati e le relative sequenze sono pubblicamente accessibili nella rete globale (www.ncbi.nlm.nih.gov). Le specie coltivabili in laboratorio, tuttavia, non esauriscono la biodiversità degli archei. Intorno alla metà degli anni Ottanta del Novecento è risultato evidente che il 99% delle specie microbiche presenti nei 'campioni ambientali' sfugge ai classici metodi di arricchimento microbiologico. Questa grave limitazione è stata in parte superata grazie allo sviluppo e all'impiego di sonde di DNA (brevi sequenze di 17-24 coppie di basi) che ‒ confrontate con il DNA totale estratto dai campioni ambientali ‒ formano ibridi specifici con i geni degli Ssu-RNA dei soli archei. I DNA dei geni selezionati dalle sonde potevano essere amplificati mediante PCR (Polymerase Chainreaction) e sequenziati. Eventuali sequenze geniche ignote venivano sottoposte a un'analisi filogenetica per stabilire l'esatta collocazione, tra gli archei, degli organismi non coltivabili ai quali appartenevano le 'sequenze ambientali'.
Questa strategia sperimentale ha permesso di stabilire che ‒ oltre a diverse specie ipertermofiliche (derivate in gran parte da una sorgente termale del Parco Nazionale di Yellowstone, Wyoming) e psicrofile (derivate dal plancton oceanico) collocate a diversi livelli tra i crenarcheoti e gli euriarcheoti conosciuti ‒ il dominio Archaea contiene anche un nuovo gruppo di archei ipertermofili, che emerge dal tronco comune degli archei prima della biforcazione euriarcheoti/crenarcheoti e rappresenta un terzo regno di archei, i 'korarcheoti' (dal greco kóre, 'giovane'), le cui uniche proprietà conosciute sono quelle desumibili dalle condizioni fisico-chimiche esistenti nella nicchia ecologica dalla quale il campione è stato prelevato.
Caratteristiche biochimiche degli archei
Alla peculiarità filogenetica degli archei corrispondono singolari proprietà biochimiche nella struttura e nella composizione degli involucri cellulari e nella complessità strutturale degli apparati di espressione genica e di duplicazione del DNA.
Membrane cellulari
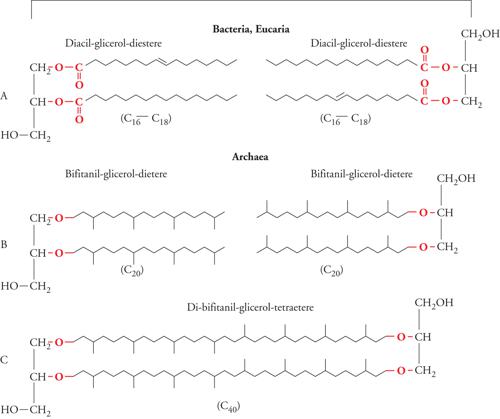
La struttura chimica delle membrane costituisce uno dei più robusti supporti biochimici in favore dell'identità filogenetica degli archei come dominio monofiletico-olofiletico. Le membrane cellulari degli eucarioti e dei batteri sono assemblaggi di glicerolipidi (più abbondanti) e di sfingolipidi. Questi ultimi sono costituiti ‒ nella loro componente apolare ‒ da un amminoalcol (la sfingosina) e un acido grasso uniti da un legame ammidico (NH−CO). I glicerolipidi sono (nella componente apolare) diacil-glicerol-diesteri (fig. 3A) formati per esterificazione di due acidi grassi (C16-C18) ai carboni 1 e 2 del glicerolo nella configurazione stereochimica 1,2-sn.
Le membrane degli archei possiedono lipidi di tipo inedito, la cui componente apolare è costituita esclusivamente e tipicamente dall'uno o dall'altro di due tipi di isopranil-glicerol-eteri: i bifitanil-glicerol-dieteri (archeolo) e i di-bifitanil-glicerol-tetraeteri (caldarcheolo). Nell'archeolo (fig. 3B) i gruppi alcolici degli atomi di carbonio in posizione 2 e 3 del glicerolo nella configurazione stereochimica 2,3-sn sono legati con legami di tipo etere ad altrettante catene di diidrofitolo (fitanolo), un alcol di una catena ramificata (isoprenoide) a 20 atomi di carbonio. I di-bifitanil-glicerol-tetraeteri si formano per dimerizzazione di due molecole di archeolo mediante saldatura 'testa a testa' delle catene di fitolo (fig. 3C); l'unità idrofobica elementare della membrana è quindi costituita da due catene isoprenoidi a 40 atomi di carbonio unite mediante legame etere agli atomi di carbonio in posizione 2 e 3 di due molecole di glicerolo.
Infine, in alcuni archei (ordine Sulfolobales), in seguito a processi di ciclizzazione interna, le catene C40 dei di-bifitanil-glicerol-tetraeteri contengono da due a quattro anelli ciclopentenici. In generale, quattro elementi sono diagnostici e distintivi degli archei: l'assenza di lipidi privi di glicerolo; la natura isoprenoide della catena alifatica; la struttura stereochimica 2,3-sn del glicerolo; il legame (di tipo etere) del glicerolo con le catene alifatiche.
Pareti cellulari
Tra gli eucarioti una parete cellulare rigida è presente solo nei funghi (chitina) e nelle piante verdi (cellulosa). I batteri, Gram positivi e Gram negativi, con la sola eccezione degli ordini Micoplasmales e Planctomicetales, sono dotati di una parete cellulare rigida (sacculus) di mureina (peptidoglicano) formata da strati multipli di un'estesa rete di catene polisaccaridiche parallele (filamenti di glicano) unite a intervalli regolari da legami covalenti. I filamenti di glicano sono costituiti da unità alternate di N-acetil-D-glucosammina (D-GlcNAc) e acido N-acetil-murammico (D-MurNAc), un monosaccaride complesso a 9 atomi di carbonio) unite da legami β‒1,4 glicosidici. Al carbonio 3′ di ogni residuo di acido N-acetil-murammico è legata una catena laterale tetra- o pentapeptidica tipicamente costituita da D-amminoacidi; i glicani adiacenti sono uniti da brevi 'ponti' peptidici che collegano trasversalmente, con legami covalenti, le 'appendici' tetrapeptidiche. A differenza dei batteri, gli archei presentano una notevole varietà di involucri cellulari esterni.
In particolare, i crenarchaeoti e la maggior parte degli euriarcheoti possiedono, invece di un sacculus rigido, l'una o l'altra di tre strutture parietali alternative: (a) una semplice 'tunica' proteinacea; (b) un mosaico regolare (S-layer) di subunità proteiche glicosilate (S-layer glicoproteico); (c) un mosaico regolare di subunità proteiche non glicosilate (S-layer proteico).
Un sacculus è presente solo in alcuni euriarcheoti metanogeni e alofili; la struttura e la composizione chimica del sacculus sono tuttavia altamente diversificate all'interno del dominio (Tav. I). In particolare, in alcuni euriarcheoti (Methanobacteriales e Methanopyrus) il sacculus è tipicamente costituito da pseudomureina, un peptidoglicano strutturalmente simile alla mureina ma distinto da quest'ultima per alcuni dettagli chimici: la sostituzione dell'acido N-acetil-murammico con un monosaccaride più complesso (l'acido talosamminouronico); l'assenza di D-amminoacidi nell'appendice tetrapeptidica dell'acido talosamminouronico; il tipo di legame (β-1,3glicosidico invece che β-1,4glicosidico) che unisce i residui monosaccaridici della catena polisaccaridica.
Negli euriarcheoti dell'ordine Halococcales il sacculus è, invece, un eteropolisaccaride sulfonato caratterizzato dalla presenza di due monosaccaridi di tipo inedito: un acido N-acetilgulosaminouronico (GulNA) e una glucosammina N-glicil sostituita. Infine, negli euriarcheoti della famiglia Metanosarcinaceae il sacculus è costituito da un polisaccaride complesso ‒ la metanocondroitina ‒ chimicamente molto simile al condroitin-solfato del tessuto connettivo degli animali.
Trascrizione negli archei
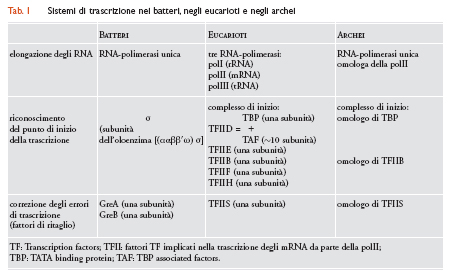
Come i batteri, gli archei possiedono un unico tipo di RNA-polimerasi e presentano un'organizzazione operonica dei geni funzionalmente correlati. Il loro apparato di trascrizione, tuttavia, è sorprendentemente simile al sistema di trascrizione degli eucarioti, del quale è in effetti una versione semplificata (tab. 1).
Nei batteri la sintesi dei diversi tipi di RNA è affidata a un unico tipo di RNA-polimerasi DNA-dipendente costituita da un nucleo pentamerico (subunità ααββ′ω) e da una subunità monomerica labilmente associata (σ) che conferisce al nucleo (ααββ′ω) la capacità di riconoscere specificamente, sia in vivo che in vitro, due brevi sequenze (6 coppie di basi) situate sul promotore a 10 e 35 coppie di basi a monte del punto di inizio della trascrizione. Il complesso [(ααββ′ω)σ] è in grado di trascrivere DNA in modo specifico in vitro in assenza di altri fattori, ed è inibito dall'antibiotico rifampicina che inattiva, selettivamente, la subunità σ. La fedeltà della trascrizione durante la fase di elongazione è affidata a due 'fattori di ritaglio' ‒ le proteine GreA e GreB (molto simili tra loro) ‒ che attivano una intrinseca capacità della polimerasi di rimuovere nucleotidi non pertinenti aggiunti erroneamente all'estremità 3′-OH delle catene di RNA in corso di allungamento e assicurano il reinizio della trascrizione a partire dal 'nuovo' terminale 3′-OH.
L'apparato di trascrizione degli eucarioti è decisamente più ricco di componenti: (a) le cellule eucariotiche contengono tre diverse RNA-polimerasi DNA-dipendenti (polI, polII e polIII) specializzate nella trascrizione di classi distinte di geni (rispettivamente, i geni per gli rRNA, gli mRNA e i tRNA; (b) le tre polimerasi (nessuna delle quali è sensibile alla rifampicina) sono proteine di elevatissima complessità strutturale, costituite da 11-17 subunità; di queste, cinque sono omologhe delle subunità α, β, β′ e ω della polimerasi batterica mentre le rimanenti sono omologhe di subunità condivise da tutte le tre polimerasi eucariotiche; (c) a differenza della polimerasi batterica, le polimerasi degli eucarioti non sono in grado di trascrivere DNA in vitro in assenza di proteine accessorie: l'inizio della trascrizione (esemplificato dalla polII) richiede l'interazione della RNA-polimerasi con almeno quindici proteine aggiuntive (componenti dei 'fattori generali di trascrizione' denominati TFIID, TFIIB TFIIF TFIIE e TFIIH) essenziali per l'ancoraggio della polII in corrispondenza di una regione del promotore che comprende una sequenza di consenso TATAAT (TATA box) 'centrata' a circa 25 coppie di basi a monte del punto di inizio della trascrizione, e una sequenza più breve denominata BRE (TFIIB responsive element) situata immediatamente a monte della precedente. Il fattore TFIID ‒ la chiave di volta del sistema trascrizionale eucariotico ‒ è un assemblaggio proteico comprendente una subunità TBP (TATA binding protein) che riconosce specificamente la TATA box, e circa 10 polipeptidi associati alla TBP e collettivamente designati con l'acronimo TAF (TBP associated factors). Il processo inizia con il legame (mediato dalla subunità TBP) del complesso TFIID al promotore; al complesso [TATA box/TFIID] si aggiungono quindi ‒ nell'ordine ‒ il fattore TFIIB che riconosce specificamente la sequenza BRE (dalla quale dipende il corretto orientamento del complesso di inizio rispetto al punto di inizio della trascrizione),l'RNA-polimerasi II, e infine i fattori TFIIF, TFIIE e TFIIH. La fosforilazione di alcuni residui C-terminali della subunità più grande della polimerasi promuove il distacco dell'enzima dall'assemblaggio di fattori generali di trascrizione e innesca il reclutamento, sul corpo della polimerasi, di altre proteine (fattori di elongazione) necessarie per la trascrizione degli mRNA. La fedeltà della trascrizione, affidata nei batteri ai due fattori di ritaglio GreA e GreB, è demandata, negli eucarioti, a un diverso 'fattore di ritaglio' TFIIS che (come GreA e GreB) attiva una latente capacità della polimerasi di rimuovere nucleotidi non pertinenti erroneamente aggiunti all'estremità 3′-OH delle catene di RNA in corso di allungamento.
L'apparato trascrizionale degli archei è una variante semplificata (o ancestrale) dell'apparato eucariotico. Come i batteri, gli archei possiedono un unico tipo di RNA-polimerasi DNA-dipendente. A differenza dei batteri tuttavia, l'RNA-polimerasi degli archei è costituita da non meno di dieci subunità che presentano un'elevata omologia di sequenza con le subunità della RNA-polimerasi II degli eucarioti; la sola differenza degna di rilievo è rappresentata dalla subunità più pesante che, negli archei, è amputata della regione C-terminale implicata, negli eucarioti, nella fosforilazione dell'oloenzima. La RNA-polimerasi degli archei (come accade anche negli eucarioti) non è in grado di stabilire un legame specifico con i promotori in assenza di fattori aggiunti. L'ancoraggio dell'enzima in corrispondenza del punto di inizio della trascrizione richiede infatti una proteina TBP che presenta un'elevata omologia di sequenza con la proteina TBP degli eucarioti, e una proteina designata TFB che presenta un'elevata omologia di sequenza con il fattore eucariotico TFIIB. Come negli eucarioti, la proteina TBP degli archei riconosce specificamente una TATA box situata a circa 25 paia di basi a monte del punto di inizio della trascrizione e, come negli eucarioti, il fattore TFB riconosce specificamente una sequenza simile alla sequenza BRE situata immediatamente a monte della TATA box. A differenza degli eucarioti, tuttavia, gli archei non contengono omologhi delle proteine TAF e dei fattori di trascrizione TFIIF, TFIIE e TFIIH. La formazione di un complesso di inizio della trascrizione richiede pertanto le sequenze adiacenti TATA box e BRE, una TBP, un fattore TFB omologo del fattore eucariotico TFIIB, e una RNA-polimerasi simile per complessità e composizione alla polimerasi II eucariotica. Infine, la fedeltà della trascrizione, controllata nei batteri dai fattori di ritaglio GreA e GreB, è affidata, negli archei, a un fattore di ritaglio designato TFS omologo, per sequenza e modalità di azione, del fattore di ritaglio eucariotico TFIIS.
Apparato di traduzione
Come per la trascrizione, l'analisi del sistema di traduzione degli archei ha rivelato inattese similarità con il sistema eucariotico, ma anche idiosincrasie tipiche del dominio Archaea. I ribosomi degli archei (70S) contengono rRNA di dimensioni simili a quelle dei batteri (RNA 16S, circa 1500 nucleotidi; RNA 23S, circa 3000 nucleotidi) ma differiscono dagli omologhi batterici per un complemento proteico più abbondante che comprende proteine 'condivise' con i ribosomi 8OS degli eucarioti.
Trascurando per semplicità descrittiva la ripartizione delle diverse proteine tra le due subunità del ribosoma, di 102 diversi tipi di proteine ribosomiali finora rivelate dal sequenziamento dei DNA genomici, i ribosomi dei batteri, degli archei e degli eucarioti contengono, rispettivamente, 57, 68 e 78 diverse proteine ribosomiali. Poiché 34 proteine sono componenti universali presumibilmente presenti nel più recente progenitore comune dei tre domini (ed ereditati da esso), batteri, archei ed eucarioti possiedono rispettivamente 23, 34 e 44 proteine ribosomiali in più del corredo comune. Sorprendentemente, le 23 proteine dei ribosomi batterici che eccedono il corredo comune non sono rappresentate (ossia non hanno omologhi) negli altri due domini, mentre le 34 proteine dei ribosomi degli archei sono rappresentate interamente nel corredo proteico dei ribosomi degli eucarioti che avrebbero così acquisito ‒ rispetto agli archei ‒ 11 nuovi tipi di proteine ribosomiali.
Deviazioni dal paradigma descritto sono state evidenziate solo nelle ramificazioni più recenti del dominio Archaea (Thermoplasmales, Methanobacteriales, Methanomicrobiales e alofili estremi) e sono ragionevolmente interpretabili come semplificazioni di un più abbondante corredo proteico originale durante le fasi tardive dell'evoluzione degli euriarcheoti. In accordo con la complessità del loro corredo proteico, i ribosomi della maggior parte degli archei possiedono diverse caratteristiche morfologiche condivise con i ribosomi eucariotici (un 'becco' e due 'lobi' della subunità minore; due lobi della subunità maggiore) e presentano uno spettro variegato di sensibilità agli antibiotici inibitori della sintesi proteica, essendo insensibili alla maggior parte degli antibiotici inibitori dei ribosomi batterici e sensibili a diversi inibitori dei ribosomi 80S (eucarioti).
Oltre alle caratteristiche dei loro ribosomi, gli archei condividono con gli eucarioti significative similarità riguardanti il meccanismo di inizio della traduzione. La reazione di inizio negli eucarioti è decisamente più complessa che nei batteri. In particolare, in questi ultimi l'inizio è affidato a tre sole proteine monomeriche (IF1, IF2 e IF3); il fattore IF2 rappresenta la chiave di volta del sistema promuovendo il legame GTP-dipendente di un formil-metionil-tRNA (f-Met-tRNAfMet ) al codone di inizio AUG posizionato sul sito P della subunità minore del ribosoma, mentre i fattori IF1 e IF3 svolgono ruoli ancillari di controllo dell'efficienza del processo. Negli eucarioti lo stesso processo è affidato a un vasto repertorio di fattori proteici, dalla nomenclatura a tutt'oggi impervia che comprende (a) un fattore eIF-4F (o cap-binding factor) che assicura lo svolgimento del filamento di mRNA facilitando l'interazione mRNA-ribosoma; (b) un fattore eIF-1 implicato nel riconoscimento del codone di inizio (AUG); (c) i fattori eIF-2 (una proteina etero-trimerica; subunità α, β, γ), eIF-5 ed eIF-2B (una proteina eteropentamerica; subunità α, β, γ, δ, ε) che promuovono il legame GTP-dipendente, del metionil-tRNA iniziatore (Met-tRNAiMet) al codone AUG sul sito P della subunità ribosomiale 40S. Il fattore trimerico eIF-2 è la chiave di volta del processo svolgendo, negli eucarioti, lo stesso ruolo fondamentale giocato nei batteri dal fattore monomerico IF2.
Orbene, come per la trascrizione, anche il sistema d'inizio della traduzione negli archei è una versione semplificata (o ancestrale) del sistema eucariotico: come gli eucarioti, gli archei usano, come amminoacido iniziatore, una metionina non formilata legata a uno specifico tRNA iniziatore (tRNAiMet) distinto da quello usato per la traduzione dei codone AUG interni e possiedono: (a) un omologo del fattore eIF-1A implicato, negli eucarioti, nel riconoscimento del codone d'inizio; (b) un omologo del fattore trimerico eIF-2 e una versione semplificata (priva delle subunità δ e γ) del fattore eteropentamerico eIF2-B. A differenza degli eucarioti gli archei non possiedono omologhi del fattore monomerico eIF-5. Gli archei, infine, differiscono dai batteri, e sono simili agli eucarioti, anche riguardo al sistema di terminazione della traduzione. Nei batteri il distacco dal ribosoma della catena polipeptidica completata è affidato a due fattori di terminazione (RF1 e RF2) che riconoscono codoni di terminazione diversi (rispettivamente UAA/UAG, e UAA/UGA) mentre gli eucarioti dispongono di un unico fattore ‒ eRF1 ‒ non omologo dei fattori di terminazione batterici. Gli archei usano come gli eucarioti un unico fattore di terminazione ‒ aRF1 ‒ omologo del fattore eucariotico eRF1.
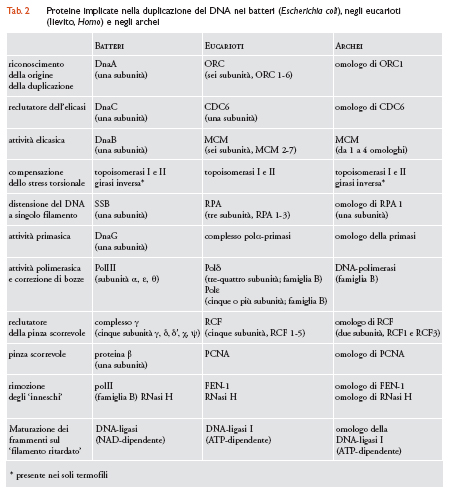
Duplicazione del DNA
Come tutti i procarioti gli archei hanno un unico cromosoma circolare, un'unica origine della duplicazione e una replicazione bidirezionale del DNA; ciononostante, il loro apparato di duplicazione è straordinariamente simile a quello degli eucarioti.
Nei batteri (assunti qui come paradigma di riferimento) la duplicazione del DNA richiede l'operazione sequenziale di diverse proteine (tab. 2). In particolare: (a) una proteina monomerica DnaA, 4-5 copie della quale si associano a specifiche sequenze ripetute (DnaA boxes) nella regione oriC del cromosoma; (b) una proteina monomerica DnaC (o 'reclutatore dell'elicasi') copie multiple della quale si aggiungono al complesso oriC-DnaA; (c) una proteina anulare esamonomerica DnaB ad attività elicasica, 'arruolata' sul DNA dalla precedente, che circonda la doppia elica e promuove lo svolgimento ATP-dipendente dei due filamenti; (d) le proteine monomeriche SSB (Single stranded DNA binding proteins) che rivestono e mantengono estesi i due filamenti svolti dall'elicasi; (e) la proteina 'monomerica' DnaG ‒ una primasi che si associa all'elicasi e sintetizza brevi 'inneschi' di RNA man mano che la forca di replicazione avanza; (f ) una proteina eteropentamerica definita 'complesso γ' o 'reclutatore della pinza scorrevole' che si associa al DNA in corrispondenza del terminale 3′-OH dell'innesco; (g) la proteina β (o 'pinza scorrevole'), un omodimero anulare arruolato sul DNA dalla proteina precedente, che attiva e trattiene sul DNA la replicasi; (h) la DNA-polimerasi III (polIII, una replicasi eterotrimerica con attività catalitica e di correzione di bozze); (i) una DNA-polimerasi I (polI) e una RNasi H che presiedono alla digestione degli inneschi di RNA e alla loro sostituzione con segmenti di DNA; (l) una DNA-ligasi NAD-dipendente che unisce i brevi frammenti di DNA sul filamento ritardato del DNA. Infine, due topoisomerasi (topoisomerasi I e II) e una girasi inversa (presente nei soli termofili) sono adibite alla compensazione dello stress torsionale imposto alla doppia elica dall'azione dell'elicasi.
L'apparato di duplicazione degli archei è una versione semplificata (o ancestrale) dell'apparato eucariotico (tab.2). In particolare: (a) negli eucarioti la proteina DnaA è sostituita da un assemblaggio multiproteico ORC (Origin recognition complex) costituito da sei diverse proteine (denominate ORC 1-6) che si associano alle origini di duplicazione; negli archei la stessa funzione è espletata da un omologo della subunità più grande (ORC1) del complesso ORC; (b) il ruolo di 'reclutatore dell'elicasi' (la proteina DnaC batterica) è giocato, negli eucarioti, da una proteina monomerica CDC6 (Cell division cycle protein 6); un omologo della proteina CDC6 è anche usato dagli archei per la stessa funzione; (c) l'attività elicasica della proteina DnaB è demandata, negli eucarioti, a un assemblaggio anulare esamerico costituito da due copie di un eterotrimero i cui membri (subunità MCM4, MCM6, MCM7) appartengono a una famiglia proteica MCM 2-7 (Mini chromosome maintanance protein; subunità 2-7) che comprende sei polipeptidi molto simili tra loro (e probabili varianti di una stessa proteina ancestrale); un membro della famiglia MCM è usato dagli archei per assemblare un'elicasi anulare esamonomerica; (d) le proteine monomeriche SSB, che nei batteri mantengono distesi i due filamenti separati del DNA, sono sostituite, negli eucarioti, da un eterotrimero RPA (Replication protein A, subunità 1-3); gli archei impiegano, per la stessa funzione, la subunità più pesante (RPA1) dell'eterotrimero eucariotico; (e) la sintesi degli inneschi di RNA che nei batteri è affidata alla proteina monomerica DnaG, è assistita, negli eucarioti, da un complesso tetramerico DNA-polimerasi α-primasi (polα-pri) una subunità del quale, la proteina p48, sintetizza i brevi segmenti di RNA di un innesco misto RNA-DNA; gli archei usano per la sintesi degli inneschi un omologo della componente primasica del complesso polα-pri degli eucarioti; (f) il ruolo di 'reclutatore della pinza scorrevole' (complesso γ eteropentamerico dei batteri) è affidato, negli eucarioti, a un diverso complesso eteropentamerico designato RFC (Replication factor; subunità 1-5): gli archei usano come 'reclutatore della pinza' un complesso RFC eterodimerico, costituito dalle sole subunità RFC1 e RFC3; (g) la funzione di pinza scorrevole è espletata, negli eucarioti, dalla proteina eterotrimerica PCNA (Proliferating cell nuclear antigen), un assemblaggio anulare solo formalmente simile alla pinza scorrevole (l'omodimero β) dei batteri; gli archei usano come pinza scorrevole un eterotrimero strettamente omologo dell'eterotrimero PCNA; (h) la maturazione dei frammenti di DNA sul filamento ritardato, affidata nei batteri all'azione combinata delle proteine polI e RNasi H, è demandata negli eucarioti a una proteina FEN-1 (Flap endonuclease) e alla RNasi H; gli archei usano per la stessa funzione omologhi delle proteine FEN-1 e RNasi H; (i) la saldatura dei frammenti di DNA sul filamento ritardato del DNA, affidata nei batteri alla DNA-ligasi NAD-dipendente, è demandata, negli eucarioti, a una diversa (non omologa) DNA-ligasi I ATP-dipendente; gli archei impiegano, per la stessa funzione, un omologo della ligasi ATP-dipendente eucariotica; (l) lo stress torsionale della doppia elica viene compensato, negli eucarioti, da due topoisomerasi omologhe delle topoisomerasi I e II dei batteri; gli archei possiedono omologhi delle due topoisomerasi e quelli ipertermofili hanno, inoltre, una girasi inversa (inizialmente identificata nell'archeo Sulfolobus acidocaldarius) che introduce superavvolgimenti positivi del DNA ed è presente anche nei membri termofili del dominio Bacteria; (m) negli eucarioti le funzioni delle DNA-polimerasi batteriche polIII e polI sono esercitate da tre polimerasi replicative; la DNA-polimerasi α (che fa parte del già citato complesso polα-pri) e le DNA-polimerasi δ (polδ) ed ε (polε) molto simili tra loro e funzionalmente intercambiabili (per questo talvolta indicate come polε/δ); le tre polimerasi eucariotiche α, δ ed ε appartengono a una famiglia di sequenze (la famiglia B) che ha come 'prototipo' la polimerasi II dei batteri (polII, implicata nella rimozione degli inneschi) e include le 3 DNA-polimerasi replicative degli archei ma non le polimerasi batteriche di tipo I (polI) e di tipo III le cui sequenze configurano rispettivamente le famiglie A e C.
La radice dell'albero universale e la posizione filogenetica degli archei
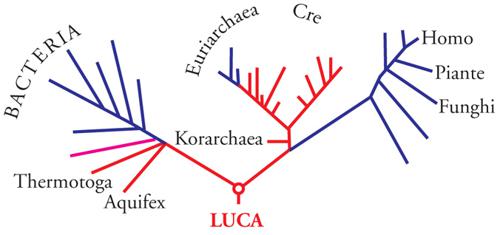
Le similarità esistenti tra gli apparati informazionali degli eucarioti e degli archei sono interpretabili alla luce di ricostruzioni filogenetiche che hanno permesso di stabilire l'ordine di radiazione dei tre domini. L'albero universale dedotto dalle sequenze degli Ssu-RNA e di diverse proteine ubiquitarie (fig. 2) è infatti un semplice 'cladogramma' (un albero senza radice) che illustra la suddivisione della biosfera in tre domini (o 'cladi'). Tuttavia, l'antichità relativa delle tre cladi rispetto a un antenato comune (la 'radice' dell'albero universale) è indeterminata.
La posizione della radice ‒ che può essere localizzata in tre posizioni alternative e, implicitamente, l'ordine di divergenza delle tre cladi ‒ è stata stabilita negli anni 1989-1998 utilizzando un espediente noto come 'radicazione paraloga'. In breve, se due geni paraloghi (geni con funzioni diverse originati dalla duplicazione di uno stesso gene ancestrale) sono presenti indistintamente in tutti i viventi, la duplicazione che li ha generati deve aver preceduto la prima radiazione evolutiva, a livello della cellula primigenia designata dall'acronimo LUCA (Last universal common ancestor). Pertanto, una filogenesi composita derivata dalle sequenze di due geni paraloghi universali (o delle proteine corrispondenti) deve generare due alberi (uno per ogni paralogo) uniti da un segmento che li 'radica' reciprocamente inserendosi, per così dire, nell'uno o nell'altro dei tre rami principali di ciascuno dei due alberi.
Delle diverse coppie di paraloghi universali individuate e usate finora per radicare l'albero universale, solo due hanno fornito risultati ineccepibili. La prima coppia è rappresentata dai fattori di elongazione EF-Tu e EF-G (denominati EF-1α ed EF-2 negli eucarioti e negli archei) implicati, rispettivamente, nel legame degli amminoacil-tRNA al ribosoma e nella traslocazione del peptidil-tRNA. La seconda coppia è formata dalle proteine monomeriche SRP54 e SRα (denominate Ffh e Ftsy nei batteri) universalmente implicate nella traslocazione cotraduzionale delle proteine secretive attraverso le membrane cisternali (negli eucarioti) o attraverso la membrana plasmatica (nei procarioti). I due membri di ogni coppia di proteine paraloghe condividono una regione di circa 200 amminoacidi ereditata dal gene parentale originario.
Le filogenesi costruite dalle sequenze condivise delle proteine EF-Tu(1α)-EF-G(2) e quelle ricostruite dalle sequenze condivise SRP54 (Ffh)-SR α Ftsy) sono concordanti: ambedue le coppie generano due alberi (uno per ciascun paralogo) reciprocamente radicati nel ramo dei batteri. Pertanto, la radice dell'albero universale (e implicitamente la posizione del LUCA) divide il ramo dei batteri del cladogramma rappresentato nella fig. 2 in due rami: uno destinato a generare il dominio batteri; un altro demandato a divergere ulteriormente nei domini Archaea ed Eucaria (fig. 4). Pertanto, archei ed eucarioti sono domini fratelli che hanno condiviso un percorso comune. È presumibile che durante questo percorso comune siano sorte le numerose invenzioni evolutive che accomunano i due domini.
Bibliografia
Amils 1993: Amils, Riccardo - Cammarano, Piero - Londei, Paola, Translation in archaea, in The biochemistry of archaea, edited by Morris Kates, Donn Kushner, A.T. Matheson, Amsterdam-New York, Elsevier, 1993, pp. 223-255.
Bell, Jackson 2001: Bell, Stephen D. - Jackson, Stephen P., Mechanism and regulation of transcription in archaea, "Current opinion in microbiology", 4, 2001, pp. 208-213.
Brown 2001: Brown, James R. e altri, Universal trees based on large combined protein sequence data sets, "Nature genetics", 28, 2001, pp. 281-285.
Delong, Pace 2001: Delong, Edward F. - Pace, Norman R., Environmental diversity of bacteria and archaea, "Systematic biology", 50, 2001, pp. 470-478.
Gribaldo, Cammarano 1998: Gribaldo, Simonetta - Cammarano, Piero, The root of universal tree of life inferred from anciently duplicated genes encoding components of the protein-targeting machinery, "Journal of molecular evolution", 47, 1998, pp. 508-516.
Huber 2003: Huber, Harald e altri, The phylum Nanoarchaeota: present knowledge and future perspectives of a unique form of life, "Research in microbiology", 154, 2003, pp. 165-171.
Iwabe 1989: Iwabe, Naoyuki e altri, Evolutionary relationship of archaebacteria, eubacteria, and eukaryotes inferred from phylogenetic trees of duplicated genes, "Proceedings of the National Academy of Sciences USA", 86, 1989, pp. 9335-9339.
Kandler 1994: Kandler, Otto, in: Molecular biology of archaea, edited by Felicitas Pfeifer, Peter Palm, Karl-Heinz Schleifer, Stuttgart, Fischer, 1994, pp. 1-9.
Kates 1993: Kates, Morris, Membrane lipids of archaea, in: The biochemistry of archaea, edited by Morris Kates, Donn Kushner and A.T. Matheson, Amsterdam-New York, Elsevier, 1993, pp. 261-292.
Kelman 2000: Kelman, Zvi, DNA replication in the third domain (of life), "Current protein and peptide science", 1, 2000, pp. 139-154.
Lange, Hausner 2004: Lange, Udo - Hausner, Winfried, Transcriptional fidelity and proofreading in archaea and implications for the mechanism of TFS-induced RNA cleavage, "Molecular microbiology", 52, 2004, pp. 1133-1143.
Langer 1995: Langer, David e altri, Transcription in archaea: similarity to that in eucarya, "Proceedings of the National Academy of Sciences USA", 92, 1995, pp. 5768-5772.
Laptenko 2003: Laptenko, Oleg e altri, Transcript cleavage factors GreA and GreB act as transient catalytic components of RNA polymerase, "The EMBO journal", 22, 2003, pp. 6322-6334.
Laursen 2005: Laursen, Brian S. e altri, Initiation of protein synthesis in bacteria, "Microbiology and molecular biology review", 69, 2005, pp. 101-123.
Londei 2006: Londei, Paola, Translation, in: Archaeal molecular cell biology, edited by Ricardo Cavicchioli, ASM Press, 2006 (in corso di stampa).
Snel 1999: Snel, Berend - Bork, Peer - Huynen, Martijn A., Genome phylogeny based on gene content, "Nature genetics", 21, 1999, pp. 108-110.
Stanier 1970: Stanier, Roger Y., Some aspects of the biology of cells and their possible evolutionary significance, "Symposium of the Society for General Microbiology", 20, 1970, pp. 1-38.
Wind, Reines 2000: Wind, Megan - Reines, Daniel, Transcription elongation factor SII, "Bioessays", 22, 2000, pp. 327-336.
Woese 1987: Woese, Carl R., Bacterial evolution, "Microbiological reviews", 51, 1987, pp. 221-271.
Woese 1990: Woese, Carl R. - Kandler, Otto - Wheelis, Mark L., Towards a natural system of organisms, "Proceedings of the National Academy of Sciences USA", 87, 1990, pp. 4576-4579.
Woese, Fox 1977: Woese, Carl R. - Fox, George E., Phylogenetic structure of the prokaryotic domain: the primary kingdoms, "Proceedings of the National Academy of Sciences USA", 74, 1977, pp. 5088-5090.
Woychik, Hampsey 2002: Woychik, Nancy A. - Hampsey, Michael, The RNA polymerase II machinery: structure illuminates sanction, "Cell", 108, 2002, pp. 453-463.
Zillig 1979: Zillig, Wolfram - Stetter, Karl O. - Janekovic, Davorin, DNA-dependent RNA polymerase from the archaeabacterium Sulfolobus acidocaldarius, "European journal of biochemistry", 96, 1979, pp. 597-604.
Zuckerkandl, Pauling 1965: Zuckerkandl, Emile - Pauling, Linus, Molecules as documents of evolutionary history, "Journal of theoretical biology", 8, 1965, pp. 357-366.
Siti internet
www.ncbi.nlm.nih.gov