Biosfera. Aspetti genomici dell'oceanografia microbica
Biosfera. Aspetti genomici dell'oceanografia microbica
Gli ecosistemi marini sono complessi e dinamici. La conoscenza dei meccanismi della suscettibilità degli ecosistemi marini alle variazioni ambientali globali e ai cambiamenti climatici dovuti ai gas serra necessita di una descrizione dettagliata di vari fattori, tra cui le interazioni marine fisiche, chimiche e biologiche, con soglie e meccanismi di feedback positivi e negativi e altre interazioni non lineari. I flussi di materia e di energia, e i microbi che li mediano, sono di importanza cardinale nell'oceano, eppure rimangono poco chiari. Negli ultimi trent'anni, dettagliati studi sul campo hanno corroborato l'ipotesi del microbial loop (catena microbica) nella quale i microbi hanno un ruolo centrale per la conversione a livelli trofici superiori del materiale organico disciolto. Tuttavia, tale teoria non è stata ancora messa alla prova in modo esaustivo e specifico. Inoltre, l'importante ruolo dell'attività microbica nel mantenimento dello stato ossidativo del nostro pianeta e nei cicli bio-geochimici diversi da quello del carbonio, non sono catturati al meglio dall'ipotesi del loop microbico. Permangono dunque ostacoli notevoli da superare nella misura e nella modellizzazione dei processi microbici oceanici.
Una delle sfide principali dei microbiologi oceanografi è quella di incorporare in modo più accurato i dettagli della diversità, della fisiologia e dell'ecologia nei modelli oceanografici e bio-geochimici. Per esempio, i processi delle comunità di batterioplancton sono spesso stati modellizzati come se fossero solamente eterotrofi, sottovalutando la diversità di fisiologie e processi metabolici che operano in situ. Di fatto, l'importanza dei picoplancton fotoautotrofi, oggi riconosciuti come componenti dominanti delle comunità planctoniche, era stata trascurata nei primi modelli oceanografici. Inoltre, si riteneva che il metabolismo del plancton microbico rassomigliasse a quello dei ceppi comuni da laboratorio, alcuni dei quali di dubbia rilevanza ecologica. Scoperte recenti, come i nuovi approcci alla coltura, le strategie di identificazione e di enumerazione indipendenti dalla coltura, e la genomica ambientale, stanno migliorando la situazione. Per esempio, ora siamo in grado di identificare i geni e i percorsi biochimici che differenziano le specie microbiche di diversi habitat. Molto presto sarà possibile definire una mappa spaziotemporale della distribuzione metabolica e funzionale dei geni per le specie microbiche indigene provenienti da diverse province oceaniche. Anche la recente creazione di osservatori microbici in siti determinati sarà di grande aiuto in direzione di questa meta. La correlazione tra le caratteristiche genomiche di organismi e habitat e altre variabili fisiche, chimiche e biotiche contribuirà ad affinare la nostra comprensione dei processi microbici e bio-geochimici dei sistemi oceanici.
Tutti questi passi in avanti ‒ una migliore coltura, gli approcci genomici ambientali e gli osservatori microbici in situ ‒ fanno sperare in uno sviluppo della nostra conoscenza del sistema oceanico vivente.
Un breve riassunto storico
La conoscenza microbica marina è notevolmente migliorata durante gli ultimi trent'anni. In questo lasso di tempo i ricercatori hanno capito quanto fosse cruciale il ruolo svolto dai microbi all'interno degli ecosistemi oceanici. Questo si deve in parte ai progressi tecnologici, come per esempio l'evolversi delle tecnologie di epifluorescenza microscopica e le misurazioni di biomassa basate sull'ATP, che hanno rivelato la presenza di quantità di batterioplancton nettamente superiori a quanto precedentemente stimato. Inoltre, l'uso di radiotraccianti per calcolare il tasso di crescita e il ricambio dei batteri planctonici ha portato a rivedere i modelli qualitativi e quantitativi della contribuzione microbica alle reti alimentari marine. Per mezzo del conteggio cellulare per autofluorescenza e della citometria di flusso, si è infine arrivati alla scoperta di molti picoplancton fotoautotrofi, comprese le specie dei generi Synechococcus e Prochlorococcus, che dominano le attività di fotosintesi nelle correnti di circolazione oceanica. Più o meno contemporaneamente sono state scoperte le bocche idrotermali ricche di macrofauna e microflora, e i primi isolati batterici la cui crescita è obbligata dall'elevata pressione idrostatica. Lo sviluppo di analisi filogenetiche indipendenti dalle colture che utilizzano il sequenziamento dell'RNA ribosomiale (rRNA) e l'ibridazione fluorescente in situ hanno preparato il terreno per gli odierni studi di genomica ambientale. Poco dopo la loro scoperta, le tecniche di analisi filogenetiche basate sull'rRNA che utilizzavano la (allora) nuova tecnica di reazione a catena della polimerasi (PCR) svelarono l'ampia distribuzione e l'abbondanza di diversi gruppi microbici marini fino ad allora sconosciuti, tra cui il Pelagibacter (anche noto come SAR11), molti nuovi gruppi di Archaea planctonici marini e nuovi picoplancton eucariotici.
Più recentemente, è stata rilevata la prevalenza di batterioplancton contenenti batterioclorofilla e rodopsina, dando luogo a nuove prospettive sulla natura delle strategie di uso della luce nelle acque oceaniche di superficie. Stranamente, solo molto di recente è stato riconosciuto che le particelle virali potrebbero superare di un ordine di grandezza il numero totale di cellule microbiche nel plancton marino, e che rappresentano vettori potenzialmente importanti per la mortalità del batterioplancton e per il trasferimento genetico orizzontale.
Si continuano a descrivere molte specie microbiche marine e ci attendono ulteriori scoperte e descrizioni di inaspettate proprietà fisiologiche e biochimiche. Abbiamo ancora molto da imparare sulla distribuzione, variabilità e influenza bio-geochimica dei microbi naturali del mare. Nuove tecnologie stanno ora fornendo alle scienze della vita e a quelle oceaniche importanti opportunità di ricerca, e in particolare i progressi ottenuti dalla scienza e dalla tecnologia genomiche stanno svelando le interdipendenze che collegano i processi di evoluzione, ecologici e bio-geochimici delle comunità microbiche marine naturali. Per esempio, la coevoluzione e lo scambio genetico tra genomi nucleari e cloroplasti testimoniano eventi che hanno alterato profondamente i modi e i meccanismi predominanti della fotosintesi ossigenica sulla Terra.
La nostra conoscenza sulla frequenza e sui meccanismi di acquisizione di geni e caratteristiche funzionali sta cambiando radicalmente grazie agli studi genomici che indicano la centralità del trasferimento genetico orizzontale. In modo analogo, il recente riconoscimento dell'enorme diversità di specie simpatriche nel plancton microbico sta correggendo i punti di vista odierni su forma, funzioni e speciazione nel mare. La maggiore disponibilità di campioni della diversità genomica microbica naturale rivelerà maggiori dettagli del processo evolutivo e delle dinamiche ecologiche microbiche, e la loro relazione con i processi ambientali. In quest'ottica, molti sforzi sono ora concentrati sul sequenziamento di interi genomi microbici marini oltre che sull'analisi genomica di comunità microbiche marine complete. I risultati di questi sforzi potrebbero migliorare le descrizioni delle strutture e delle funzioni del sistema vivente oceanico.
Genomica delle specie microbiche marine coltivate
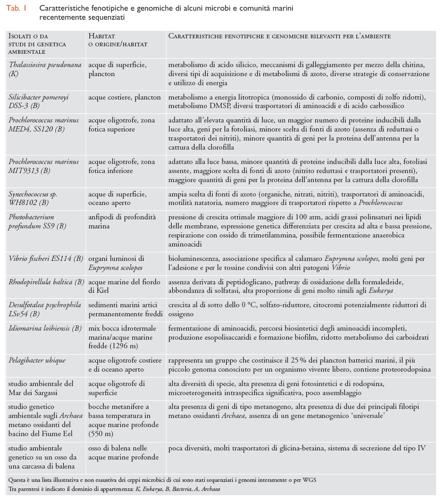
L'adattamento alle caratteristiche peculiari del loro ambiente è ciò che definisce l'essenza delle specie microbiche marine. Alto livello di salinità, basse e variabili concentrazioni di nutrienti ed elevati gradienti di temperatura, luce, pressione e nutrienti al variare della profondità, sono alcune delle variabili ambientali che influenzano l'evoluzione di forma e funzione dei plancton microbici. Le istruzioni adattative volte ad affrontare l'ambiente marino sono codificate nei modelli genomici dei microbiota oceanici. Il sequenziamento genomico è dunque uno strumento utile per esplorare la relazione tra genotipo, fenotipo e ambiente nelle specie microbiche marine naturali (tab.1). Alcuni esempi forniti di seguito illustrano in che modo la recente informazione ottenuta dai sequenziamenti abbia contribuito ad ampliare la nostra conoscenza sulle capacità di adattamento, la fisiologia e l'ecologia dei microbi marini.
Prospettive genomiche nelle foreste marine
Le diatomee, un tipo di alghe, sono tra le più cospicue forme vegetali marine. Il loro gruppo fornisce gran parte della produttività marina primaria contemporanea, specialmente nelle regioni vicino alla costa. Cosa possiamo imparare dal notevole successo evolutivo di questi protisti attraverso lo studio della struttura dei loro cromosomi del nucleo e dei cloroplasti? Che relazione esiste tra le caratteristiche genomiche delle diatomee e le loro proprietà ecologiche e fenotipiche? Può l'analisi genomica fornire nuove informazioni sulle proprietà metaboliche e fisiologiche di questo importante plancton? Le informazioni ottenute dal recente sequenziamento delle 34 Mbp (mega-base pairs, milioni di coppie di basi) del genoma di Thalassiosira pseudonana hanno risposto ad alcune di queste domande.
I 24 cromosomi diploidi di T. pseudonana contengono geni per le proteine associate alla costruzione della parete cellulare silicea, tra cui i geni per l'utilizzo dell'acido silicico, la sintesi di poliamina e la produzione di sillafin. Questi nuovi geni recentemente identificati, potranno ora essere sfruttati in esperimenti per studiare e modellizzare la sintesi della parete cellulare delle diatomee, oltre che per modellizzare i meccanismi reattivi di regolazione implicati nella limitazione di crescita causata dalla mancanza di silice. T. pseudonana e altre specie di diatomee estrudono le fibre di chitina dai pori delle pareti cellulari, presumibilmente per aumentare l'attrito e quindi diminuire la loro velocità di affondamento. Il genoma del T. pseudonana comprende molti geni per la sintesi e la degradazione della chitina, tra cui 22 geni putativi per la chitinasi, indicando un ciclo di sintesi/degradazione della chitina dinamico e forse altamente regolato per controllare il galleggiamento e i cicli dei nutrienti. La crescita delle diatomee è solitamente limitata dalla mancanza di nutrienti inorganici (azoto, fosforo e silice) e risponde al flusso variabile di nutrienti dell'ambiente che li circonda. Le caratteristiche del genoma di T. pseudonana riflettono il suo tipo di vita fotosintetico ma autotrofo, e comprendono una grande varietà di trasportatori di nutrienti, geni per l'acquisizione di azoto che permettono la trasformazione da diversi tipi di fonti di azoto sia organiche sia inorganiche, e molteplici vie per la conservazione dell'energia che utilizzano le vie di degradazione della sintesi di carboidrati e lipidi.
Un rapido sguardo genomico sui batteri costieri
Le trasformazioni fisiche, chimiche e biologiche che avvengono in una sezione marina che si estende dalla costa al mare aperto sono profonde. Le proprietà specifiche dei batterioplancton costieri li differenziano dai loro parenti in mare aperto? Possono le proprietà genomiche fisiologiche dei batterioplancton spiegare almeno in parte la loro distribuzione così come riscontrata? Queste diverse caratteristiche biologiche possono fornirci informazioni sulle potenziali differenze regionali nei cicli microbici di energia e materia? Sebbene sia troppo presto per proporre ampie generalizzazioni, gli studi più recenti hanno iniziato a fornire le risposte ad alcuni di tali quesiti. Sembra, per esempio, che alcuni gruppi batteriologici come l'α-proteobatterio del clade Roseobacter siano più numerosi nelle acque vicino alla costa, piuttosto che in mare aperto. Il genoma di Silicibacter pomeroyi, un membro del clade Roseobacter, mostra delle forme di adattamento che sembrano adeguarsi alla vita negli ambienti costieri. Secondo il suo contenuto genomico, pare che S. pomeroyi non consumi soltanto carbonio organico disciolto ma anche composti inorganici ridotti come il monossido di carbonio e lo zolfo: contiene infatti i geni necessari per generare energia da questi composti.
Esperimenti preliminari hanno confermato che il monossido di carbonio e lo zolfo ridotto sono substrati importanti del metabolismo di S. pomeroyi. Come il T. pseudonana, questo batterio costiero dispone di un'ampia gamma di trasportatori dei nutrienti per poterli ricavare dai diversi composti di azoto, come gli aminoacidi, l'ammonio e l'urea.
Il genoma di S. pomeroyi codifica inoltre diversi trasportatori per l'assorbimento di osmoliti, come il dimetilsulfoniopropionato (DMSP) e la glicina betaina probabilmente prodotti dai suoi coabitanti algali abbondanti negli ambienti costieri. Sia le analisi genomiche sia quelle fisiologiche suggeriscono che S. pomeroyi abbia una strategia metabolica lito eterotrofa che gli permetterebbe di produrre indifferentemente energia dai composti organici e inorganici. In genere, queste caratteristiche genomiche e fisiologiche di S. pomeroyi sembrano essere degli adattamenti specifici alle micronicchie tipiche degli ecosistemi costieri.
La varietà è il sale della vita marina
Per i microbiologi è comodo organizzare i gruppi microbici secondo le associazioni funzionali coinvolte in determinati processi, per esempio riduttori di solfati, metanogeni e fotoautotrofi ossigenici. È ormai chiaro che all'interno di questi gruppi, e persino all'interno della stessa specie, esista un'enorme diversità biologica. Le varianti fisiologiche e genetiche strettamente imparentate e apparentemente molto affini a determinate condizioni ecologiche sono state battezzate ecotipi. Cosa potrebbero rivelarci i modelli genomici sulle differenze eco-fisiologiche tra ecotipi? L'analisi del linkage dei geni in questi ecotipi potrebbe fornire informazioni sulle interdipendenze metaboliche? E scendendo nel dettaglio genomico, quante differenze devono accumularsi per far sì che un ecotipo sia ecologicamente distinto da un altro?
Uno dei migliori esempi documentati di ecotipo ambientalmente adattato è quello dei ceppi di Prochlorococcus isolati da diverse profondità. Le sequenze genomiche di questi ecotipi rivelano alcune delle strategie di adattamento uniche adoperate in diverse parti delle profondità marine. Sono stati sequenziati di recente diversi tipi di Prochlorococcus adattati ad alta e a bassa luce. Il ceppo MIT9313 adattato alla poca luce isolato dalla zona eufotica inferiore ha molti più geni sul suo genoma di 2,4 Mbp rispetto al suo parente di alta luce MED4, che ha un genoma di 1,66 Mbp. Tuttavia, la grandezza genomica non è necessariamente un'accurata indicazione di adattamento alla luce, visto che il genoma di un altro ceppo adattato alla poca luce, il Prochlorococcus SS120, è di 1,75 Mbp. I geni riscontrati nei ceppi da poca luce ma non nei loro parenti da alta luce includono quelli per il trasporto e l'assimilazione dei nitrati. Ciò coincide con il tipo di nutrienti disponibili in fondo alla zona eufotica. Il ceppo MED4 codifica molti più geni per proteine inducibili dalla luce alta del suo cugino da bassa luce. Il ceppo adattato alla poca luce MIT9313, invece, contiene più geni associati alla fotosintesi.
La sequenza genomica del Synechococcus WH8102 ha dimostrato caratteristiche diverse da quelle riscontrate nei Prochlorococcus. Il genoma di Synechococcus sembrerebbe indicare una maggiore versatilità rispetto al Prochlorococcus, con un'ampia gamma di capacità cataboliche e di acquisizione di nutrienti.
Database in espansione
Il sequenziamento completo di microbi marini provenienti da diversi habitat e ceppi filogenetici è una promessa per il futuro degli studi genomici comparativi. Altre sequenze marine di genomi microbici recentemente pubblicate includono quelle del solfato-riduttore psicrofilo Desulfotalea psychrophila, il bioluminescente simbiotico del calamaro Vibrio fischeri, il planctomicetomarino Rhodopirellula baltica, il batterio eterotrofo proveniente da una bocca idrotermale marina Idiomarina loihiensis, il batterio di profondità adattato all'alta pressione Photobacterium profundum e molti altri microbi marini che sono in corso di sequenziamento.
La continua analisi comparativa dei genomi microbici marini promette di arricchire sia i database sia la nostra conoscenza odierna. Tuttavia, la fenomenale microdiversità, i diversissimi percorsi evolutivi e le variabili strategie ecologiche delle specie microbiche marine impongono un'attenta campionatura da ulteriori habitat e l'acquisizione di ancora più sequenze genomiche, sia da specie diverse sia da quelle imparentate. Si stanno sviluppando con successo nuovi metodi per la coltura di specie microbiche comuni ma precedentemente resistenti alla coltura, il che dovrebbe aumentare l'importanza dei database genomici esistenti. Un ottimo esempio di ciò è stata la coltura di uno dei più comuni batteri oceanici, Pelagibacter ubique. La sequenza genomica di questo onnipresente microrganismo marino ha rivelato la sua natura lineare, e la presenza di una fotofisiologia basata sulla proteorodopsina. Gli sforzi in questa direzione continueranno ad aumentare la formazione di database comparativi per determinare il repertorio genomico delle specie microbiche coltivate, e a fornire il contesto cruciale necessario per interpretare le analisi ambientali genomiche in corso sulle comunità microbiche marine.
Osservazioni genomiche delle comunità microbiche naturali
I processi biologici che si verificano nelle comunità microbiche naturali presentano reazioni intra- e intercellulari diverse, complesse e interdipendenti. Una buona parte di questa complessità è codificata nella struttura, la distribuzione e la dinamica dei genomi che interagiscono con l'ambiente. Recentemente è diventato possibile accedere direttamente ai genomi di specie microbiche coesistenti nelle comunità naturali en masse, senza colture, attraverso gli approcci di genomica ambientale. A prescindere dal fatto che la si chiami genomica ambientale, metagenomica o ecogenomica, l'approccio genomico indipendente dalla coltura può fornire nuove prospettive sul mondo microbico naturale.
I primi studi di genomica ambientale si concentrarono sul picoplancton marino. Dapprima, grandi frammenti genomici venivano clonati direttamente dalle comunità microbiche marine per poter osservare le caratteristiche dei gruppi filogenetici ancora non coltivati, come gli Archaea marini. Studi genomici similari hanno rivelato recentemente l'esistenza della rodopsina nei batteri, nei quali non si immaginava potessero esistere queste fotoproteine. Le ipotesi a proposito di queste nuove rodopsine batteriche potrebbero essere testate sia in vitro sia in vivo attraverso espressioni eterologhe nell'Escherichia coli e susseguente caratterizzazione biofisica e biochimica. Successivi studi sul campo hanno rivelato la diversità e l'abbondanza delle rodopsine del batterioplancton. La progressione dalla sequenza genomica alla sperimentazione biochimica e biofisica, fino alle più raffinate osservazioni ecologiche rappresenta un modello promettente per lavori futuri con l'obiettivo di ottenere nuovi dati di sequenziamento genomico per la ricerca fisiologica, ecologica e oceanografica.
Un'altra applicazione recente della genomica marina utilizza il metodo di sequenziamento denominato WGS (Whole-genome shotgun), un sequenziamento rapido dell'intero genoma. Notevolmente utile per il sequenziamento di interi genomi microbici, il WGS comporta il sequenziamento casuale di piccoli (∼3 kbp) inserti di librerie e il successivo assemblaggio di lunghe sequenze consenso di DNA in silico. La velocità di questo metodo lo rende molto interessante, ma le variazioni ambientali in ricchezza, omogeneità ed eterogeneità genetica intraspecifica pongono diversi problemi per il riassemblaggio di lunghe sequenze di DNA partendo da sequenze di frammenti più piccoli. Tuttavia, l'approccio WGS può essere utile per ottenere grandi quantità di dati. L'esempio più eclatante è stato quello dell'osservazione genomica di comunità microbiche nel Mar dei Sargassi, dove sono stati recuperati più di 1,6 miliardi di coppie di basi e circa 1,2 milioni di geni da un assemblaggio microbico collettivo. Quest'esperienza ha dimostrato il pieno potenziale dello studio WGS per il sequenziamento di DNA: sono stati recuperati una sorprendente quantità di nuovi geni e famiglie di geni, e ulteriori risultati continueranno a essere raccolti. Tuttavia, ottenere sequenze di DNA lunghe e attendibili a partire da brevi frammenti ottenuti da popolazioni di batteri complesse ed eterogenee, rappresenta ancora un problema irrisolto.
Dagli studi in corso è evidente che un ruolo importante nell'interpretazione dei dati di sequenziamento WGS di comunità microbiche complesse sarà giocato dalla disponibilità di genomi di riferimento appropriati, e di ampie sequenze di cloni da utilizzare. Recentemente sono state confrontate, gene per gene, similitudini e differenze tra comunità microbiche provenienti dal Mar dei Sargassi, da un osso di balena, dal bacino di drenaggio acido di una miniera e da un silos di campagna, utilizzando i dati WGS. L'approccio di sequenziamento gene-centrico piuttosto che genoma-centrico ha reso possibile paragonare schemi di occorrenza di specifiche categorie di geni per mettere insieme dei profili comunitari per il contenuto funzionale dei geni. Le fin troppo presenti categorie specifiche delle EGT (etichette genetiche ambientali) in diversi campioni (per es., una presenza sproporzionata dei geni fotosintetici e di rodopsina nel campione del Mar dei Sargassi) confermano l'utilità di questo approccio nell'identificare caratteristiche metaboliche uniche di specifici assemblaggi microbici.
Esistono diversi modi per la futura applicazione dei dati genomici delle comunità microbiche all'oceanografia microbica e all'ecologia: (a) una lista completa di elementi microbici (per es., i geni, i taxon e i processi biochimici presenti) degli abitanti microbici oceanici sarà in grado di fornire una visione più dettagliata del loro potenziale fisiologico. Questi dati possono essere utili per formulare delle ipotesi biochimiche e fisiologiche da testare in laboratorio e sul campo; (b) dettagliati dati genomici basati sulle sequenze forniranno inoltre strumenti informatici e reagenti, sotto forma di sonde molecolari e target molecolari e organiche previste, per poter tracciare e quantificare meglio specifici microrganismi e processi microbiologici. Per esempio, è ora vicina la possibilità di mappare quantitativamente più gruppi microbici nel tempo e nello spazio utilizzando la PCR quantitativa, e in futuro anche le tecniche di DNA microarray potrebbero svolgere un ruolo importante. Miglioramenti nella mappatura dei geni e di espressione genica, e inventari ottenuti sul campo, come anche tecniche quantitative più specifiche, potranno portare a misurazioni bio-geochimiche più significative. A sua volta, questa mappatura di geni, taxon e metabolismi, potrebbe permettere lo sviluppo di modelli che riflettano in modo più accurato i dettagli biologici riscontrati in situ; (c) le analisi comparative di interi assemblaggi microbici potrebbero anche svelare degli schemi di interazione biochimica su scala più grande e delle correlazioni specifiche per habitat che potrebbero sfuggire negli studi sulle singole specie. Per esempio, lo studio genomico comparativo delle comunità potrebbe rivelare delle interazioni sino a ora sconosciute: l'utilizzo della luce mediata dalla rodopsina nei batteri, era completamente ignoto prima dell'applicazione della genomica ambientale.
Sarà infine possibile sovrapporre geni funzionali microbici, vie metaboliche e distribuzioni tassonomiche ad altri dati fisiologici, chimici e biologici marini per poter mappare le caratteristiche microbiche delle diverse province oceaniche. Lo studio della struttura e delle dinamiche genomiche in situ fornirà dunque l'informazione necessaria per interpretare i processi evolutivi ‒ come la deriva genetica, la ricombinazione e il trasferimento genetico laterale ‒ che controllano l'adattamento e le divergenze microbiche. A loro volta questi dati forniscono elementi di previsione per modellizzare la varietà di forme, processi e funzioni che compongono le interazioni fisiche, chimiche e biotiche del mare.
Sfide contemporanee della oceanografia microbica
Una delle critiche più comuni nei confronti della continua raccolta di dati è che l'accumulo di informazioni non corrisponde all'aumento di conoscenze. Sebbene sia corretto dire che ci sono molte sfide da superare, esse non sono però insormontabili. I primi compiti da svolgere sono quelli di organizzare, standardizzare e coordinare l'annotazione, l'accesso e l'integrazione dei dati di derivazione genomica. La comunità scientifica deve riconoscere che le serie di dati genomici sono sostanzialmente diverse da altri database esistenti in quanto non provengono da una singola specie, taxon o locus genetico. Piuttosto, ogni serie discreta di dati rappresenta i geni collettivi recuperati casualmente da un singolo assemblaggio microbico. Queste serie di dati deriva da popolazioni e ambienti microbici dinamici, nei quali la ricchezza, omogeneità e composizione delle specie (e dunque anche la ricchezza, omogeneità e composizione dei geni) sono variabili nel tempo e nello spazio. Questa è una delle ragioni principali per cui ogni tentativo di assemblaggio deve essere qualificato e specificato, perché le singole sequenze di un qualsiasi assemblaggio potrebbero provenire da diversi campioni, tempi e origini. Le letture di sequenza originali e i loro metadati devono essere esplicitamente collegati agli assemblaggi in quanto sono di origine potenzialmente eterogenea.
Diverse altre sfide informatiche dovranno essere prese in considerazione via via che il campo della genomica ambientale si evolverà. La gestione, l'annotazione e l'archiviazione delle serie di dati sono in corso e ora molti, se non tutti, i meccanismi necessari per gestire grandi serie di dati stanno emergendo o sono già disponibili. Una delle questioni da valutare è la determinazione di standard e requisiti per i metadati di genomica ambientale. Inoltre, devono essere sviluppati meglio gli strumenti e gli algoritmi per l'annotazione dei dati di genomica ambientale. I modelli nascosti di Markov, efficaci nell'identificazione di geni per intere sequenze genomiche, non sono applicabili nell'annotazione di sequenze più corte tipiche delle informazioni metagenomiche. Un'altra importante area di sviluppo sono i nuovi algoritmi per l'identificazione dei geni. In parallelo alle questioni riguardanti i database, c'è una forte necessità di sviluppare ulteriormente gli strumenti computazionali per analizzare e confrontare le serie di dati genomici sulle comunità microbiche. Infine, l'interoperabilità tra i database di sequenze di dati genomiche e metagenomiche e i database ambientali ed ecologici sarà senz'altro fondamentale in futuro.
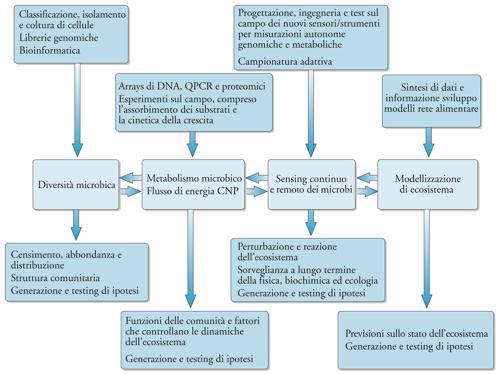
L'ecologia microbiologica e la genomica si trovano oggi a un bivio di opportunità. È importante capire i processi marini e collocare il sapere genomico sui microrganismi marini all'interno del contesto ecologico dei cicli di carbonio, azoto, fosforo e degli altri cicli bio-geochimici correlati. Sono in gioco benefici importantissimi sia per la scienza sia per la società. Il piano di ricerca deve partire dalla completa integrazione di microbiologia, biologia dei sistemi, dinamica degli ecosistemi e oceanografia, allo scopo di produrre dei modelli esaurienti, essenziali per poter determinare la scala delle interazioni e dei processi di determinati microrganismi in qualsiasi bacino marino. La fig. 5 mostra alcuni degli strumenti, approcci, processi e flussi di informazione necessari per ottenere questa nuova sintesi. Per avere un'idea della dimensione di questa sfida pensiamo al fatto che le previsioni climatiche ipotizzano un oceano globale più caldo e stratificato con più acque acide vicino alla superficie, e tutto ciò per lo più come conseguenza del crescente peso del CO2 per l'atmosfera. Come reagiranno i genotipi di oggi di fronte a queste nuove variabili? Siamo pronti per l'osservazione e l'interpretazione delle risposte fenotipiche espresse dall'oceano? Saranno necessari degli approcci nuovi e integrativi che permettano di collegare i processi microbici e quelli globali.
Una strategia per la sorveglianza dell'ecosistema che aumenterebbe la nostra conoscenza di questi complessi processi sarebbe la creazione di un network globale di stazioni oceaniche per l'analisi temporale, che permetterebbero di ottenere ripetute osservazioni della diversità microbica. Malgrado la loro importanza sia più che riconosciuta nel campo delle scienze della terra e del mare, gli osservatori microbiologici di lungo termine posti in habitat oceanici chiave sono rari. Negli ultimi quarant'anni le osservazioni ripetute della corrente subtropicale del Nord Pacifico hanno registrato mutamenti fondamentali nella struttura delle comunità microbiche e nei cicli dei bioelementi più importanti. Si ipotizza che la variabilità del clima nel bacino del Nord Pacifico abbia portato un'intensificazione della fissazione di azoto a base microbica oltre a uno spostamento fondamentale della limitazione di azoto a quella di fosforo, con diverse conseguenze ecologiche, comprese le alterazioni alle strutture delle reti alimentari e i cambiamenti alla sequestrazione del carbonio. Questi cambiamenti si sono verificati per via di sottili modifiche nella stratificazione oceanica vicino alla superficie e alla diminuzione dei flussi di ambienti inorganici dal basso. Questi mutamenti nell'habitat sono controllati, in parte, dalla frequenza e dalla durata dei sistemi climatici el Niño e PDO (Pacific decadal oscillation). Il potenziale di fissaggio di azoto dell'oceano globale potrebbe, in ultima analisi, essere controllato dal deposito atmosferico di ferro, un processo che dipende a sua volta dal tempo e dal clima. Il trasporto di polveri atmosferiche totale è influenzato dall'umanità, inclusa la demografia, l'economia globale, e gli schemi di utilizzo della terra. Queste complesse forze naturali e antropogeniche, ognuna delle quali possiede molteplici cicli di potenziale feedback e possibili effetti soglia, hanno un impatto globale significativo sui microbi dell'oceano, che solo ora sta cominciando a essere apprezzato. L'influenza delle variabilità climatiche, specialmente quelle che si verificano nel corso di decenni o secoli, sulla bio-geochimica microbica marina non è ancora ben compresa.
Conclusioni
La presenza dei microbi marini è di qualche miliardo di cellule per litro di acqua marina. Raddoppiano ogni pochi giorni e vengono consumati più o meno allo stesso ritmo dai parassiti vitali e dai protisti predatori. Queste attività catturano e processano energia e agiscono da motore per i cicli elementari principali. Nascoste tra questi assemblaggi dinamici e strutture genomiche molteplici, si trovano lezioni fondamentali, anche se tuttora incomplete sulla percezione dell'ambiente, le reazioni e l'adattamento, la regolazione genetica, l'interazione tra specie e comunità e la plasticità e l'evoluzione genetica. La diversità genomica, le dinamiche evolutive e i processi ecologici di queste popolazioni hanno effetti globali sui flussi di energia e materia nel mare, sui cicli bio-geochimici, sulla composizione atmosferica e sui trend climatici della Terra. Nei 3,8 miliardi di anni di vita sulla Terra, i microbi sono stati i 'maggiordomi' dell'equilibrio geochimico, e come registratori biotici della storia evolutiva la loro memoria biologica va più indietro nel tempo di quella di altre forme di vita. Inoltre, sono i biosensori della Natura par excellence, ma dobbiamo ancora decifrare il loro codice per poter interpretare il loro impatto e comprendere le strategie sottostanti e i meccanismi utilizzati per la loro sopravvivenza in Natura. La convergenza di microbiologia, ecologia, genomica e oceanografia offre la possibilità di concentrarsi, come mai prima, sull' oceanografia microbiologica. Cooperando i biologi molecolari, i microbiologi e gli oceanografi potranno trovare nuovi modi per migliorare le osservazioni, i metodi e la teoria, e per descrivere meglio il sistema vivente oceanico.
Bibliografia
Armbrust 2004: Armbrust, E. Virginia e altri, The genome of the diatom Thalassiosira pseudonana: ecology, evolution, and metabolism, "Science", 306, 2004, pp. 79-86.
Béjà 2001: Béjà, Oded e altri, Proteorhodopsin phototrophy in the ocean, "Nature", 411, 2001, pp. 786-789.
DeLong 2005: DeLong, Edward F., Microbial communitygenomics in the ocean, "Nature reviews: microbiology", 3, 2005, pp. 459-469.
Dufresne 2003: Dufresne, Alexis e altri, Genome sequence of the cyanobacterium Prochlorococcus marinus SS12O, a nearly minimal oxyphototrophic genome, "Proceedings of the National Academy of Sciences USA", 100, 2003, pp. 10020-10025.
Giovanonni 2005: Giovanonni, Stephen J. e altri, Genome streamlining in a cosmopolitan oceanic bacterium, "Science", 309, 2005, pp. 1242-1245.
Glöckner 2003: Glöckner, Frank O. e altri, Complete genome sequence of the marine planctomycete Pirellula sp. strain 1, "Proceedings of the National Academy of Sciences USA", 100, 2003, pp. 8298-8303.
Hallam 2004: Hallam, Steven J. e altri, Reverse methanogenesis: testing the hypothesis with environmental genomics, "Science", 305, 2004, pp. 1457-1462.
Kolber 2000: Kolber, Zbigniew S. e altri, Bacterial photosynthesis in surface waters of the open ocean, "Nature", 407, 2000, pp. 177-179.
Lerat 2005: Lerat, Emmanuelle e altri, Evolutionary origins of genomic repertoires in bacteria, "PloS biology", 3, 2005,p. e130.
Palenik 2003: Palenik, Brian e altri, The genome of a motile marine Synechococcus, "Nature", 424, 2003, pp. 1037-1042.
Rabus 2004: Rabus, Ralf e altri, The genome of Desulfotalea psychrophila, a sulfate-reducing bacterium from permanently cold Arctic sediments, "Environmental microbiology", 6, 2004, pp. 887-902.
Rappé 2002: Rappé, Michael S. e altri, Cultivation of the ubiquitous SAR11 marine bacterioplankton clade, "Nature", 418, 2002, pp. 630-633.
Ruby 2005: Ruby, Edward G. e altri, Complete genome sequence of Vibrio fischeri: a symbiotic bacterium with pathogenic congeners, "Proceedings of the National Academy of Sciences USA", 102, 2005, pp. 3004-3009.
Stahl, Tiedje 2002: Stahl, David A. - Tiedje, James M., Microbial ecology and genomics: a crossroads of opportunity,Washington D.C., American Society for Microbiology, 2002.
Thompson 2005: Thompson, Janelle R. e altri, Genotypic diversity within a natural coastal bacterioplankton population, "Science", 307, 2005, pp. 1311-1313.
Tringe 2005: Tringe, Susannah G. e altri, Comparative metagenomics of microbial communities, "Science", 308, 2005, pp. 554-557.
Vezzi 2005: Vezzi, Alessandro, Life at depth: Photobacterium profundum genome sequence and expression analysis, "Science", 307, 2005, pp. 1459-1461.