Biosfera. Ecologia del plancton microbico
Biosfera. Ecologia del plancton microbico
A causa di alcune delle sue caratteristiche ‒ in particolare il basso contenuto di nutrienti prevalente sulla superficie dell'oceano ‒ l'ambiente oceanico viene spesso percepito come un ecosistema estremo. Spesso forme fissate di azoto, fosforo e ferro si trovano a livelli molto bassi o impercettibili nelle correnti circolari dell'oceano, e cioè in circa il 70% degli oceani. La fotosintesi è la fonte principale dell'energia metabolica e la base della catena alimentare; il fitoplancton oceanico rappresenta circa il 50% della fissazione di carbonio totale, e metà del carbonio fissato in materia organica viene respirato rapidamente da microrganismi eterotrofi. La maggior parte delle cellule sono liberamente sospese in una colonna d'acqua per lo più ossica, ma alcune si attaccano ad aggregati. In genere, queste cellule sopravvivono attraverso la fotosintesi o l'ossidazione di materia organica disciolta (MOD) o di composti inorganici, utilizzando l'ossigeno come elettron-accettore.
La concentrazione di cellule microbiche è tipicamente di circa 105 cellule/ml sulla superficie oceanica (0÷300 m), e l'assorbimento di timidina nel DNA microbico indica un tasso di crescita medio di circa 0,15 divisioni al giorno. Un efficiente riciclaggio di nutrienti, con un'intensa competizione per le scarse risorse, sostiene questa crescita, mentre la predazione di virus e protozoi contiene la popolazione e causa un alto tasso di turnover. Nonostante questa competizione, la concentrazione di carbonio organico disciolto (DOC) allo stato stabile è spesso di molte volte superiore al carbonio riscontrato in biomasse microbiche viventi. Tuttavia l'età media del pool di DOC nell'oceano profondo, pari a circa 5000 anni (determinato con datazione isotopica), suggerisce che una gran parte della MOD è resistente alla degradazione. Anche se la MOD costituisce un'enorme risorsa che può rivaleggiare con il CO2 atmosferico come pool di carbonio, a causa della sua complessità i chimici sono riusciti a caratterizzarla solo in termini generici.
Quanto si è detto fino a ora descrive le caratteristiche salienti dell'ambiente oceanico, ma tralascia i complessi schemi di variazioni fisiche, chimiche e biologiche che spingono l'evoluzione e la diversificazione dei microrganismi. Per esempio, i componenti del genere Vibrio ‒ che comprende alcuni tra i più frequenti batteri planctonici che possono essere isolati su una piastra di agar ‒ crescono facilmente in modo anaerobico attraverso la fermentazione. Il ciclo di vita di alcune delle specie Vibrio include una fase anossica associata a un ospite animale, ma il quadro generale della loro ecologia negli oceani non è stato ancora caratterizzato. Lo stesso si dica per la maggior parte dei gruppi microbici che verranno citati più avanti: la mappa filogenetica è dettagliata, ma il panorama ecologico è appena abbozzato. Gli studi biologici e metagenomici in corso stanno tuttavia producendo nuove conoscenze riguardo la coltura di organismi chiave. È molto probabile che nei prossimi anni la biologia dei gruppi di plancton microbico riveli i suoi segreti.
Esamineremo tutti gli aspetti, attualmente noti, della diversità marina dei batteri e degli archei, a partire dalla filogenesi dei geni recuperati dalla colonna d'acqua oceanica, e verranno considerate le implicazioni della diversità microbica per la comprensione dell'ecologia degli oceani. Molti degli argomenti che verranno presentati si riferiscono anche ai protisti, e ci si concentrerà soprattutto sullo strato oceanico di superficie (0÷300 m), facendo riferimento occasionalmente anche alle profondità abissali.
La diversità filogenetica nell'oceano
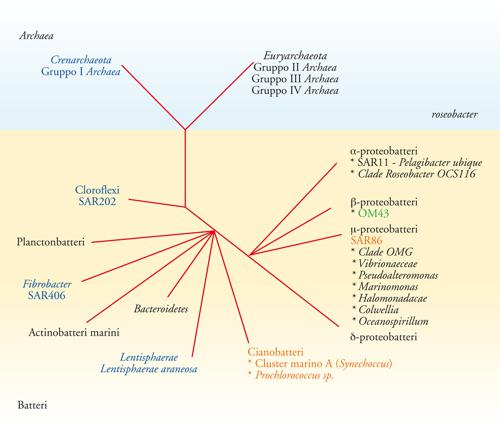
I geni dell'RNA ribosomico (rRNA) sono utilizzati come marcatori filogenetici universali e costituiscono il principale criterio di identificazione e nomenclatura dei gruppi di plancton microbico. Gran parte dei gruppi microbici marini sono stati identificati attraverso l'analisi delle sequenze di geni rRNA clonati da campioni di acqua di mare, e non sono ancora stati cresciuti in coltura. Dopo i primi studi, è divenuto chiaro che la maggior parte dei geni recuperati appartenevano a un gruppo di circa 20 cladi microbici. La fig. 2 è una rappresentazione schematica della filogenesi dei principali cladi di plancton.
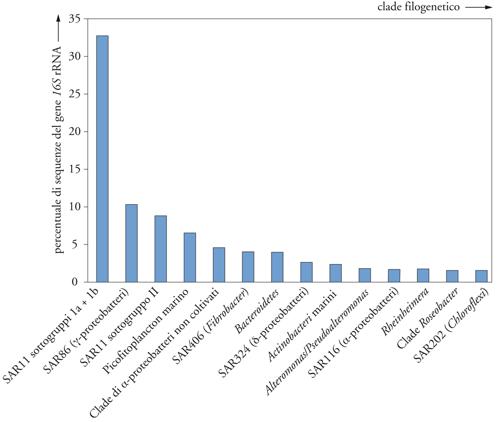
La recente attività di sequenziamento rapido su larga scala del DNA dell'acqua di mare sta fornendo con un'alta risoluzione la filogenesi dei frammenti dei geni 16S dell'rRNA (un gene universale nei procarioti, la cui variabilità lo rende molto utile nella classificazione), permettendo di mappare con precisione le distribuzioni biogeografiche del plancton microbico marino. La prima raccolta di campioni, effettuata nel 2003 dalla spedizione Sorcerer II di J. Craig Venter nel Mar dei Sargassi, ha fruttato 1184 frammenti di geni 16S rRNA (fig. 3). Essi rappresentano una ricca risorsa scientifica per due motivi: (a) non sono contaminati da artefatti dovuti all'amplificazione per mezzo della PCR (Polymerase chain reaction) che raramente interferiscono con il corretto posizionamento dei geni nelle categorie filogenetiche, ma rappresentano un problema significativo nella ricostruzione di schemi evolutivi a livello popolazionale; (b) l'enorme numero di geni raccolto dalla spedizione Sorcerer II sta rivelando la distribuzione e l'abbondanza di gruppi microbici che compongono soltanto una minima parte della comunità. Come vedremo in seguito, potrebbero rientrare in questa categoria alcuni ceppi opportunistici che sfruttano condizioni transitorie.
L'interpretazione dei dati relativi alla sequenza ambientale del Mar dei Sargassi è già al centro di interessanti dibattiti. L'insieme dei dati della sequenza dei geni 16S rRNA del Mar dei Sargassi viene riportato nella fig. 3, senza le sequenze di Burkholderia e Shewanella, rare in quell'ecosistema e sospettate di essere dei possibili contaminanti. Classificare il plancton microbico per cladi, come è stato fatto nella fig. 2, è una convenzione adottata dalla maggior parte degli oceanografi e si basa su principî evolutivi. Con poche eccezioni, i dati del Mar dei Sargassi rientrano nei cladi di plancton microbico già precedentemente identificati. Tuttavia, Venter ha sottolineato la nuova diversità che risulta dai dati, concludendo che nei campioni recuperati dal Mar dei Sargassi erano presenti 1900 specie genomiche di batteri e 145 nuovi filotipi. Egli è arrivato a questa conclusione applicando una regola approssimativa che presuppone che le sequenze di geni 16S rRNA che sono simili per meno del 97% provengano da specie diverse. Come discusso più avanti, le origini della diversità di sequenza dei geni 16S rRNA all'interno dei cladi microbici identificati è un tema scottante.
Anche se la maggior parte dei principali cladi microbici hanno una distribuzione cosmopolita, continuano a emergere nuovi cladi di plancton microbico marino da studi che si concentrano su caratteristiche idrografiche uniche. Per esempio, i due nuovi gruppi di Archaea Crenarchaeota ed Euryarchaeota, sono stati scoperti al limite tra acqua marina e acqua ipersalina dello Shaban Deep, nel Mar Rosso.
Schemi nel tempo e nello spazio
La biologia molecolare ha colmato dei vuoti sulla storia naturale del plancton microbico marino. Man mano che sono aumentati i marcatori genetici disponibili per studi ecologici, si è scoperto che alcuni dei cladi dominanti di plancton microbico sono stratificati verticalmente (fig. 2). Le prime indicazioni di questi schemi sono emerse dalle distribuzioni dei cloni dei geni rRNA all'interno delle librerie raccolte a diverse profondità. Anche se lo studio della stratificazione della comunità microbica non è ancora completo, in molti casi (cianobatteri marini unicellulari, SAR11, SAR202, SAR406, SAR324, Archaea marini del gruppo I) la stratificazione verticale delle popolazioni è stata confermata da approcci sperimentali alternativi. L'interpretazione più logica è che molti di questi gruppi si specializzino nello sfruttamento di schemi verticali in fattori fisici, chimici e biologici. Un chiaro esempio è dato dai cianobatteri marini unicellulari. In qualità di batteri fototrofi obbligati, questi cianobatteri rimangono confinati alla zona fotica. Uno schema similare si trova nel clade SAR86 del γ-proteobatterio. Sono stati trovati frammenti dei geni per la proteorodopsina (una proteina che reagisce alla luce) nei genomi del SAR86, il che fa pensare che questo clade abbia il potenziale per la metabolizzazione fototrofica. Anche molti degli enigmatici gruppi microbici per cui non è stata identificata alcuna particolare strategia metabolica sono stratificati. Il confine tra la zona fotica e il buio della zona mesopelagica superiore è particolarmente sorprendente ‒ al di sotto della zona fotica l'abbondanza del picofitoplancton e del SAR86 cala drasticamente, e gli Archaea marini del gruppo I, il SAR202, il SAR406 e il SAR324 acquistano un ruolo preponderante. Le implicazioni di queste osservazioni sono chiare: la comunità del mesopelagico superiore è quasi certamente specializzata nella raccolta di risorse provenienti dalla zona fotica. Tuttavia, con l'eccezione degli Archaea marini del gruppo I, le informazioni disponibili sull'attività individuale dei gruppi del mesopelagico superiore sono scarse.
Esistono anche delle differenze significative tra le popolazioni di plancton microbico costiere e di mare aperto (fig. 2). Tipicamente, le piattaforme continentali sono molto più produttive delle correnti circolari oceaniche perché i processi fisici come la risalita e il rimescolamento delle acque portano i nutrienti in superficie. Ne consegue che il fitoplancton eucariotico costituisce la parte maggiore della biomassa nelle zone costiere, e le specie si differenziano in popolazioni costiere e oceaniche. La maggior parte dei gruppi batterici trovati nelle correnti circolari si presentano numerosi anche sulle coste, ma un certo numero di cladi di plancton microbico, e in particolare i β-proteobatteri, hanno un ecotipo costiero o sembrano prevalentemente limitati alle zone costiere.
Uno dei gruppi microbici più enigmatici nell'oceano è quello degli Archaea marini del gruppo I. Interessanti testimonianze geochimiche sembrano indicare che questi sono organismi chemioautotrofi. Negli anni Novanta del Novecento è stato chiarito che gli Archaea sono ampiamente distribuiti e numericamente significativi nella colonna d'acqua marina. Gli Archaea marini del gruppo I sono i Crenarchaeota, che vivono principalmente nel mesopelagico, ma d'inverno si trovano anche in superficie nelle acque fredde dell'oceano meridionale. Con la tecnica di ibridazione a fluorescenza in situ si è dimostrato che le popolazioni di Archaea marini del gruppo I costituiscono circa il 40% delle comunità microbiche mesopelagiche in tutta l'area oceanica, e sono quindi tra gli organismi più abbondanti del pianeta. Nessuno degli Archaea marini è però ancora stato cresciuto in coltura.
Nuovi dati sulle distribuzioni microbiche hanno fornito importanti indizi sull'attività geochimica, ma gli sviluppi di queste ricerche sono venuti soprattutto dalle colture o da approcci sperimentali a partire dalle popolazioni native. Sono stati usati sia la tecnica di citofluorimetria a flusso sia l'ibridazione a fluorescenza in situ per separare le popolazioni e misurarne l'assorbimento di substrati radioattivi. Nel testare ipotesi scaturite dall'analisi delle sequenze genomiche, gli oceanografi sono stati sorpresi dal fatto che i batteri marini unicellulari, e in particolare il Prochlorococcus, possono assimilare amminoacidi liberi, mentre fino a quel momento si era creduto che potessero contare esclusivamente sull'azoto inorganico.
La questione delle specie
La questione di come denominare le specie di plancton microbico non è banale. Per gli oceanografi il problema è dove tracciare il confine in modo che organismi con diverse proprietà geochimiche possano avere nomi diversi. Da una prospettiva evolutiva, la domanda potrebbe porsi diversamente: in che modo si possono suddividere popolazioni di cellule che utilizzano le stesse risorse e possiedono gli stessi meccanismi adattativi ereditati da un comune antenato? La confusione nasce dal fatto che non vi è accordo sulla definizione di specie microbica. La regola del 97% è semplice da applicare ma non tiene conto della complessa struttura dei cladi microbici. Per esempio, i cianobatteri marini unicellulari formano un clade che potrebbe costituire un'unica specie secondo la regola del 97%; tuttavia, c'è un generale accordo sul fatto che questo clade contiene molte specie dai fenotipi diversi. Cladi come il SAR86 e il SAR11 presentano maggiore diversità, ma possono chiaramente essere divisi in sub-cladi. Una delle teorie avanzate è che alcuni di questi sub-cladi 'folti' siano degli ecosistemi ‒ popolazioni con caratteristiche simili e nicchie uniche.
Lo studio della struttura dei cladi ha comunque mostrato che la maggior parte della variabilità delle sequenze si raggruppa verso le estremità dei rami filogenetici, e potrebbe essere attribuita a mutazioni neutrali accumulatesi nelle popolazioni. Inoltre, esaminando la divergenza sequenziale e la variabilità del genoma in un gruppo di 232 isolati di Vibrio splendidus prelevati dalla stessa zona costiera in tempi diversi, è stato notato che gli isolati differivano meno dell'1% in termini di sequenze del gene rRNA, ma mostravano grande variazione nelle dimensioni del genoma e nella diversità allelica. Tali risultati potrebbero spiegare perché il gruppo di Venter ha avuto difficoltà ad assemblare i genomi microbici marini dai dati ottenuti attraverso il metodo di sequenziamento rapido utilizzato.
Tuttavia i modelli selettivi elaborati da Motoo Kimura nel 1962 prevedono che la selezione ha maggiori possibilità di agire su piccole modifiche di fitness man mano che la popolazione cresce, e che pertanto popolazioni grandi e stabili dovrebbero essere maggiormente ottimizzate dalla selezione. Nello specifico, Kimura ha coniato l'espressione 'dimensione effettiva della popolazione' per indicare la dimensione minima raggiunta da una popolazione che va incontro a fluttuazioni. Per questi microbi marini, è possibile che laddove popolazioni grandi non abbiano avuto recenti episodi di selezione purificatrice, siano in grado di mantenere grandi riserve di variabilità genetica neutrale. Se questa ipotesi si rivelasse giusta, allora, almeno all'interno degli ecotipi del plancton microbico, ci si aspetterebbe di trovare un insieme base di geni che conferisce un fenotipo relativamente conservato.
Il clade SAR11 ha dato la prima dimostrazione che i subcladi di cluster di geni ambientali potrebbero essere ecotipi. Un'indagine del rRNA ha rivelato la presenza di un clade di superficie (IA) e uno in profondità (II), ma non ha identificato la nicchia di un terzo subclade (IB). Più di recente, la nicchia del gene SAR11 subclade IB è apparsa in uno studio della transizione dalle condizioni di primavera-fioritura a quelle di estate-stratificazione nel Mar dei Sargassi occidentale. La tecnica di analisi multivariata non metrica ha rivelato che in primavera il subclade IB si trova ovunque nella colonna d'acqua, lasciando apparentemente posto ai subcladi più specializzati IA e II quando la colonna d'acqua diventa termicamente stratificata.
Il concetto di ecotipo acquisisce ulteriore importanza grazie al riconoscimento del fatto che molti gruppi microbici possono essere suddivisi a seconda della loro distribuzione nella colonna d'acqua. I cianobatteri unicellulari sono il miglior esempio. Si possono differenziare facilmente due ecotipi di Prochlorococcus per il loro rapporto clorofilla b/clorofilla a ‒ un ceppo altamente adattato alla luce (rapporto b/a alto) e un ceppo poco adattato alla luce (rapporto b/a basso). L'evidenza filogenetica derivata dallo studio sugli spaziatori interni trascritti (ITS) suggerisce che i filoni ad alto rapporto b/a possono essere differenziati in quattro ceppi geneticamente diversi. Le filogenesi basate sulla tecnica ITS indicano che il cluster marino A. Synechococcus può essere suddiviso in sei cladi, tre dei quali possono essere associati a caratteristiche fenotipiche importanti dal punto di vista adattativo (motilità, adattamento cromatico, e assenza di ficourobilina). Finora, le sequenze di genoma disponibili hanno appoggiato l'ipotesi che questi ecotipi si differenzino per caratteristiche che coinvolgono la loro capacità di competere. In particolare, il ceppo SS120, a basso rapporto b/a, ha un genoma molto più piccolo degli altri e può utilizzare solamente l'ammonio o gli aminoacidi come fonti di azoto. All'estremo opposto, il Synechococcus WH8102 può utilizzare ammonio, urea, nitrite, nitrato, cianato, peptidi e aminoacidi come fonti di azoto. È interessante osservare che le popolazioni del cluster marino A. Synechococcus sembrano prosperare in periodi di risalita e di rimescolamento verticale ‒ in cui sono presenti i nutrienti ma anche condizioni transitorie di caos. Pertanto, come osservato nel clade SAR11, i cianobatteri unicellulari marini sembrano avere degli specialisti per stagioni e degli specialisti per stratificazione.
Il fatto che i principali cladi di plancton microbico si siano separati in ecotipi rappresenta una forte evidenza in favore del fatto che la selezione crea delle entità funzionalmente e geneticamente uniche, nonostante l'influenza della variazione neutrale possa causare una divergenza relativamente marcata nelle sequenze di genomi. Tuttavia, anche se i cianobatteri marini unicellulari sono un ottimo modello per quanto ci riserva il futuro, il dibattito sulla diversità non è certo esaurito.
Vecchi paradigmi sfidati da nuove forme di fototrofia
L'inizio del nuovo millennio è stato caratterizzato dalle scoperte di nuove forme di fototrofia sulla superficie oceanica, che a loro volta hanno cambiato radicalmente la prospettiva sulle food web microbiche. Omologhi del gene per la batteriorodopsina collegati ai geni SAR86 rRNA sono presenti in cloni di cromosomi artificiali batterici provenienti da Monterey Bay. Inoltre, per mezzo della spettroscopia di fluorescenza a infrarossi ad alta frequenza di ripetizione, è stato possibile identificare le labili fluorescenze emanate dai batterioclorofilla a in acqua di mare. Dal punto di vista biochimico nessuno di questi due sistemi fotosintetici rappresenta una novità: entrambi sono già noti in oscuri gruppi microbici. I due sistemi analoghi utilizzano l'energia della luce per estrarre i fotoni dalle cellule, e si ritiene funzionino caricando il potenziale di transmembrana e supportando reazioni in salita, come il trasporto attivo e la fosforilazione dell'adenin dinucleotide fosfato (ADP). La novità sta nell'idea che il batterioplancton potesse utilizzare queste strategie su vasta scala per canalizzare l'energia in cellule che all'apparenza sono solo eterotrofi per la respirazione. Tuttavia, non si conosce ancora nel dettaglio quali specie siano dotate di questi sistemi, quando si esprimano e quali funzioni essi supportino.
La grande abbondanza e la diversità degli omologhi della batteriorodopsina sulla superficie del Mar dei Sargassi suggeriscono l'importanza di questi geni. Nella missione di Venter, sono state recuperate 743 sequenze del tipo della proteorodopsina e 1,164 sequenze rRNA: un rapporto di 0,7. Tuttavia, finora solo uno studio offre evidenza diretta che le proteorodopsine siano attive in acqua di mare; un altro studio mostra che l'incubazione di acqua di mare al buio non ha alcun effetto sulle popolazioni microbiche, anche se inibisce la crescita dei cianobatteri marini unicellulari. Una questione chiave è se la famiglia del gene della proteorodopsina svolga anche altre funzioni ‒ gli omologhi della proteorodopsina possono anche funzionare come ricettori e pompare altri ioni oltre ai protoni.
Strategie adattative
I dati disponibili sembrano suggerire che i batteri possono dividersi in due strategie ecologiche generali: quelle che sfruttano vuoti nell'ambiente e quelle che competono efficacemente a livello di nutrienti ambientali. La maggior parte dei ceppi studiati dai pionieri della microbiologia marina erano γ-proteobatteri eterotrofi che crescevano molto velocemente sul nutriente marino agar. Questi organismi si osservano raramente nelle ricerche sulla diversità del gene 16S rRNA, ma frequentemente dominano gli esperimenti in cui si manipolano comunità naturali di plancton microbico.
Il problema di dover ottenere colture biologicamente rilevanti ha portato i microbiologi marini a concentrare i loro sforzi su nuovi isolati eterotrofi che si replicano efficacemente a bassissimi livelli di concentrazione di nutrienti. Questi organismi, chiamati oligotrofici, spesso non rispondono bene a livelli di concentrazione di nutrienti maggiori. Una teoria sostiene che gli organismi oligotrofici non abbiano le complesse risposte regolatrici e le vie metaboliche che permettono alle loro controparti più robuste di crescere velocemente in ambienti ad alta concentrazione di nutrienti. Il primo isolato di SAR11, Pelagibacter ubique cresce lentamente in acqua di mare, raddoppiando la sua popolazione ogni 29 ore, e il tasso di crescita non aumenta in risposta a una maggior quantità di nutrienti di carbonio organico. Il suo genoma è piccolo, circa 1,3 milioni di paia di basi. Si è anche suggerito che i batteri ad alta e bassa crescita si differenzino fondamentalmente per il tasso di traduzione per ribosoma.
La modellizzazione metabolica ancora non è arrivata in microbiologia, ma si affaccia all'orizzonte. I modelli di flusso per processi geochimici a livello di ecosistema sono da sempre temi centrali nell'oceanografia biologica. Sembra essere solo una questione di tempo prima che lo sforzo postgenomico di creare modelli in silico per cellule si fonda con i modelli di flusso degli ecosistemi. È comunque stato sviluppato un modello matematico per cellule oligotrofiche che ne predice le proprietà da principî elementari. Il concetto centrale in questa teoria è che l'evoluzione ha agito sulle cellule oligotrofiche ai fini di massimizzare un termine chiamato la costante di affinità specifica. La costante di affinità specifica tiene conto dell'abilità generica delle cellule di trasformare i nutrienti in biomassa considerando il numero di trasportatori e la superficie della cellula, oltre alle affinità del substrato del trasportatore. Questa teoria prevede che la selezione agisca per ridurre la dimensione degli organismi oligotrofici affinché la capacità del citoplasma di processare il substrato non ecceda la capacità dei sistemi di trasporto di fornire il substrato.
I concetti indicati sopra sono generici e non prendono in considerazione gli adattamenti specializzati che permettono agli organismi di sfruttare caratteristiche specifiche dell'ecosistema. Per esempio, i batterioplancton sembrano essere per lo più cellule solitarie che si muovono nella colonna d'acqua, ma esiste un sottoinsieme di gruppi microbici marini che si specializza nella colonizzazione di particelle. Queste cellule hanno un significato geochimico particolare perché le particelle affondano, portando con sé carbonio e altri nutrienti dalla superficie nel mesopelagico. È questa l'essenza della pompa biologica, che in effetti riduce la pressione parziale del biossido di carbonio sulla superficie dell'oceano, e aumenta così la velocità con cui il CO2 atmosferico si dissolve nell'acqua di mare. Una classe di particelle particolarmente importante è costituita da aggregati macroscopici noti come neve di mare, che tende a formarsi nella zona fotica inferiore e scompare man mano che le particelle affondano nel mesopelagico superiore. Lo studio degli organismi associati con la neve di mare ha mostrato poca sovrapposizione tra questi organismi e le specie liberamente sospese nella colonna d'acqua. I membri dei phyla Planctobacteria e Bacteroidetes sono particolarmente abbondanti nelle particelle di neve di mare. La maggior parte dei Planctobacteria si sono specializzati in strutture di attaccamento alle superfici. I membri dei Bacteroidetes spesso usano una motilità scivolante per attraversare le superfici, e sono stati coinvolti nella degradazione dei biopolimeri associati al detrito. Il phylum Lentisphaerae, di recente scoperta, ha un fenotipo inusuale che sembra indicare che partecipi alla formazione delle particelle. I Lentisphaerae formano una rete di polisaccaridi che rende viscosa l'acqua. Non è stata ancora provata una funzione per questo fenomeno, ma l'ipotesi che esso serva a intrappolare le particelle che affondano sembra adattarsi bene alla localizzazione di questo organismo, che risiede nel mesopelagico superiore, appena al di sotto della zona fotica.
La mortalità virale e il rapporto predatore-preda dominano il pensiero attuale sulle interazioni tra specie di plancton microbico, ma è difficile credere che la colonna d'acqua non abbia generato altri meccanismi biologici insidiosi per ottenere un vantaggio competitivo. Alcuni studi recenti si sono concentrati sull'interazione tra batterioplancton e fitoplancton. Sono stati descritti diversi ceppi di batteri alghicidi che producono peptidi alghicidi, agenti anti-fouling o peptidi antimicrobici. Il genere Pseudoalteromonas appare spesso in questo contesto. Tuttavia, per ora, il modello passivo di catena microbica, in cui il plancton microbico eterotrofo sfrutta il carbonio organico perduto dal fitoplancton per fuoriuscita o lisi, riflette la visione prevalente.
Le specie rare possono essere importanti
Alcuni organismi rari che non si trovano spesso nelle librerie di cloni di geni possono contribuire sostanzialmente ai processi geochimici oceanici. Per esempio, il tasso di fissazione dell'azoto è basso negli oceani perché l'azoto fissato viene riciclato efficacemente nella zona fotica, e altri nutrienti limitati prevengono l'espandersi di comunità di plancton. Tuttavia la fissazione dell'azoto ha un ruolo chiave nel sostenere la produttività sostituendo l'azoto fissato disperso dalla superficie oceanica. Con approcci indipendenti dalle colture, sono stati identificati diversi geni di nitrogenasi in acqua di mare: a partire da ciò, sono stati osservati nuovi cianobatteri unicellulari che sembrano contribuire significativamente alla fissazione dell'azoto negli oceani.
La coltura di specie chiave
Una coltura di nuovi microrganismi fa notizia se rappresenta gruppi microbici con un ruolo importante nell'ambiente. La maggiore attenzione dedicata alle colture sta producendo un flusso continuo di nuovi isolati da diversi gruppi di ricerca. Il valore delle colture è stato sottolineato dai problemi riscontrati dal gruppo di Venter nella ricostruzione dei genomi dell'insieme di dati del metagenoma del Mar dei Sargassi. Genomi completi ottenuti da colture sono ora considerati punti di riferimento cruciali per raccogliere dati sul metagenoma e, più importante ancora, i ceppi in coltura forniscono un modo di studiare la fisiologia cellulare e di testare ipotesi sorte dai dati della sequenza del genoma. Ancora una volta, gli organismi fototrofi ossigenici unicellulari marini offrono l'esempio migliore. La coltura delle specie marine Synechoccoccus e Prochlorococcus ha portato a grandi avanzamenti nella conoscenza della biologia di questi organismi. Un altro gruppo che è stato studiato in coltura è il clade Roseobacter. Si è dimostrato che un membro del clade Roseobacter, il Sulfitobacter, utilizza dimetilsolfoniopropionato (DMSP) e può produrre energia metabolica attraverso l'ossidazione del solfuro. Un altro cianobatterio che per decenni si è sottratto alla coltura, il Trichodesmium, è ora in coltura in vari laboratori intorno al mondo.
Il processo di isolare cellule per diluizione (con colture d'estinzione) in acqua di mare naturale è stato iniziato da Davina K. Button e suoi collaboratori, e in seguito è stato utilizzato per ottenere il Sphingopyxis alaskensis e il Marinobacter arcticus, che sono diventati organismi modello per lo studio delle proprietà delle cellule oligotrofiche. Versioni di colture d'estinzione ad alto output hanno portato all'isolamento di molte altre specie oligotrofiche, la maggior parte delle quali non replica o replica male il nutriente marino agar. Alcuni degli isolati importanti che emergono da questo approccio sono il Pelagibacter ubique, il primo membro in coltura del clade SAR11, e anche l'OM43, il Lentisphaera e il gruppo oligotrofico marino γ-proteobatterio. Tuttavia, molti dei gruppi di plancton marino dominanti, compresi il cluster SAR86, SAR202, SAR324 l'actinobatterio marino, il SAR406 e l'Archaea del gruppo marino II, non sono ancora stati coltivati. Dato il numero di colture ad absorbimento di acqua di mare ormai esaminate, è chiaro che alcuni dei cladi principali che ancora non sono stati coltivati, non potranno crescere senza ulteriori sviluppi delle tecniche di coltura.
Prospettive future
Storicamente l'oceanografia biologica si è concentrata sullo sviluppo di modelli quantitativi di dinamiche della rete alimentare. Con l'emergere dell'ipotesi della catena microbica nel 1983, i batteri hanno assunto un ruolo principale nei modelli di rete alimentare, ma sono stati trattati come una scatola nera nella creazione di modelli flussi geochimici. A livello concettuale la scatola nera microbica è stata trasformata ripetutamente da scoperte di nuovi microrganismi fotosintetici e da nuove forme di fotoeterotrofia. Se le tradizioni dell'oceanografia si uniranno mai alle nuove correnti della microbiologia, allora i futuri modelli metabolici per i principali cladi di batterioplancton potranno diventare elementi di modelli più grandi per la previsione dei flussi geochimici nella colonna d'acqua oceanica.
bibliografia
Acinas 2004: Acinas, Silvia G. e altri, Fine-scale phylogenetic architecture of a complex bacterial community, "Nature", 430, 2004, pp. 551-554.
Béjà 2000: Béjà, Oded e altri, Bacterial rhodopsin: evidence for a new type of phototrophy in the sea, "Science", 289, 2000, pp. 1902-1906.
Carlson 2002: Carlson, Craig A. e altri, Effect of nutrient amendments on bacterioplankton production, community structure, and DOC utilization in the northwestern Sargasso Sea, "Aquatic microbial ecology", 30, 2002, pp. 19-36.
Cho 2004: Cho, Jang Cheon e altri, Lentisphaera araneosa gen. nov., sp. nov., a transparent exopolymer producing marine bacterium, and the description of a novel bacterial phylum, Lentisphaerae, "Environmental microbiology", 6, 2004, pp. 611-621.
DeLong, Karl 2005: DeLong, Edward F. - Karl, David M., Genomic perspectives in microbial oceanography, "Nature", 437, 2005, pp. 336-342.
Ducklow 2000: Ducklow, Hugh, Bacterioplankton production and biomass in the oceans, in: Microbial ecology of the oceans, edited by David L. Kirchman, New York, Wiley-Liss, 2000, pp. 85-120.
Eder 2002: Eder, Wolfgang e altri, Prokaryotic phylogenetic diversity and corresponding geochemical data of the brine-seawater interface of the Shaban Deep, Red Sea, "Environmental microbiology", 4, 2002, pp. 758-763.
Fuhrman 1992: Fuhrman, Jed A. - McCallum, Kirk - Davis, Alison A., Novel major archaebacterial group from marine plankton, "Nature", 356, 1992, pp. 148-149.
Karner 2001: Karner, Markus B. - DeLong, Edward F. - Karl, David M., Archaeal dominance in the mesopelagic zone of the Pacific Ocean, "Nature", 409, 2001, pp. 507-510.
Kolber 2000: Kolber, Zbigniew S. e altri, Bacterial photosynthesis in surface waters of the open ocean, "Nature", 407, 2000, pp. 177-179.
Moran 2004: Moran, Mary Ann e altri, Genome sequence of Silicibacter pomeroyi reveals adaptations to the marine envir-onment, "Nature", 432, 2004, pp. 910-913.
Rocap 2003: Rocap, Gabrielle e altri, Genome divergence in two Prochlorococcus ecotypes reflects oceanic niche differentiation, "Nature", 424, 2003, pp. 1042-1047.
Suttle 2005: Suttle, Curtis, Viruses in the sea, "Nature", 437, 2005, pp. 356-361.
Venter 2004: Venter, Craig J. e altri, Environmental genome shotgun sequencing of the Sargasso Sea, "Science", 304, 2004, pp. 66-74.