crossing-over
crossing-over
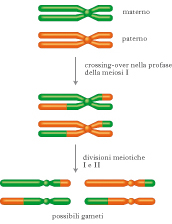
Scambio reciproco di segmenti di DNA fra i componenti di una coppia di cromosomi omologhi durante la meiosi, da cui risulta la ricombinazione di geni materni e paterni nella progenie. Durante la profase della prima divisione meiotica gli omologhi materno e paterno di ciascuna coppia di cromosomi si appaiano formando strutture transitorie, dette bivalenti, costituite da quattro cromatidi e situate sulla piastra metafasica. È durante questo appaiamento che avviene il processo di ricombinazione del c., che comporta la rottura della doppia elica in uno dei cromatidi di origine paterna e in uno dei cromatidi di origine materna, e lo scambio reciproco di segmenti cromosomici, in modo casuale. Questo processo, insieme all’assortimento indipendente dei cromosomi omologhi che avviene durante l’anafase, è alla base della grande variabilità genetica dei gameti prodotti da ciascun individuo.
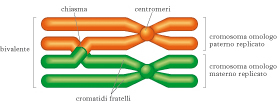
Appaiamento dei cromosomi omologhi
Durante il c. i cromosomi che formano i bivalenti sono molto condensati. Il meccanismo che permette l’appaiamento degli omologhi non è noto, ma si ritiene che questa disposizione sia necessaria affinché avvenga il crossing-over. La connessione (osservabile al microscopio) tra i cromosomi omologhi nel punto in cui avviene il c. si chiama chiasma e corrisponde allo scambio di segmenti fra due cromatidi non fratelli di un bivalente. In questo stadio della meiosi ogni coppia di cromosomi è tenuta insieme da almeno un chiasma e in molti bivalenti ne è visibile più di uno. Si pensa che i chiasmi abbiano anche una funzione strutturale necessaria per la segregazione, in quanto mantengono uniti sul fuso i cromosomi omologhi fino all’anafase della prima divisione meiotica.
Crossing-over diseguale
Di solito il c. avviene tra sequenze di DNA identiche o molto simili, dando luogo a eventi di ricombinazione omologa. In alcuni casi può verificarsi un c. diseguale, ossia la ricombinazione tra sequenze non alleliche, su cromatidi non fratelli, di una coppia di cromosomi omologhi. Spesso le sequenze in cui avviene il c. diseguale presentano un’omologia considerevole, che stabilizza l’appaiamento errato dei cromosomi: di solito si tratta di regioni con sequenze ripetute in tandem e il loro scambio reciproco ha come risultato una delezione su un cromatidio e una inserzione sull’altro, con possibili conseguenze patogene.
Frequenza di ricombinazione
Un c. che si verifichi tra due loci darà origine a due cromatidi ricombinanti, mentre gli altri due cromatidi della coppia di omologhi rimangono invariati. Tuttavia, poiché la frequenza di ricombinazione è funzione della distanza fra due loci, il c. separa raramente due loci che si trovano molto vicini tra loro. Quindi, gruppi di alleli di loci contigui (aplotipi) tendono a essere trasmessi in blocco lungo le generazioni di un dato albero genealogico. Le ricerche più recenti hanno suggerito che la ricombinazione non sia casuale a livello di DNA, ma che i cromosomi umani siano costituiti da blocchi conservati, lunghi tipicamente 20÷50 kb, separati da regioni dette punti caldi di ricombinazione.