Editing dell'RNA
Editing dell'RNA
L'espressione editing dell'RNA è usata per descrivere una serie di fenomeni mediante i quali le sequenze degli mRNA, degli rRNA e dei tRNA vengono modificate dopo la trascrizione. I diversi tipi di editing dell'RNA variano dall'inserimento o delezione di uridina (U) o citosina (C) nelle regioni codificanti degli mRNA mitocondriali dei tripanosomi alla sostituzione di specifici residui di citosina con residui di uridina negli mRNA mitocondriali delle piante, fino alla sostituzione di specifici residui di adenina (A) con residui di inosina (I) negli mRNA codificati nel nucleo delle cellule dei mammiferi. Le modificazioni sono spesso regolate e hanno significative conseguenze fenotipiche poiché introducono un ulteriore elemento di versatilità nei numerosi meccanismi di modulazione dell'espressione genica e della sintesi proteica operanti nel contesto di ogni cellula eucariotica.
La scoperta dell'editing dell'RNA e il dogma centrale della genetica molecolare
Una delle assunzioni di base della genetica molecolare è che la sequenza nucleotidica dell'RNA messaggero (mRNA) sia ma copia perfetta della sequenza del corrispondente DNA, come conseguenza della regola dell'appaiamento delle basi. La prima sfida a questa idea è venuta dalla scoperta delle sequenze non codificanti, contenute all'interno dei geni jegli organismi superiori, che vengono rimosse con precisione dall'mRNA mentre i frammenti di RNA codificanti, jetti esoni, vengono ricongiunti per costituire la sequenza codificante completa. Questa scoperta, in realtà, non è in contraddizione con il principio che le sequenze di mRNA siano una copia del DNA corrispondente, in quanto gli esoni sono effettivamente delle copie perfette.
Tuttavia dalla metà degli anni Ottanta, in seguito a esperimenti compiuti su un antico gruppo di protozoi flagellati Jarassiti, detti cinetoplastidi, sono state ottenute prove sperimentali che indicano che la sequenza nucleotidica degli mRNA può essere modificata in modo consistente dopo la trascrizione; questo fenomeno è noto con il nome di editing dell'RNA. Inizialmente questi risultati hanno spinto alcuni ricercatori a mettere in discussione il dogma centrale della genetica molecolare, secondo cui l'informazione genetica si trasferisce dal DNA all'RNA e viceversa, e dall'RNA alle proteine. Infatti, l'informazione genetica per i cambiamenti della sequenza degli mRNA, e quindi anche la sequenza amminoacidica delle proteine, non sembrava essere codificata dagli acidi nucleici dell'organismo. Oggi questi risultati possono essere spiegati in termini di un modello che non contraddice il dogma centrale ma che comunque mostra caratteristiche nuove e interessanti.
Sono stati descritti, in altri organismi, altri esempi di modificazioni della sequenza degli RNA non codificate dagli acidi nucleici e sono stati anch'essi denominati editing dell'RNA, sebbene i meccanismi sembrino essere piuttosto diversi. In alcuni casi, la sito-specificità degli eventi di editing può essere spiegata assumendo che esistano alcune proteine capaci di riconoscere specifiche sequenze o strutture dell'RNA, ma in altri casi la sito-specificità è ancora un mistero.
Una storia personale dello studio dell'editing dell'RNA nei tripanosomi
l tripanosomi appartengono a un vasto gruppo di protozoi parassiti noti come protozoi cinetoplastidi. Il termine tripanosoma o tripanosomatide è in effetti il nome delle cellule che, all'interno della famiglia, appartengono al genere Trypanosoma, ma viene genericamente adoperato per descrivere qualunque protozoo cinetoplastide. Il nome cinetoplastide scaturisce dalle osservazioni dei primi studiosi che avevano rilevato un piccolo granulo, o cinetoplasto, alla base del flagello, quando le cellule venivano trattate con certi coloranti. In realtà il cinetoplasto rappresenta una porzione dell'unico e complesso mitocondrio presente in questo tipo di cellule; esso contiene una enorme massa compatta di DNA mitocondriale, che è proprio ciò che viene evidenziato con la colorazione.
I tripanosomi sono repellenti e affascinanti allo stesso tempo. Molte specie sono pericolosi parassiti dell'uomo, in grado di causare malattie come il morbo di Chagas, la malattia del sonno africana e la leishmaniosi, un'ulcerazione della pelle che in alcuni casi può estendersi a livello della bocca e del naso causando danni considerevoli. Non esistono vaccini per nessuna di queste malattie e i trattamenti, quando possibili, non sono mai molto soddisfacenti. In Africa, i bovini d'allevamento (ma non quelli selvatici!) muoiono per una specie di tripanosoma trasmessa dalla mosca tse-tse, e persino le palme da cocco sono colpite da un tipo di tripanosoma chiamato Phytomonas. Di fatto, i primi lavori sui tripanosomi sono stati condotti in gran parte da medici interessati a queste malattie. Comunque, molte specie di tripanosomi sono parassiti solo degli insetti e non hanno rilevanza dal punto di vista medico; altre invece sono parassite sia degli insetti sia dei vertebrati.
I tripanosomi sono affascinanti per i moderni biologi cellulari e molecolari in quanto rappresentano una delle più primitive linee di discendenza delle cellule eucariotiche. Probabilmente è per questa ragione che possiedono una serie di caratteristiche davvero insolite, che non si riscontrano in altre cellule eucariotiche. Per esempio, i tripanosomi hanno un solo mitocondrio che contiene un tipo singolare di DNA mitocondriale noto come DNA cinetoplastidico. Tutti gli organismi eucarioti unicellulari odierni, i cui antenati si originarono prima di quelli dei tripanosomi (come per esempio Giardia o Trichomonas), mancano di mitocondri e quindi non respirano ossigeno. È possibile che la linea evolutiva da cui, circa un miliardo di anni fa, si sono originati i protozoi flagellati euglenozoi (tra cui i protozoi cinetoplastidi ed Euglena, un protozoo flagellato verde) fosse strettamente correlata a quella cellula ancestrale primitiva che inglobò una cellula batterica aerobica, dando origine all'organulo intracellulare obbligato, oggi noto con il nome di mitocondrio.
Il DNA del cinetoplasto
Il mio interesse per l'insolito mitocondrio dei tripanosomi ebbe inizio quando stavo conseguendo il dottorato. A quel tempo tutto quello che sapevamo era che questo mitocondrio contiene una grande massa compatta di DNA, nota come DNA del cinetoplasto, che si poteva ben evidenziare con certi coloranti. Il nostro sistema modello era il tripanosoma non patogeno Leishmania tarentolae che in origine era parassita di un geco. Ben presto sia noi sia altri gruppi dimostrammo che questo DNA è costituito da migliaia (5000÷lO.000) di piccole molecole circolari di DNA, i minicircoli, tutte concatenate come anelli di una catena a formare una rete gigante di DNA, e da un piccolo numero (20 ÷ 50) di molecole circolari più grandi, i maxicircoli, anch'esse legate alla rete (Simpson e Da Silva, 1971). I minicircoli, sebbene identici per dimensione in ognuna delle specie, sono costituiti da molecole a sequenza diversa persino all'interno della stessa rete. Le loro dimensioni variano da 460 a 2500 paia di basi (bp) nelle diverse specie e ogni minicircolo è organizzato in una o più regioni conservate e regioni variabili. Una regione del minicircolo è intrinsecamente piegata (Marini et al., 1982), ma il ruolo genetico di tale struttura non è chiaro. I minicircoli non sembravano codificare informazioni per la sintesi di proteine ma di certo si replicavano molto attivamente, e hanno tenuto impegnati per anni numerosi laboratori nel tentativo di chiarire i meccanismi della replicazione e della segregazione della rete (Ryan et al., 1988; Shapiro e Englund, 1995). La funzione genetica dei minicircoli è rimasta a lungo un mistero e non è stata risolta fino alla scoperta degli RNA guida, nel 1990 (Blum et al., 1990).
La molecola del maxicircolo, le cui dimensioni variano da 23.000 a 36.000 bp nelle diverse specie, rappresenta la molecola di DNA che porta l'informazione del mitocondrio e contiene molti degli stessi geni trovati nelle molecole di DNA mitocondriale umano: gli RNA ribosomali mitocondriali maggiore e minore, tre subunità della citocromoossidasi, il citocromo b, diverse subunità della NADH-deidrogenasi, una subunità della ATPasi Fl-FO e varie altre proteine non ancora identificate. Tutti i geni strutturali identificati sono coinvolti nei processi di trasporto elettronico e di fosforilazione ossidativa a livello della membrana interna dell'organulo, come avviene nelle cellule umane.
Le prime indicazioni sui geni criptici
Verso la metà degli anni Ottanta è stata fatta la sconcertante scoperta che molti geni trovati nel genoma mitocondriale umano e di lievito sono apparentemente assenti nei mitocondri del tripanosoma, come per esempio i geni per i tRNA mitocondriali. L'apparente assenza dei geni per i tRNA era davvero sorprendente, in quanto tutti i mitocondri studiati fino ad allora contenevano dei tRNA coinvolti nella sintesi delle proteine mitocondriali. Per verificare se alcuni dei minicircoli o alcune regioni dei maxicircoli che non erano ancora state sequenziate codificassero dei tRNA, ibridammo il DNA marcato di minicircolo e maxicircolo con RNA di piccola massa molecolare isolato dai mitocondri di L. tarentolae, che era stato separato per elettroforesi su gel di acrilammide e trasferito su un filtro (Simpson et al., 1989). Con nostra sorpresa, l'unico RNA che si ibridava era un insieme di 20 ÷ 30 bande - ciascuna delle quali differiva in lunghezza dalle altre per un nucleotide che migravano molto oltre gli abbondanti tRNA mitocondriali, a indicare che questi RNA erano persino più piccoli dei tRNA. Queste bande erano una componente talmente minima dell'RNA isolato che non potevamo nemmeno vederle colorando il geli Oggi sappiamo che si trattava della prima osservazione degli RNA guida che avevano contaminato le nostre preparazioni di RNA, ma a quel tempo non avevamo idea di cosa fossero quei trascritti. Tuttavia era chiaro che nessun tRNA mitocondriale veniva codificato nel DNA mitocondriale e sembrava quindi probabile che questi tRNA venissero importati nel mitocondrio del tripanosoma dal citoplasma, come era stato precedentemente suggerito per Tetrahymena da Y. Suyama (Chiu et al., 1975). Per quei tempi questa era un'idea eretica, alla quale oggi si guarda con più favore grazie all'esistenza di evidenze sperimentali, sia in vivo sia in vitro, che dimostrano che i tRNA vengono importati nel mitocondrio in diversi organismi (Tarassov et al., 1995; Hauser e Schneider, 1995; Lima e Simpson, 1996).
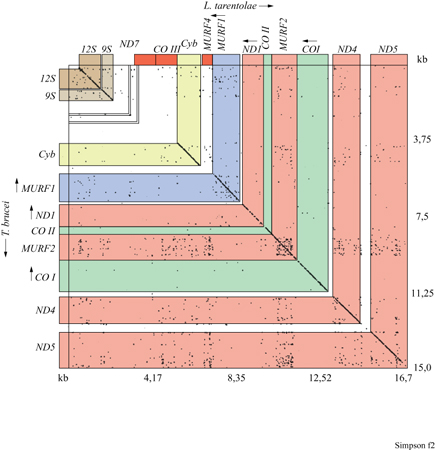
Vi erano numerosi altri segnali che suggerivano l'esistenza di qualcosa di insolito in questo genoma mitocondriale: due geni (COII e ND 7) sembravano avere un nucleotide in più o in meno che creava uno spostamento del modulo di lettura che, se non corretto, avrebbe potuto causare la terminazione della traduzione (Hensgens et al., 1984; De La Cruz et al., 1984). Si dimostrò che questi spostamenti più uno o meno uno sono localizzati nelle stesse posizioni relative sui geni di tre specie di tripanosoma appartenenti a tre generi diversi, che sapevamo essere separati da almeno 100 milioni di anni di storia evolutiva. Un altro problema era che numerosi geni erano privi dei codoni AuG codificanti metionina per l'inizio della traduzione. Infine, nonostante la localizzazione relativa dei geni nei maxicircoli di L. tarentolae e Trypanosoma brucei fosse molto simile (fig. 2), tre dei geni presenti in L. tarentolae mancavano nel maxicircolo di T. brucei (ND7, COllI e A6=MURF4). Questi geni erano sostituiti da sequenze più corte relativamente ricche di guanina (G) (Simpson et al., 1987).
La scoperta dell'editing
Nel 1986 il problema fu risolto, ma si aprì un altro vaso di Pandora pieno di problemi quando R. Benne pubblicò una sequenza dell'mRNA di COlI corrispondente alla regione conservata contenente lo spostamento del modulo di lettura, e trovò quattro uridine (O) in più che non erano codificate dal DNA del maxicircolo (Benne et al., 1986)! La presenza di queste quattro U inserite in tre siti annullava gli effetti dello spostamento di tipo meno uno, consentendo la traduzione dell'mRNA. Ben presto vennero scoperti molti altri casi di editing dell'RNA anche più eclatanti. Per esempio, si scoprì che l'mRNA di Cyb era sottoposto a editing alla sua estremità 5', per inserimento di 39 U in 15 siti, aggiungendo così 20 nuovi amminoacidi all'estremità amminica della proteina, tra cui un AuG (corrispondente a metionina) per l'inizio della traduzione (Feagin et al., 1988a). Inoltre, sebbene con minore frequenza, in certi geni si osservò l'eliminazione di alcune U, come per esempio in COllI di L. tarentolae (Shaw et al., 1988). Per descrivere i geni i cui trascritti subiscono editing nelle regioni codificanti, venne coniato il termine criptogeni, o geni criptici, mentre le regioni dell'mRNA che devono essere sottoposte a editing vennero dette regioni pre-edited (Simpson e Shaw, 1989).
Nel 1988 J. Feagin scoprì in T. brucei il gene COllI, fino ad allora ritenuto assente. In realtà il gene era sempre stato presente, ma era un gene criptico ben nascosto, in quanto il trascritto era soggetto a un editing così esteso, con aggiunta di centinaia di U su tutta la lunghezza, che l'mRNA maturo così modificato misurava quasi il doppio dell'intero gene (Feagin et al., 1988b). Nel laboratorio di K. Stuart si scoprì che ciò accadeva anche per gli altri due geni mancanti di T. brucei (Koslowsky et al., 1990; Bhat et al., 1990). Decidemmo di chiamare questo tipo di editing così estensivo pan-editing (pan = tutto), per differenziarlo dal semplice editing interno del gene COlI e dall'editing dell'estremità 5' del gene Cyb.
La polarità dell'editing
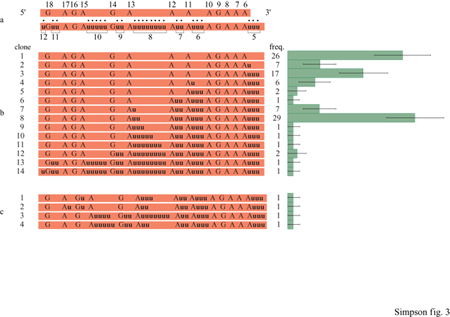
Si riuscì frnalmente a vedere un po' più chiaro nei meccanismi dell'editing quando K. Stuart scoprì che il pan-editing dell'mRNA di COllI di T. brucei sembrava progredire dall'estremità 3' a quella 5' del trascritto (Abraham et al., 1988). Questa osservazione suggeriva che il processo dovesse avvenire dopo la trascrizione, in quanto la trascrizione procede dall'estremità 5' a quella 3', e spiegava anche perché messaggeri che avessero subito editing parziale non riuscivano a essere tradotti: il sito di legame al ribosoma è l'ultima sequenza creata dall'editing. Scoprimmo che lo stesso fenomeno si verificava nei geni di L. tarentolae che subiscono editing all'estremità 5' (Sturm e Simpson, 1990a). Vennero sequenziati più di 400 mRNA con editing parziale, derivanti dai geni Cyb e COlI I, generati per amplificazione con il metodo della reazione a catena della polimerasi (peR, Polymerase Chain Reaction) a partire da RNA mitocondriale, e si scoprì che la regione compresa tra l'estremità 3', a editing completo, e quella 5', che non aveva subito alcun editing, presentava numerose configurazioni di editing parziale. Tale regione venne chiamata regione di giunzione. Nel caso degli RNA prodotti dal gene Cyb, quasi tutte queste configurazioni potevano essere ordinate secondo una precisa direzionalità dell'editing, dall'estremità 3' verso quella 5' (fig. 3).
Tuttavia, nel caso di COIlI solo il 58% delle diverse configurazioni di editing mostrava questa precisa polarità. Il resto mostrava delle modalità di editing inatteso, in cui delle V venivano aggiunte in siti non soggetti a editing nel trascritto maturo, oppure nella regione 5' prima che fossero aggiunte in quella 3'. C.J. Decker e B. Sollner-Webb (1990) esaminarono gli mRNA soggetti a editing parziale dei geni Cyb e COllI di T. brucei e trovarono in entrambi i geni una elevata percentuale di editing inattesi nelle regioni di giunzione. Quando ci si rese conto che l'editing avveniva dopo la trascrizione ed era polarizzato e progressivo, si ipotizzò che dovesse essere in atto un meccanismo di taglio e giunzione dei trascritti. Comunque, il problema principale era dato dal fatto che non sembrava esistere uno stampo di acido nucleico per questa aggiunta di informazione. Sembrava che l'informazione di sequenza derivasse dal nulla e che, nel caso dei geni a pan-editing, si avesse la costruzione di geni del tutto nuovi. Queste scoperte avevano quindi profonde implicazioni sul dogma centrale del trasferimento dell'informazione genetica.
La scoperta degli RNA guida
In ogni caso, non abbandonammo il dogma centrale che aveva continuato a mantenersi valido per così tanti anni. Venne effettuata una semplice ricerca al computer sul DNA noto del maxicircolo di L. tarentolae, in cerca di sequenze brevi di DNA che potessero dare origine a RNA in grado di ibridarsi con sequenze, sia intere sia parziali, di RNA modificati dall'editing. Oltre ai classici appaiamenti di I.D. Watson e F.H.C. Crick, C-G e A-V, decidemmo di ritenere valide anche le coppie di basi G-V, che negli rRNA e nei tRNA rappresentano appaiamenti possibili, e proprio questo si rivelò essere il trucco!
Con questa ricerca al computer, effettuata su tutto il maxicircolo tra i suoi geni noti, vennero subito scoperte sette brevi sequenze di questo tipo per quattro dei geni che subivano editing (Blum et al., 1990). Ottenemmo presto prove definitive dell'esistenza di piccoli RNA nel mitocondrio, derivati dalla trascrizione di queste sequenze. Questi RNA contenevano anche delle sequenze alla loro estremità 5' che potevano formare regioni duplex appaiando si con l'mRNA subito a valle delle regioni pre-edited. Abbiamo chiamato queste sequenze regioni di ancoraggio, in quanto forniscono una maniera ideale per ancorare l'RNA guida (gRNA) all'mRNA, formando un ibrido a doppio filamento nella regione appena successiva a quella che deve subire editing (fig. 4).
Questi piccoli RNA avevano una mobilità elettroforetica insolita, migrando come 20 ÷ 30 bande ognuna delle quali possedeva un nucleotide in più delle altre. Ci siamo subito resi conto che questi erano gli stessi RNA che due anni prima avevano contaminato le nostre preparazioni di tRNA (Simpson et al., 1989)! Presto ci siamo accorti che l'insolita mobilità elettroforetica era dovuta alla presenza di code di oligo-V non codificate, lunghe fino a 24 nucleotidi, alle estremità 3' delle molecole di RNA (Blum e Simpson, 1990). Abbiamo chiamato queste molecole RNA guida, o gRNA, in quanto contengono l'informazione di sequenza per l'editing. Gli RNA guida sono così giunti in soccorso del dogma centrale! Naturalmente eravamo molto eccitati, ma allo stesso tempo un po' delusi dalla scoperta che la risposta ai segreti dell'editing non era in realtà niente di completamente nuovo e sconvolgente, bensì qualcosa che obbediva alle semplici leggi dell'appaiamento tra le basi. Tuttavia dovevamo ancora spiegare come facessero questi gRNA a operare l'editing delle molecole di mRNA.
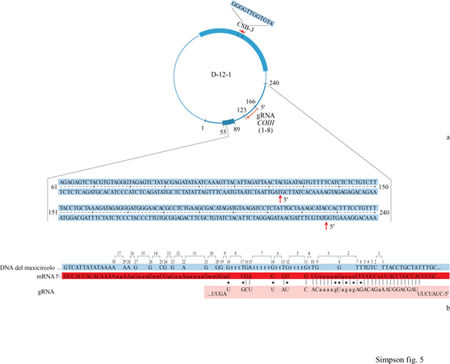
Fino ad allora avevamo identificato sette geni per gRNA sparsi lungo tutto il genoma del maxicircolo che non avevano alcuna relazione con i criptogeni, nell'editing dei quali sono coinvolti. Questi gRNA contenevano l'informazione per la modifica di quattro dei cinque trascritti dei criptogeni noti ma non riuscivamo a trovare un gRNA per il trascritto del gene COIlI di L. tarentolae, che subisce editing all'estremità 5'. Poiché si sapeva che il DNA dei minicircoli è capace di ibridare con dei piccoli trascritti caratterizzati dalla stessa insolita mobilità elettroforetica dei gRNA, vennero esaminate le sequenze note dei minicircoli e fu così trovato, nel minicircolo Dl2 (fig. 5), un gene per un gRNA contenente informazioni di sequenza per i primi otto siti di editing dell'mRNA di COllI (Sturm e Simpson, 1990b).
Questa era la prima indicazione di una funzione genetica per il minicircolo, che spiegava l'eterogeneità di sequenza osservata in queste molecole di DNA: ogni classe di sequenze codifica nella regione variabile, per un gRNA diverso! In totale, nel ceppo di laboratorio di L. tarentolae dell'università della California, vennero identificate, per clonaggio e sequenziamento, 17 diverse classi di sequenza dei minicircoli, ognuna presente in quantità diversa (Masio v e Simpson, 1992).
Presto vennero trovati dei gRNA codificati dai minicircoli anche in T. brucei (Corell et al., 1993; Pollard et al., 1990). La principale differenza osservata sta nel fatto che ogni minicircolo di T. brucei codifica tre gRNA diversi piuttosto che un singolo gRNA e che i geni corrispondenti sono localizzati nella regione variabile, quasi perfettamente in alternanza con tre gruppi di ripetizioni invertite di 18 nucleotidi che non sono presenti in L. tarentolae (fig. 6). Un'altra differenza è che in precedenza era stata dimostrata l'esistenza di più di trecento diverse classi di sequenza dei minicircoli in T. brucei, invece del limitato numero riscontrato in L. tarentolae. Questo significa che il numero complessivo di diversi gRNA in T. brucei può essere di oltre 900. Una caratteristica dei gRNA di T. brucei è la loro elevata ridondanza. l gRNA ridondanti possiedono sequenze diverse, ma contengono la stessa informazione per l'editing; ciò è dovuto alla possibilità di appaiamento tra le basi G-U. Di recente è stata osservata anche in T. cruzi un'alta percentuale di gRNA ridondanti (Avila e Simpson, 1995).
La perdita dei minicircoli durante il periodo di crescita in coltura e la perdita dell'editing
In seguito a un primo confronto tra i genomi mitocondriali d L. tarentolae e T. brucei, avevamo notato l'esistenza di numerosi tratti di DNA relativamente ricchi di residui di G (Simpson et al., 1987). In T. brucei tre di questi, chiamati ND7, COllI e MURF4, si rivelarono essere tre criptogen nascosti i cui trascritti sono soggetti a pan-editing, ma ir entrambe le specie vi erano altre sei regioni ricche di G localizzate tra geni noti. In L. tarentolae il trascritto derivate dalla sesta di queste regioni subisce pan-editing, per aggiunte di 117 U in 49 siti ed eliminazione di 32 U in 13 siti raggruppati in tre domini di editing, dando origine a un mRNA che codifica una proteina della subunità minore del ribosoma mitocondriale (Maslov et al., 1992).
È stato dimostrato che anche le altre regioni ricche di G in T. brucei (chiamate G l-G 5) sono criptogeni i cui trascritti sono soggetti a pan-editing ma, nel vecchio ceppo di L. tarentolae in uso nei laboratori dell'università della Califomia, non furono mai identificati dei trascritti di questi geni che avessero subito editing completo. È stato poi dimostrato che gli mRNA maturi di T. brucei derivati da Gl, G 2 e G 5 codificano le subunità dell'NADH-deidrogenasi (Bhat et al., 1990; Souza et al., 1992; Read et al., 1992; Souza et al., 1993).
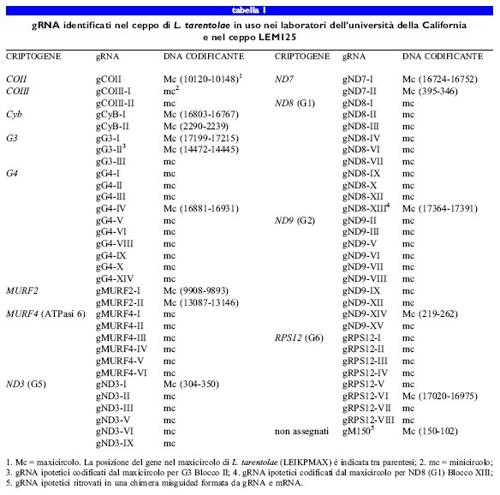
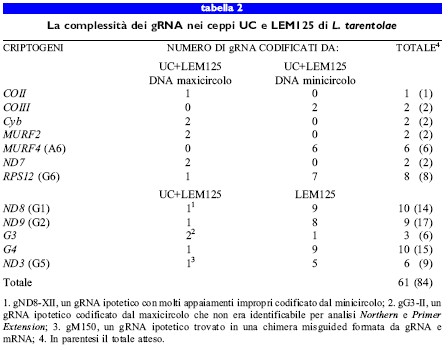
L'analisi del ceppo LEM125 di L. tarentolae recentemente isolato ha suggerito che il ceppo adoperato nei laboratori dell 'università della Califomia avesse in realtà un difetto genetico per quanto riguarda l' editing dei trascritti dei criptogeni da Gl a G5 (Thiemann et al., 1994). Il ceppo LEM125 conteneva mRNA completamente maturi corrispondenti ai geni da Gl a G5, oltre a un repertorio più complesso di gRNA codificati dai minicircoli. Nel ceppo LEM125 furono individuati almeno altri 32 gRNA codificati dai minicircoli, assenti nel ceppo utilizzato all'università della California (tabb. 1, 2). Abbiamo ipotizzato che specifiche classi di sequenza dei minicircoli, codificanti i gRNA implicati nei processi di editing dei trascritti dei geni da Gl a G5, fossero andate perdute durante il lungo periodo di mantenimento in coltura del ceppo di laboratorio dell'università della California, probabilmente a causa della mancata necessità dei rispettivi prodotti proteici nella fase del ciclo biologico di Leishmania mantenuta in coltura.
Modelli del meccanismo di editing del tipo inserimento/delezione di U
Prima della scoperta dei gRNA, avevamo isolato, da mitocondri purificati di L. tarentolae, una terminaluridiltransferasi (TUTasi), un enzima in grado di aggiungere delle U all'estremità 3' di qualunque molecola di RNA (Bakalara et al., 1989). Questo enzima era presumibilmente responsabile dell' aggiunta di U all'estremità 3' dei gRNA. Avevamo anche dimostrato la presenza di una RNA-ligasi mitocondriale in grado di legare insieme in modo covalente due molecole di RNA, e avevamo osservato che se la TUTasi dell'estratto mitocondriale veniva inibita dall'eparina o degradata per digestione con proteinasi K, si metteva in azione un'attività ribonucleasica criptica che tagliava l'mRNA di Cyb già sottoposto a pre-editing, proprio all'interno della regione modificata (Simpson et al., 1992). Una attività endonucleasica simile era stata individuata anche in estratti mitocondriali di T. brucei (Harris et al., 1992).
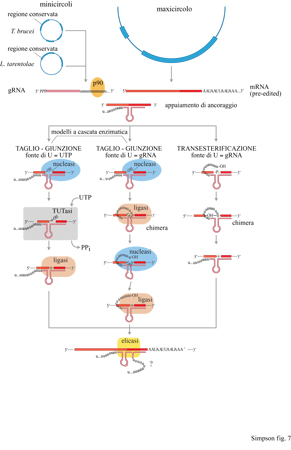
Basandosi sull' esistenza nel mitocondrio di queste attività enzimatiche e sulla progressione in direzione 3'-5' del meccanismo di editing dell'mRNA, è stato suggerito un modello per spiegare il ruolo dei gRNA nel processo di editing (Blum et al., 1990). Tale modello, poiché postulava una serie di reazioni che accadono in un complesso multienzimatico legato alI 'mRNA, è stato chiamato modello a cascata enzimatica. Abbiamo proposto che l'interazione iniziale coinvolgesse la formazione di un appaiamento di ancoraggio tra il gRNA e l'estremità 3' della regione pre-editing dell'mRNA. Oltre alle interazioni RNARNA coinvolte nell'ancoraggio, crediamo che alcuni fattori proteici recentemente trovati, che formano complessi con i gRNA (Byrne et al., 1995; Bringaud et al., 1995), assistano lo svolgimento di questa specifica interazione iniziale, forse riconoscendo delle strutture secondarie formate dallo stesso mRNA o dall'ibrido gRNA-mRNA. È stato ipotizzato che il passo successivo fosse un taglio specifico dell'mRNA all'altezza della prima base appaiata in modo non corretto, così da esporre un gruppo 3'-OH libero. Questo frammento di mRNA tagliato sarebbe un substrato per l'enzima TUTasi, che può aggiungervi una o più U al 3'-terminale. Le U aggiunte potrebbero quindi appaiarsi ai nucleotidi A o G del gRNA e le due estremità dell'mRNA potrebbero essere ricongiunte dalla RNA-ligasi. Questo meccanismo porterebbe a una sorta di apertura a cerniera della doppia elica in una direzione che va dal 3' al 5' (sull'mRNA), e l'intero processo inizierebbe di nuovo alla successiva base appaiata impropriamente (fig. 7).
Il modello a cascata enzimatica ha fornito una spiegazione per la polarità 3'-5' del pan-editing: tale polarità era dovuta alla creazione, a opera dei gRNA a valle, di sequenze di mRNA modificate e complementari alle sequenze di ancoraggio dei gRNA adiacenti a monte (fig. 8).
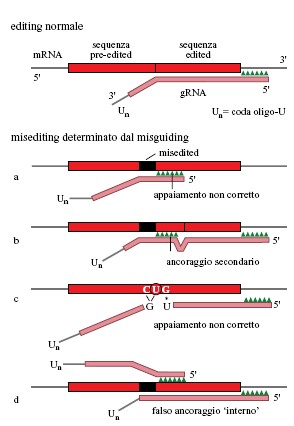
Il modello spiegava anche l'esistenza di configurazioni di editing inattese nelle regioni di giunzione degli mRNA parzialmente modificati. Abbiamo suggerito che queste diverse configurazioni rappresentassero il risultato di un normale editing effettuato però da gRNA sbagliati, oppure da gRNA appropriati in un sito o in un modulo di lettura sbagliati. Questi processi sono stati chiamati rispettivamente misediting (editing errato) e misguiding (guida errata), e sarebbero indotti dalla presenza di appaiamenti di basi 'vacillanti' (wobble) G-U e forse anche A-C (Sturm et al., 1992). La formazione di un appaiamento di ancoraggio a opera del gRNA sbagliato, o di un ancoraggio secondario in un sito sbagliato a opera del gRNA giusto, potrebbe dare origine a un editing inatteso, il quale a sua volta potrebbe interrompere il processo di editing perché non si è formata una sequenza di ancoraggio corretta per il successivo gRNA. In ogni caso, le sequenze che hanno subito misediting nella regioni di giunzione potrebbero essere nuovamente modificate con l'intervento del corretto gRNA. Sono stati in effetti osservati molti esempi di misediting/misguiding che avvalorano questa ipotesi (fig. 9).
Comunque è stata proposta anche un'altra interpretazione degli editing inattesi (Decker e Sollner-Webb, 1990): è stato suggerito che l' editing sia completamente casuale e avvenga per ciascun nucleotide all'interno di un dominio di editing, e che, una volta formatasi la sequenza corretta, questa venga fissata mediante appaiamenti con il gRNA. Questo problema non sarà del tutto risolto fino a quando non acquisiremo una conoscenza completa del contenuto complessivo di gRNA nel mitocondrio, così da confrontare tutte le possibili configurazioni inattese di editing con le sequenze di gRNA note. Il modello a cascata enzimatica si accorda con molte osservazioni sperimentali, compresa quella nota della polarità 3'-5' dell'editing, ma non spiega in modo soddisfacente l'esistenza della coda di oligo-D sui gRNA.lnizialmente è stato ipotizzato che il ruolo della coda di oligo-D fosse la stabilizzazione dell'ibrido iniziale, in quanto le D possono appaiarsi con le G e le A nella regione di pre-editing (Blum e Simpson, 1990). Tuttavia, nel 1991 abbiamo suggerito che la coda di oligo-D potesse svolgere un ruolo più attivo, in particolare che fosse la fonte delle D aggiunte durante l' editing (Blum et al., 1991). È stato proposto un modello secondo cui l'OH 3'-terminale del gRNA attaccherebbe un fosfato lungo l'mRNA, all'altezza della prima base appaiata in modo improprio tra gRNA e mRNA, producendo una transesterificazione e lo scambio dell'OH con il fosfato (vedi figura 7).
La chimica di questa reazione è simile a quella che si verifica in altre cellule durante il processo di autosplicing delle molecole di RNA. Dn modello analogo di transesterificazione è stato indipendentemente proposto anche da T.R. Cech (1991). Dna delle previsioni di questo modello riguarda l'esistenza di intermedi chimerici, formati da gRNA legati in modo covalente a molecole di mRNA nei siti di editing per mezzo della coda di oligo-U Abbiamo cercato e trovato queste molecole chimeriche per tre diversi geni (Blum et al., 1991). Questa scoperta è stata confortante, ma non ha realmente dimostrato il modello della transesterificazione, in quanto molecole chimeriche potevano essersi formate in altri modi, specialmente in un sistema che, come già sapevamo, contiene enzimi capaci di tagliare e legare insieme le molecole di RNA (vedi figura 7).
Dal punto di vista teorico il modello della transesterificazione era attraente, poiché richiede la stessa chimica e lo stesso tipo di sequenze guida adoperate nel ben compreso meccanismo di autosplicing degli introni, mentre il modello a cascata enzimatica prevede una nuova serie di reazioni. Tuttavia, di recente si sono avute diverse evidenze che indicano fortemente che le chimere rappresentano prodotti terminali abortivi piuttosto che intermedi delle reazioni. Tali evidenze sono state ottenute da un sistema di delezione in vitro delle D, mediata dai gRNA, in estratti mitocondriali di T. brucei (Seiwert et al., 1996), da uno studio della stereochimica di una attività di inserzione in vitro delle D indipendente dai gRNA (Frech e Simpson, 1996; Connell et al., 1997) e anche da una attività di inserzione in vitro delle D dipendente dai gRNA in estratti mitocondriali di L. tarentolae (Byrne et al., 1996). Queste osservazioni suggeriscono che il meccanismo coinvolge una reazione di taglio-giunzione mediata da proteine enzimatiche, come nel modello originale a cascata enzimatica, più che transesterificazioni mediate dall'RNA. Tuttavia, per un completo chiarimento del meccanismo, bisogna attendere l'isolamento, l'espressione e la ricostituzione delle componenti enzimatiche dell' apparato di editing.
L'evoluzione dell'editing del tipo inserimento/delezione di U
L'origine e l'evoluzione dell'editing dell'RNA nei tripanosomi per l'inserimento o la delezione di D è un argomento importante e interessante. Il pan-editing mediato dai gRNA è stato riscontrato in tutte le specie di tripanosomi finora analizzate (Fernandes et al., 1993; Landweber e Gilbert, 1994; Maslov et al., 1994). L'origine dell' editing si è potuta far risalire al progenitore dell'intera linea evolutiva dei cinetoplastidi con la scoperta dell' editing mediato dai gRNA in Trypanoplasma borreli, che appartiene ai bodonini, un altro sottordine di cinetoplastidi (Lukes et al., 1994; Maslov e Simpson, 1994). La presenza dell'editing nel mitocondrio di Euglena è ancora argomento di discussione, sebbene abbiamo dimostrato (Yasuhira e Simpson, 1997) che il trascritto dell 'unico gene mitocondriale di questa specie clonato fino a oggi, COI, non subisce editing e che in questo organismo non si possono mettere in evidenza molecole simili ai gRNA mediante il metodo di marcatura dell' estremità 5' con [α-³²P]GTP e guanililtransferasi del virus del vaiolo bovino.
Poiché l'analisi filo genetica delle proteine mitocondriali COI e Rsp60 (codificata nel nucleo) indica un' origine monofiletica dei mitocondri di Euglena e dei tripanosomi (Yasuhira e Simpson, 1996), una dimostrazione definitiva dell' assenza di editing nei mitocondri di Euglena indicherebbe che l'editing è una caratteristica acquisita durante l'evoluzione dei cinetoplastidi. La soluzione di questo problema deve attendere l'analisi di altri geni mito condriali di questo organismo.
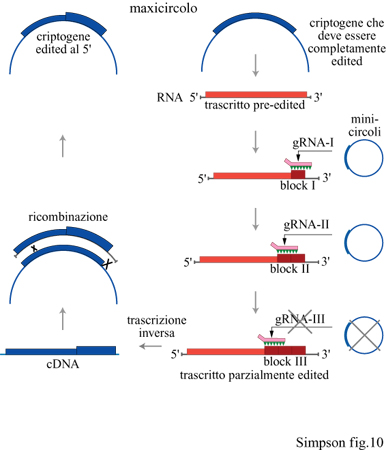
Nell' ambito della linea evolutiva dei cinetoplastidi (fig. 10), la limitazione dell'editing alla regione 5' di tutti i geni a editing parziale e la presenza di geni omologhi soggetti a pan-editing in specie correlate, suggeriscono che i criptogeni a editing limitato all'estremità 5', come i geni COllI e ND7 di Leishmania e Crithidia, siano derivati dalla retroposizione di cDNA corrispondenti a mRNA con editing parziale, che hanno sostituito i criptogeni originali nel genoma del maxicircolo (Simpson e Maslov, 1994). Ciò implica che questo tipo di editing dell'RNA è una caratteristica genetica instabile che può essere facilmente perduta nel corso dell'evoluzione. La perdita di molte classi di sequenza dei minicircoli codificanti i gRNA implicati nell' editing degli mRNA G1 ÷ G5, nella lunga storia di coltura del ceppo di laboratorio di L. tarentolae dell 'università della California, si accorda perfettamente con questa ipotesi (Thiemann et al., 1994). Ciò suggerisce che deve esservi stato un vantaggio selettivo nel mantenimento di questo sistema genetico nel corso dell'evoluzione di questo tipo di cellule. La pressione selettiva può essere correlata al fatto che l'editing, almeno nel complesso ciclo biologico di T. brucei, è regolato e, in questo tipo di cellule, viene utilizzato come meccanismo di controllo traduzionale per regolare la biosintesi del mitocondrio. Tuttavia questo non spiega il mantenimento dell'editing nei tripanosomi monogenetici parassiti degli insetti, come Crithidia, a meno che non esistano degli stadi non noti in cui si verifichino repressione e derepressione mitocondriale, come nei tripanosomi digenetici africani. Tutto ciò rimane per il momento in discussione.
Se il meccanismo di editing dell'RNA che opera nei tripanosomi si dovesse rivelare una caratteristica esclusiva dei parassiti e non fosse presente nelle cellule umane, allora questo processo potrebbe essere un eccellente bersaglio per la chemioterapia. Un farmaco che potesse colpire selettivamente gli enzimi necessari per l'editing potrebbe in teoria uccidere i parassiti senza danneggiare le cellule umane ospiti. La speranza è che l'applicazione pratica delle ricerche su questo bizzarro fenomeno genetico possa un giorno mostrarsi utile nel trattamento delle numerose malattie umane e animali causate dai tripanosomi nei paesi del Terzo Mondo.
Altri tipi di editing dell'RNA
Un tipo ancora più complesso di editing per inserimento di basi è stato osservato nei trascritti mitocondriali del fungo mucillaginoso Physarum (Gott et al., 1993; Mahendran et al., 1994). In questo organismo tutti i trascritti del genoma mitocondriale, rRNA, tRNA e mRNA, vengono modificati: principalmente per inserimento di residui di C, ma anche di U, G e A, di dinucleotidi o per sostituzioni di C con U. Ancora non si conosce il meccanismo, o i meccanismi, di questi eventi di editing.
Un altro tipo di editing è stato scoperto in un gene nucleare di mammifero, che codifica l'apolipoproteina B (apoB), e nei mitocondri e nei cloroplasti delle piante, in cui C viene sostituita da U in siti ben precisi (Riesel et al., 1989; Covello e Gray, 1989). Questo è stato chiamato editing per sostituzione, per distinguerlo dall'editing per inserimento e delezione osservato nei tripanosomi e in Physarum. l cambiamenti da C a U sembrano coinvolgere una deamminazione dei residui di C presenti ma non è chiaro come questo evento venga limitato a un certo numero di siti specifici nel genoma, tranne che nel caso del singolo evento di editing della apoB, in cui specifiche proteine riconoscono brevi sequenze di DNA adiacenti al sito di editing (Backus e Smith, 1992; Backus e Smith, 1994). Un editing per sostituzione dei tRNA è stato osservato anche nei mitocondri dell'eucariote inferiore Acanthamoeba castellani e nei mitocondri delle chiocciole di terra: singoli nucleotidi appaiati in modo improprio nel braccio accettore del tRNA vengono sostituiti da nucleotidi in grado di appaiarsi correttamente (Lonergan e Gray, 1993). Nei mitocondri dei marsupiali e dei ratti è stato osservato un unico tipo di editing per sostituzione, in cui si verifica un singolo cambiamento da C a U all'interno dell'anticodone di un tRNA o in prossimità di esso (Morl et al., 1995).
Un' altra classe di editing per sostituzione è stata rilevata in numerosi mRNA umani per i recettori del glutammato; essa comporta la deamminazione dei residui di A, dando origine alla inosina (1), riconosciuta dall' apparato di traduzione come G. Questo tipo di editing da A a l dei recettori del glutammato nei mammiferi fu scoperto in un modo insolito. Inizialmente venne descritta un'attività enzimatica capace di svolgere un RNA a doppio filamento cambiando le A in l (Bass e Weintraub, 1988); poi venne purificata una adenosinadeamminasi per l'RNA a doppio filamento (dsRAD o DRADA) che catalizza la deamminazione di A in l, di cui venne poi clonato il gene (Kim et al., 1994; Wang et al., 1995). lnfine è stata attribuita a questa attività enzimatica una funzione biologica con l'osservazione che gli mRNA per i recettori del glutammato venivano modificati per sostituzione di A con G in siti specifici, e che tale sostituzione richiede la formazione di una regione di RNA a doppio filamento, la quale si ottiene con il ripiegamento di una sequenza complementare localizzata in un introne a valle (Higuchi et al., 1993; Maas et al., 1996). Dal momento che l'apparato di traduzione tratta le l come se fossero delle G, si sospettò immediatamente che la dsRAD fosse la causa principale dell'evento. Sembra tuttavia che un'altra adeninadeamminasi correlata, la RED l, sia responsabile di questo specifico evento di editing (Melcher et al., 1996). Comunque recentemente la dsRAD è stata implicata nell' editing per sostituzione di A con G che avviene nell'RNA antisenso del virus dell'epatite B delta. L'editing per conversione di A in l è probabilmente molto diffuso, vista l'ubiquità di attività del tipo dsRAD negli organismi superiori.
Un'altra modificazione dell'RNA, anch'essa chiamata editing, implica l'aggiunta di G nell'mRNA di alcuni virus (Vidaletal., 1990; Pelet et al., 1991; Curranetal., 1991). In vari paramyxovirus un unico gene P dà origine a due diversi mRNA: uno è una copia esatta del DNA corrispondente, mentre l'altro contiene una o due G in più inserite entro un tratto di cinque o sei G. Questo produce uno spostamento del modulo di lettura originaria che permette ai ribosomi di accedere a un secondo modulo di lettura più a valle, dando origine a una proteina P alternativa con una sequenza Cterminale diversa. L'aggiunta di G nell'mRNA virale è probabilmente dovuta a una specie di stuttering (tartagliamento) dell'RNA-polimerasi durante la trascrizione.
Conclusioni
La modificazione delle sequenze di RNA dopo la trascrizione è un fenomeno ampiamente diffuso nelle cellule eucariotiche e coinvolge molti tipi diversi di meccanismi. Il tipo di editing per inserimento e delezione di U osservato nei mitocondri dei tripanosomi sembra essere caratteristico dei protozoi cinetoplastidi. L'editing per inserimento di C osservato nei mitocondri di Physarum ha qualche somiglianza con l'editing dei tripanosomi, ma per giungere a una conclusione è necessario ottenere ulteriori informazioni. L'editing per sostituzione di C con U osservato nei mitocondri delle piante e nell'mRNA per la apoB, come anche quello per sostituzione di A con I dei recettori umani del glutammato, coinvolgono entrambi eventi di deamminazione, ma le caratteristiche strutturali, i meccanismi catalitici e i siti di legame alI 'RNA sono diversi per le due reazioni. Una caratteristica in comune tra l' editing per sostituzione di A con I e quello per inserimento e delezione delle U, è la necessità di RNA a doppio filamento come elemento di riconoscimento.
L'importanza biologica dei vari tipi di editing è evidenziata dal fatto che queste modificazioni sono spesso regolate e hanno conseguenze fenotipiche significative. Di certo in futuro verranno scoperti ulteriori esempi di editing, in special modo non appena saranno disponibili le sequenze genomi che complete di vari organismi.
Bibliografia citata
ABRAHAM, J., FEAGIN, J., STUART, K. (1988) Characterization of cytochrome C oxidase III transcripts that are edited only in the 3' region. CelI, 55, 267-272.
AVILA, H., SIMPSON, L. (1995) Organization and complexity of minicircle-encoded guide RNAs from Trypanosoma cruzi. RNA, l, 939-947.
BACKUS, J.W., SMITH, H.C. (1992) Three distinct RNA sequence elements are required for efficient apolipoprotein B (apoB) RNA editing in vitro. Nucleic Acids Res., 20, 6007-6014.
BACKUS, J.W., SMITH, H.C. (1994) Specific 3' sequences flanking a minimal apolipoprotein B (apoB) mRNA editing 'cassette' are criticaI for efficient editing in vitro. Biochim. Biophys. Acta, 1217, 65-73.
BAKALARA, N., SIMPSON, A.M., SIMPSON, L. (1989) The Leishmania kinetoplast-mitochondrion contains terminaI uridylyltransferase and RNA ligase activities. J. Biol. Chem., 264, 18679-18686.
BASS, B., WEINTRAUB, H. (1988) An unwinding activity that covalently modifies its double-stranded RNA substrate. CelI, 55, 1089-1098.
BENNE, R., VAN DEN BURG, J., BRAKENHOFF, J., SLOOF, P., VAN BOOM, J., TROMP, M. (1986) Major transcript of the frameshifted coxII gene from trypanosome mitochondria contains four nucleotides that are not encoded in the DNA. CelI, 46, 819-826.
BHAT, G.J., KOSLOWSKY, D.J., FEAGIN, J.E., SMILEY, B.L., STUART, K. (1990) An extensively edited mitochondrial transcript in kinetoplastids encodes a protein homologous to ATPase subunit 6. CelI, 61, 885-894.
BLUM, B., BAKALARA, N., SIMPSON, L. (1990) A model for RNA editing in kinetoplastid mitochondria: 'Guide' RNA molecules transcribed from maxicircle DNA provide the edited information. CelI, 60, 189-198.
BLUM, B., SIMPSON, L. (1990) Guide RNAs in kinetoplastid mitochondria have a nonencoded 3' oligo-(D) tail involved in recognition of the pre-edited region. CelI, 62, 391-397.
BLUM, B., STURM, N.R., SIMPSON, A.M., SIMPSON, L. (1991) Cmmeric gRNA-mRNA mo1ecu1es with oligo(U) tails covalently linked at sites of RNA editing suggest that U addition occurs by transesterification. CelI, 65, 543-550.
BRINGAUD, F., PERIS, M., ZEN, K.H., SIMPSON, L. (1995) Characterization of two nuclear-encoded protein components of mitochondrial ribonucleoprotein complexes from Leishmania tarentolae. Mol. Biochem. Parasitol., 71, 65-79.
BYRNE, E., BRINGAUD, F., SIMPSON, L. (1995) RNA-protein interactions in the ribonucleoprotein T -complexes in a mitochondrial extract from Leishmania tarentolae. Mol. Biochem. Parasitol., 72, 65-76.
BYRNE, E.M., CONNELL, G.J., SIMPSON, L. (1996) Guide RNA-directed uridine insertion editing in vitro. EMBO J., 15, 6758-6765.
CECH, T.R. (1991) RNA editing: world's smallest introns. CelI, 64, 667-669.
CHIU, N., CHIU, A., SUYAMA, Y. (1975) Native and imported transfer RNA in mitochondria. J. Mol. Biol., 99, 37-50.
CONNELL, G.J., BYRNE, E.M., SIMPSON, L. (1997) Guide RNA-independent and guide RNA-dependent uridine insertion into cytochrome b mRNA in a mitochondrial lysate from Leishmania tarentolae - Rol1e of RNA secondary structure. J. Biol. Chem., 272, 4212-4218.
CORELL, R.A., FEAGIN, J.E., RiLEY, G.R., STRICKLAND, T., GUDERIAN, J.A., MYLER, P.J., STUART, K. (1993) Trypanosoma brucei minicircles encode multiple guide RNAs wmch can direct editing of extensively overlapping sequences. Nucleic Acids Res., 21, 4313-4320.
COVELLO, P.S., GRAY, M.W. (1989) RNA editing in plant mitochondria. Nature, 341, 662-666.
CURRAN, J., BOECK, R., KOLAKOFSKY, D. (1991) The Sendai virus P gene expresses both an essential protein and an inhibitor of RNA synthesis by shuffling modules via mRNA editing. EMBO J., 10, 3079-3085.
DECKER, C.J., SOLLNER-WEBB, B. (1990) RNA editing involves indiscriminate U changes throughout precisely defined editing domains. CelI, 61, 1001-1011.
DE LA CRUZ, V., NECKELMANN, N., SIMPSON, L. (1984) Sequences of six structural genes and several open reading frames in the kinetoplast maxicircle DNA of Leishmania tarentolae. J. Biol. Chem., 259, 15136-15147.
FEAGIN, J.E., ABRAHAM, J., STUART, K. (1988b) Extensive editing of the cytochrome c oxidase III transcript in Trypanosoma brucei. CelI, 53, 413-422.
FEAGIN, J.E., SHAW, J.M., SIMPSON, L., STUART, K. (1988a) Creation of AUG initiation codons by addition of uridines within cytochrome b transcrits ofkinetoplastids. PNAS., 85, 539-543.
FERNANDES, A.P., NELSON, K., BEVERLEY, S.M. (1993) Evolution of nuclear ribosomal RNAs in kinetoplastid protozoa: Perspectives on the age and origins of parasitism. Proc. Natl. Acad. Sci. USA, 90, 11608-11612.
FRECH, G.C., SIMPSON, L. (1996) Uridine insertion into pre-edited mRNA by a mitochondrial extract from Leishmania tarentolae: stereochemical evidence for the enzyme cascade model. Mol. CelI. Biol., 16, 4584-4589.
GOTT, J.M., VISOMIRSKI, L.M., HUNTER, J.L. (1993) Substitutional and insertional RNA editing of the cytochrome c oxidase subunit 1 mRNA of Physarum polycephalum. J. Biol. Chem., 268, 25483-25486.
HARRIS, M., DECKER, C., SOLLNER-WEBB, B., HAJDUK, S. (1992) Specific cleavage of pre-edited mRNAs in trypanosome mitochondrial extracts. Mol. CelI. Biol., 12, 2591-2598.
HAUSER, R., SCHNEIDER, A. (1995) tRNAs are imported into mitochondria of Trypanosoma brucei independently of their genomic context and genetic origino EMBO J., 14, 4212-4220.
HENSGENS, A., BRAKENHOFF, J., DE VRIES, B., SLOOF, P., TROMP, M., VAN BOOM, J., BENNE, R. (1984) The sequence of the gene for cytochrome c oxidase subunit I, a frameshift containing gene for cytochrome c oxidase subunit II and seven unassigned reading frames in Trypanosoma brucei mitochondrial maxi-circle DNA. Nucleic Acids Res., 12, 7327-7344.
HIESEL, R., WISSINGER, B., SCHUSTER, W., BRENNICKE, A. (1989) RNA editing in plant mitochondria. Science, 246, 1632-1634.
HIGUCHI, M., SINGLE, F.N., KAHLER, M., SOMMER, B., SPRENGEL, R., SEEBURG, P.H. (1993) RNA editing of AMPA receptor subunit GluR-B: a base-paired intron-exon structure determines position and efficiency. CelI, 75, 1361-1370.
KIM, U., WANG, Y., SANFORD, T., ZENG, Y., NISHIKURA, K. (1994) Molecular cloning of cDNA for double-stranded RNA adenosine deaminase, a candidate enzyme for nuclear RNA editing. Proc. Natl. Acad. Sci. USA, 91, 11457-11461.
KOSLOWSKY, D.J., BHAT, G.J., PERROLLAZ, A.L., FEAGIN, J.E., STUART, K. (1990) The MURF3 gene of T. brucei contains multiple domains of extensive editing and is homologous to a subunit of NADH dehydrogenase. CelI, 62, 901-911.
LANDWEBER, L.F., GILBERT, W. (1994) Phylogenetic analysis of RNA editing: a primitive genetic phenomenon. Proc. Natl. Acad. Sci. USA, 91, 918-921.
LIMA, B.D., SIMPSON, L. (1996) Sequence-dependent in vivo importation of tRNAs into the mitochondrion of Leishmania tarentolae. RNA, 2, 429-440.
LONERGAN, K.M., GRAY, M.W. (1993) Editing of transfer RNAs in Acanthamoeba castelIani mitochondria. Science, 259, 812-816.
LUKES, J., ARTS, G.J., VAN DEN BURG, J., DE HAAN, A., OPPERDOES, F., SLOOF, P., BENNE, R. (1994) Novel pattem of editing regions in mitochondrial transcripts of the cryptobiid Trypanoplasma borreli. EMBO J., 13, 5086-5098.
MAAS, S., MELCHER, T., HERB, A., SEEBURG, P.H., KELLER, W., KRAUSE, S., HIGUCHI, M., O'CONNELL, M.A. (1996) Structural requirements for RNA editing in glutamate receptor premRNAs by recombinant double-stranded RNA adenosine deaminase. J. Biol. Chem., 271, 12221-12226.
MAHENDRAN, R., SPOTTSWOOD, M.S., GHATE, A., LING, M.-I., JENG, K., MILLER, D.L. (1994) Editing of the mitochondrial small subunit rRNA in Physarum polycephalum. EMBO J., 13, 232-240.
MARINI, J., LEVENE, S., CROTHERS, l, ENGLUND, P. (1982) A novel helical structure in kinetoplast DNA. Proc. Natl. Acad. Sci. USA, 79, 7664-7668.
MASLOV, D.A., AVILA, H.A., LAKE, J.A., SIMPSON, L. (1994) Evolution of RNA editing in kinetoplastid protozoa. Nature, 365, 345-348.
MASLOV, D.A., SIMPSON, L. (1992) The polarity of editing within a multiple gRNA-mediated domain is due to formation of anchors for upstream gRNAs by downstream editing. CelI, 70, 459-467.
MASLOV, D.A., SIMPSON, L. (1994) RNA editing and mitochondrial genomic organization in the cryptobiid kinetoplastid protozoan, Trypanoplasma borreli. Mol. CelI. Biol., 14, 8174-8182.
MASLOV, D.A., STURM, N.R., NINER, B.M., GRUSZYNSKI, E.S., PERIS, M., SIMPSON, L. (1992) An intergenic G-rich region in Leishmania tarentolae kinetoplast maxicircle DNA is a pan-edited cryptogene encoding ribosomal protein S12. Mol. CelI. Biol., 12, 56-67.
MELCHER, T., MAAS, S., HERB, A., SPRENGEL, R., SEEBURG, P.H., HIGUCHI, M. (1996) A mammalian RNA editing enzyme. Nature, 379, 460-463.
MORL, M., DORNER, M., PAABO, S. (1995) C to U editing and modifications during the maturation of the mitochondrial tRNA(Asp) in marsupials. Nucleic Acids Res., 23, 3380-3384.
PELET, T., CURRAN, J., KOLAKOFSKY, D. (1991) The P gene of bovine parainfluenza virus 3 expresses alI three reading frames from a sing1e mRNA editing site. EMBO J., 10, 443-448.
POLLARD, V.W., ROHRER, S.P., MICHELOTTI, E.F., HANCOCK, K., HAJDUK, S.L. (1990) Organization of minicircle genes for guide RNAs in Trypanosoma brucei. CelI, 63, 783-790.
POLSON, AG., BASS, B.L., CASEY, J.L. (1996) RNA editing of hepatitis delta virus antigenome by dsRNA adenosine deaminase. Nature, 380, 454-456.
READ, L.K., MYLER, P.l, STUART, K. (1992) Extensive editing of both processed and preprocessed maxicircle CR6 transcripts in Trypanosoma brucei. J. Biol. Chem., 267, 1123-1128.
RYAN, K., SHAPIRO, T., RAUCH, C., ENGLUND, P. (1988) Replication of kinetop1ast DNA in trypanosomes. Ann. Rev. Microbiol., 42, 339-358.
SEIWERT, S.D., HEIDMANN, S., STUART, K. (1996) Direct visualization of uridylate deletion in vitro suggests a mechanism for kinetoplastid RNA editing. CelI, 84, 1-20.
SHAPIRO, T.A, ENGLUND, P.T. (1995) The structure and replication of kinetoplast DNA. Annu. Rev. Microbiol., 49, 117-143.
SHAW, J., FEAGIN, J.E., STUART, K., SIMPSON, L. (1988) Editing of mitochondrial mRNAs by uridine addition and deletion generates conserved amino acid sequences and AUG initiation codons. CelI, 53, 401-411.
SIMPSON, A.M., BAKALARA, N., SIMPSON, L. (1992) A ribonuclease activity is activated by heparin or by digestion with proteinase K in mitochondrial extracts of Leishmania tarentolae. J. Biol. Chem., 267, 6782-6788.
SIMPSON, A.M., SUYAMA, Y., DEWES, H., CAMPBELL, D., SIMPSON, L. (1989) Kinetoplastid mitochondrial contain functional tRNAs which are encoded in nuclear DNA and also small minicircle and maxicircle transcripts of unknown function. Nucleic Acids Res., 17, 5427-5445.
SIMPSON, L., DA SILVA, A.M. (1971) Isolation and characterization of kinetoplast DNA from Leishmania tarentolae. J. Mol. Biol., 56, 443-473.
SIMPSON, L., MASLOV, D.A. (1994) RNA editing and the evolution of parasites. Science, 264, 1870-1871.
SIMPSON, L., NECKELMANN, N., DE LA CRUZ, V., SIMPSON, A., FEAGIN, J., JASMER, D., STUART, K. (1987) Comparison ofthe maxicircle (mitochondrial) genomes of Leishmania tarentolae and Trypanosoma brucei at the level of nucleotide sequence. J. Biol. Chem., 262, 6182-6196.
SIMPSON, L., SHAW, J. (1989) RNA editing and the mitochondrial cryptogenes of kinetoplastid protozoa. CelI, 57, 355-366.
SOUZA, A.E., MYLER, P.I., STUART, K. (1992) Maxicircle CR1 transcripts of Trypanosoma brucei are edited, developmentally regulated, and encode a putative iron-sulfur protein homologous to an NADH dehyrogenase subunit. Mol. CelI. Biol., 12, 2100-2107.
SOUZA, A.E., SHU, H.-H., READ, L.K., MYLER, P.I., STUART, K.D. (1993) Extensive editing of CR2 maxicircle transcripts of Trypanosoma brucei predicts a protein with homology to a subunit of NADH dehydrogenase. Mol. CelI. Biol., 13, 6832-6840.
STURM, N.R., MASLOV, D.A., BLUM, B., SIMPSON, L. (1992) Generation of unexpected editing patterns in Leishmania tarentolae mitochondrial mRNAs: misediting produced by misguiding. CelI, 70, 469-476.
STURM, N.R., SIMPSON, L. (1990a) Partially edited mRNAs for cytochrome b and subunit 111 of cytochrome oxidase from Leishmania tarentolae mitochondria: RNA editing intermediates. CelI, 61, 871-878.
STURM, N.R., SIMPSON, L. (1990b) Kinetoplast DNA minicircles encode guide RNAs for editing of cytochrome oxidase subunit III mRNA. CelI, 61, 879-884.
TARASSOV, I., ENTELIS, N., MARTIN, R.P. (1995) Mitochondrial import of a cytoplasmatic lysine-tRNA in yeast is mediated by cooperation of cytoplasmatic and mitochondrial LysyltRNA synthetases. EMBO J., 14, 3461-3471.
THIEMANN, O.H., MASLOV, D.A, SIMPSON, L. (1994) Disruption of RNA editing in Leishmania tarentolae by the loss of minicircle-encoded guide RNA genes. EMBO J., 13, 5689-5700.
VIDAL, S., CURRAN, J., KOLAKOFSKY, D. (1990) A stuttering model for paramyxovirus P mRNA editing. EMBO J., 9, 2017-2022.
WANG, Y., ZENG, Y., MURRAY, lM., NISHIKURA, K. (1995) Genomic organization and chromosomal location of the human dsRNA adenosine deaminase gene: the enzyme for glutamateactivated ion chaunel RNA editing. J. Mol. Biol., 254, 184-195.
YASUHIRA, S., SIMPSON, L. (1997) Phylogenetic affinity of mitochondria of Euglena gracilis and kinetoplastids using cytochrome oxidase I and hsp60. J. Mol. Evol., 44, 341-347.
Bibliografia generale
ARAYA, A., BÉGU, D., LITVAK, S. (1994) RNA editing in plants. Physiol. Plant., 91, 543-550.
BASS, B.L. (1993) RNA editing: new uses for old players in the RNA world. In The RNA world, a c. di Gesteland R.F., Atkins J.F., Plainview, Cold Spring Harbor, Cold Spring Harbor Laboratory Press, pp. 383-418.
BENNE, R. (1994) RNA editing in trypanosomes. Eur. J. Biochem., 221, 9-23.
GRAY, M.W., COVELLO, P.S. (1993) RNA editing in plant mitochondria and chloroplasts. FASEB J., 7, 64-71.
SIMPSON, L. (1972) The kinetoplast of the hemoflagellates. Int. Rev. Cytol., 32, 139-207.
SIMPSON, L. (1986) Kinetoplast DNA in trypanosomid flagellates. Int. Rev. Cytol., 99, 119-179.
SIMPSON, L. (1987) The mitochondrial genome of kinetoplastid protozoa: genomic organization, transcription, replication, and evolution. Ann. Rev. Microbiol., 41, 363-382.
SIMPSON, L., EMESON, R.B. (1996) RNA editing. Annu. Rev. Neurosei., 19, 27-52.
SIMPSON, L., MASLOV, D.A, BLUM, B. (1993) RNA editing in Leishmania mitochondria. In RNA editing - the alteration of protein coding sequences of RNA, a c. di Beune R., New York, Ellis Horwood, pp. 53-85.
SIMPSON, L., THIEMANN, O. H. (1995) Sense from nonsense: RNA editing in mitochondria of kinetoplastid protozoa and slime molds. CelI, 81, 837-840.
STUART, K. (1993) RNA editing in mitochondria of African trypanosomes. In RNA editing - the alteration of protein coding sequences of RNA, a c. di Beune R., New York, Ellis Horwood, pp. 25-52.