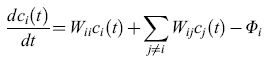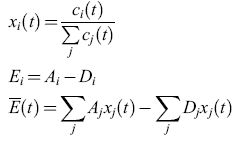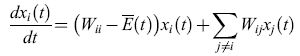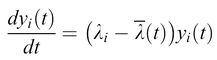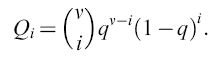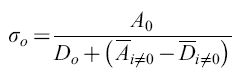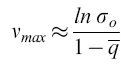Evoluzione in provetta
Evoluzione in provetta
L'evoluzione molecolare può essere descritta da un sistema di equazioni differenziali accoppiate che dimostrano come la selezione sia una proprietà intrinseca di una popolazione di molecole autocatalitiche o in grado di autoreplicarsi. II meccanismo evolutivo rappresentato da questo modello può essere esaminato con esperimenti mirati; inoltre, i principi dell'evoluzione possono integrarsi con le metodiche della biologia molecolare per lo sviluppo di nuove tecnologie. Così mediante un'evoluzione molecolare applicata, quelle molecole biologiche naturali che non si adattano in maniera ottimale a particolari fini di tipo tecnologico o medico, possono essere migliorate venendo sottoposte a cicli ripetuti di mutazione, selezione e amplificazione. L'applicazione di questa procedura a popolazioni molecolari può sostituire il metodo comunemente usato della progettazione razionale. Dalle analogie tra questa nuova tecnologia e l'evoluzione naturale scaturiscono sorprendenti soluzioni a vecchie problematiche.
Evoluzione significa ottimizzazione delle funzioni
La vita, come processo di autoperpetuazione, sfida continuamente la nostra comprensione poiché persino l'organismo unicellulare più primitivo mantiene un apparato intrinsecamente complesso ed efficiente, orchestrato magnificamente da proteine e da altre biomolecole. Charles DaIwin è stato il primo a riconoscere chiaramente che l'origine delle specie scaturiva da un adattamento ottimale all'ambiente. Egli comprese che l'adattamento a determinate condizioni è il risultato di un processo evolutivo realizzato per mezzo di una riproduzione soggetta a mutazioni e di un processo di selezione. La selezione, come concepita da Darwin, agisce sugli organismi viventi. In questo saggio ci chiediamo se i meccanismi della evoluzione naturale si applichino soltanto agli organismi viventi, o se anche alcune specie molecolari si possano adattare in maniera ottimale a funzioni specifiche. Poiché ogni processo vitale e ogni biomolecola, che svolge una particolare funzione nel processo, sono istruiti dall'informazione, ci chiediamo come possa essere generata l'informazione per la codificazione di funzioni ottimali.
La comprensione dei meccanismi complessi alla base dei sistemi viventi richiede una precisa descrizione matematica, come pure esperimenti che permettano di verificare la teoria e di derivare i parametri in essa coinvolti. Presentiamo qui alcuni modelli matematici atti a descrivere l'evoluzione molecolare e proviamo a illustrarli con esperimenti basati sull 'uso di varie strumentazioni in grado di simulare in laboratorio, in condizioni controllate, i processi evolutivi. Di conseguenza, vedremo che tali macchine che 'mimano' l'evoluzione possono essere usate per 'costruire' macromolecole funzionali (acidi nucleici o proteine) per applicazioni mediche o tecnologiche.
L'informazione dirige l'evoluzione molecolare
Gli organismi non sono soltanto complessi sistemi chimici, ma entità capaci di autoorganizzazione funzionale, che significa coordinamento di tutti quei meccanismi delle reazioni biologiche essenziali per l'esistenza dell'organismo. L'autoorganizzazione funzionale è guidata dall'informazione genetica che si genera e si preserva mediante circuiti di retro azione (feedback loops). L'informazione è codificata nelle molecole di RNA e DNA che provvedono anzitutto all'espressione dell'intero apparato di replicazione; successivamente, queste molecole vengono amplificate da tale complesso cooperativo di biomolecole. Ciò significa che l'informazione è legata alla materia e, poiché la materia si degrada continuamente, l'informazione verrebbe persa, se non fosse conservata mediante una riproduzione continua indefinita. Tuttavia la riproduzione non è un processo del tutto esatto: gli errori che avvengono durante il processo replicativo producono delle variazioni, e la nuova informazione è poi valutata in base alla funzione codificata. La riproduzione è così la causa ultima della selezione naturale. A sua volta, l' ottimizzazione funzionale è il risultato di cicli ripetuti di mutazione-selezione-amplificazione, il che sarebbe inconcepibile senza una autoriproduzione o una riproduzione per complementarità.
Il processo evolutivo a livello molecolare può essere descritto con un modello matematico. Secondo tale modello, un sistema darwiniano obbedisce a quattro condizioni (cominciando dal livello di complessità più alto):
1) La vita si origina per evoluzione molecolare.
2) L'evoluzione molecolare è una conseguenza della variazione e della selezione naturale in condizioni di non-equilibrio.
3) La selezione naturale è un effetto dell'autoriproduzione. In natura essa è associata agli acidi nucleici. La variazione si origina per autoriproduzione inesatta.
4) L'autoriproduzione è basata sulla complementarità strutturale delle molecole codificanti l'informazione. L'autoriproduzione, la mutabilità e il metabolismo sono inerenti ai sistemi darwiniani e sono rappresentabili mediante un sistema di equazioni differenziali accoppiate. Ne risulta una descrizione del comportamento dinamico di oggetti che portano l'informazione. Le molecole come l'RNA e il DNA e similmente sistemi più complessi contenenti proteine, come i virus e gli organismi cellulari, sono vettori replicativi di informazione che obbediscono alle leggi dell' evoluzione molecolare.
Le due classi più importanti di macromolecole biologiche, polipeptidi e polinucleotidi, presentano un comune principio di assemblaggio; sono prodotti di polimerizzazione lineare che hanno uno 'scheletro' regolare e ripetitivo, su cui sono legati vari gruppi funzionali. La struttura di questi polimeri permette una variabilità che va oltre la nostra immaginazione. Questa enorme variabilità è facilmente espressa dall'equazione [l], che descrive il numero N di differenti eteropolimeri di lunghezza v che possono essere costruiti a partire da un numero Il di monomeri differenti:
N=μv [l]
dove μ è 4 per gli acidi nucleici e 20 per le proteine naturali. Per esempio, anche per un polinucleotide corto, con v = 20, sono possibili circa 10¹² sequenze differenti. Similmente, per un polipeptide della stessa lunghezza, vi sono circa 10²⁶ sequenze alternative. Comunque, la maggior parte delle proteine funzionali presenti negli organismi viventi ha valori tipici di v (lunghezza della catena) più grandi di 100, e a volte anche più grandi di 1000; questo corrisponde rispettivamente a circa 10¹³⁰, e a oltre 10¹³⁰⁰ sequenze alternative. Se confrontiamo il numero delle sequenze possibili con la massa totale del nostro Universo, che equivale a circa 10⁸⁰ atomi di idrogeno, tale numero appare 'iperastronomico' anche per polimeri di lunghezza moderata.
L'autoreplicazione causa la selezione in complessi molecolari
Gli acidi nucleici sono i soli biopolimeri naturali conosciuti che possono servire come stampo per la propria replicazione. La capacità di rendere possibili le interazioni tra i loro costituenti complementari è una proprietà intrinseca della struttura molecolare degli acidi nucleici. L' appaiamento delle basi permette la sintesi di una copia complementare unica di ciascun polinucleotide. La replicazione di questo filamento complementare (filamento -) produce poi una copia del filamento originale (filamento +). Così il complesso più-meno ha un comportamento molto simile a un semplice sistema autocatalizzatore; quindi i polinucleotidi possono essere visti come autocatalizzatori intrinseci. Le cinetiche dell'autoreplicazione, piuttosto che della replicazione per complementarità, possono essere espresse da equazioni differenziali, il cui caso più semplice è rappresentato dalla equazione seguente:
formula [2]
con i,j= 1, .... , N, Wii=AiQi-Di'≥0, e dove
i: indice variabile rappresentante tutte le singole sequenze in una popolazione;
Wii: costante di velocità per la corretta replicazione della specie i;
Ai: costante di velocità per la formazione della specie i;
Qi: fattore di qualità, che rappresenta la probabilità media per una copia fedele della sequenza i. (1- Qi) è la corrispondente probabilità di una riproduzione non corretta;
Di: costante di velocità per la decomposizione della specie i;
Wij: coefficiente di mutazione, indicante la probabilità di formazione della sequenza i dovuta a una erronea riproduzione della sequenza j;
Ci(t): concentrazione della sequenza i al tempo t;
Φi: flusso che permette il controllo di α per mezzo di un input e di un output in un sistema aperto.
Un sistema di equazioni differenziali del tipo [2] descrive la crescita contemporanea in competizione di tutti gli individui presenti in una popolazione. Questo sistema di equazioni differenziali presenta le tipiche proprietà che sono state defrnite per un sistema darwiniano:
- l' autoriproduzione per mezzo di crescita autocatalitica è descritta dal primo termine;
- la mutabilità, cioè l'autoriproduzione errata, compare con
Qi< l e Wj ≥O;
- il metabolismo è collegato alla formazione irreversibile e alla degradazione delle molecole, valori espressi qui da
Aici(t), Diclt) e Φi. La competizione avviene se tutti gli individui i presenti in una popolazione sono presi in esame. Se si definiscono il numero di popolazione relativa, Xi(t), la produttività in eccesso, Ei, della specie i, e la media della produttività in eccesso, E(t), mediante le seguenti relazioni:
allora l'equazione [2] può essere scritta come
formula [3]
del tutto indipendente da qualsiasi valore di Φ (compreso Φ = O). L'equazione [3] può essere risolta in maniera esatta per mezzo di una trasformazione lineare (Thompson e McBride, 1974; Jones et al., 1976; Eigen et al., 1989) e porta a:
formula [4]
dove Λi sono gli auto valori e Yi (cioè le combinazioni lineari associate di tutte le Xi) rappresentano modi normali delle popolazioni relative, le quali costituiscono i contributi pesati dei singoli mutanti. Il valore medio degli autovalori, Λ(t), corrisponde alla media della produttività in eccesso E(t). In accordo con l'equazione [4] è ovvio che: l) tutte le distribuzioni di mutanti con Λi minore di Λ( t) muoiono, mentre: 2) tutte le distribuzioni di mutanti con Λi più grande di Λ(t) sono amplificate, aumentando quindi Λ(t) fino a raggiungere il suo valore Amax. Aumentando Λi, sempre più sequenze sono rimosse dalla competizione. Alla fine, rimane soltanto la distribuzione mutante che si replica più efficientemente, cioè quella che possiede l'autovalore Λmax maggiore. Questa categoria di comportamenti la chiamiamo selezione. In conclusione, la selezione è una proprietà intrinseca della riproduzione autocatalitica di qualsiasi popolazione di individui descritta dal modello precedente! Ciò significa che la selezione non si applica a una singola specie molecolare che si replica. L'introduzione di mutazioni descritta da un sistema di equazioni differenziali di tipo [2] produce situazioni in cui sono presenti gruppi di mutanti piuttosto che singoli individui. In analogia al concetto di specie che Darwin ha creduto essere il riferimento della sopravvivenza del più adatto nel processo evolutivo, la descritta distribuzione di mutanti con la maggiore efficienza replicativa è un modo normale che si comporta come una specie individuale, e che viene perciò chiamato quasispecie (Eigen e Schuster, 1977; Eigen et al., 1989). La quasispecie sostituisce così il precedente ceppo selvatico.
Il progresso evolutivo è basato su una copiatura errata
L'appaiamento delle basi complementari non porta a una precisione assoluta del processo di replicazione. In accordo con ciò, la procedura sopra descritta tiene conto degli errori che avvengono nel processo replicativo durante la riproduzione, includendo un termine di mutazione nell' equazione [3]. Per esempio, il virus HlV dell'AlDS incorpora (in media) un nucleotide errato ogni 10.000 basi. Mutazioni come queste sono ovviamente prodotte da eventi stocastici non riproducibili (cioè non deterministici). Questo è valido quando si considerano grandi genomi e popolazioni limitate, come per esempio popolazioni umane, dove il possibile numero di singole mutazioni (in un genoma pari a circa 3.10⁹ coppie di basi) sarebbe più grande del numero di esseri umani che siano mai esistiti sulla Terra. Invece, per la distribuzione di molecole o genomi virali che si replicano, gli errori di replicazione non sono necessariamente eventi statisticamente rari. In questi sistemi molti mutanti individuali ricorrono con una certa riproducibilità. La frequenza di produzione di un mutante mediante replicazione imprecisa alle posizioni i del genoma di lunghezza v è data dall' espressione binomiale
formula [5]
L'accuratezza con cui un dato nucleotide è incorporato in una certa posizione è espressa dalla fedeltà q, che è la media geometrica dei tassi di errore individuali (e posizione-dipendenti). Perciò, (1- q) è indice di frequenza di errore media. Una frequenza di errore di 0,1 equivale a una falsa incorporazione (in media) ogni 10 posizioni.
Dall'altro lato, lafrequenza di comparsa di un certo mutante è anche influenzata dal suo tasso di replicazione. Il principio degli 'estremali' (cioè risultanti in una funzione critica) descritto con l'equazione [4] prende in considerazione questo effetto valutando la presenza di mutanti in una distribuzione secondo la loro fitness (idoneità biologica), cioè la loro abilità a riprodursi efficientemente in date condizioni ambientali. Se la quasispecie contiene una copia master chiaramente dominante, e se trascuriamo la retromutazione, si può definire
formula [6]
che porta, con σo Qo ≥ 1 e Qo = qV, alla seguente espressione per la capacità informazionale limite:
formula [6
a ]
Qui, σo descrive la 'superiorità' o selettività della sequenza master che in questo caso domina la distribuzione di mutanti per la frequenza con cui compare (indice o). σo è una grandezza che correla la produttività della sequenza master, il suo tasso di degradazione e la sovraproduttività media di tutti i competitori i (si noti che Āi≠0 eD ̅i≠0 rappresentano medie fatte su tutti i mutanti presenti nella distribuzione, a eccezione della copia master). L'equazione [6a] è molto importante. Essa dice che una data frequenza di errore limita la lunghezza massima della sequenza della copia che mostra il massimo valore selettivo. In altre parole, definisce una soglia di errore. Una distribuzione di mutanti appena al di sotto di questa soglia ha la più grande variazione genetica possibile, pur rimanendo (meta)stabile. In realtà, queste distribuzioni non sono simmetriche. Esse includono molte specie individuali 'neutrali' o 'quasi neutrali', come pure più di una (o anche nessuna) specie dominante. Prendendo in considerazione la frequenza di retromutazione, la concentrazione relativa di ogni sequenza master può essere calcolata con esattezza. Per sequenze lunghe, i risultati non sono significativamente differenti dai valori approssimati ottenuti con l'equazione [6a]. Come conseguenza delle considerazioni sopra riportate, le sequenze di consenso sono defrnite come sequenze medie rappresentanti il 'centro di gravità' della distribuzione. l componenti individuali di tali sequenze di consenso non sono affatto le specie più adatte o più frequenti. Questo corrisponde a ciò che viene definito ceppo selvatico (wild type).
'Prove evolutive' di selezione di mutanti vantaggiosi (in condizioni ambientali che cambiano) avvengono alla periferia della distribuzione della quasispecie, nelle vicinanze di quei mutanti che ancora appaiono con frequenze sufficientemente grandi. Bisogna anche dire che la ricombinazione genetica contribuisce al comportamento della soglia di errore senza cambiame la natura (Wiehe et al., 1995; Boerlijst et al., 1996).
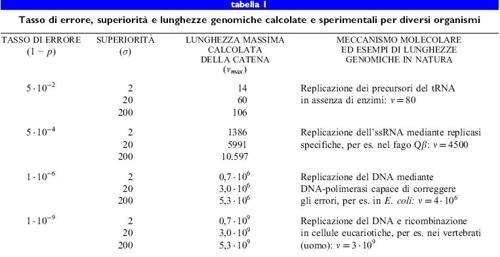
Come conseguenza, il superamento della soglia di errore porta a un accumulo di errori e quindi a una distribuzione casuale di sequenze, con completa perdita di informazione. Questo processo sembra essere equivalente a una transizione di fase (Leuthiiusser, 1986); l'informazione 'evapora' in modo simile a un liquido al suo punto di ebollizione. Inoltre l'equazione [6a] defrnisce la massima lunghezza di sequenza che può essere riprodotta stabilmente sotto l'influenza della selezione. Da parte di diversi gruppi di ricerca è stato stabilito sperimentalmente che i virus si comportano come quasispecie e operano vicino alla loro soglia di errore (Domingo et al., 1978; Domingo et al., 1985). Per esempio, sulla base del tasso di errore, e secondo l'equazione [6a], il batteriofago Qf3 dovrebbe essere codificato al massimo da 4500 nucleotidi; in effetti, si è visto che la lunghezza del suo genoma è di 4220 nucleotidi.
La popolazione di una quasispecie si comporta come una nube errante
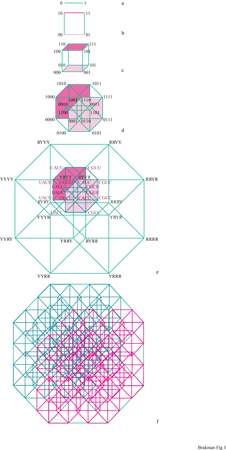
Per trattare l'enorme complessità delle sequenze di una quasispecie è necessario introdurre un metodo appropriato per rappresentare l'informazione. Un modello spaziale può riflettere correttamente le relazioni informazionali che intercorrono tra le sequenze (cioè, sequenze che differiscono soltanto per una posizione devono essere le più vicine nella rappresentazione). Lo spazio delle sequenze, inizialmente proposto da R.W. Hamming (Hamming, 1980) per un codice binario, e anche da 1. Rechenberg (Rechenberg, 1973), è stato poi esteso per l'alfabeto genetico a quattro lettere, portando a una costruzione 2v-dimensionale (Eigen et al., 1988). La costruzione di uno spazio delle sequenze è mostrata in figura (fig. 1). Ciascun punto nell'ipercubo fmale rappresenta una sequenza definita; le linee collegano quei mutanti che differiscono soltanto per una posizione. Un tratto caratteristico dello spazio delle sequenze è il suo alto potenziale di connessione, che permette a una sequenza A di raggiungere una sequenza B con un numero minimo di passaggi. Sebbene un acido nucleico lungo 10 basi abbia 4¹⁰ (circa 106) punti nel suo spazio delle sequenze, per attraversare l'intero spazio sono necessari 10 passaggi. Poiché ciascuna sequenza è rappresentata da un punto avente una posizione definita nello spazio, che riflette in maniera corretta le relazioni di parentela con tutte le altre sequenze, il processo di ottimizzazione evolutiva può essere descritto come un percorso nello spazio delle sequenze che non è necessariamente continuo ma piuttosto a punti discreti.
Per una sequenza di lunghezza normale si ha l'occupazione soltanto di una frazione molto piccola di tutti i punti nello spazio delle sequenze. L'equipartizione (cioè la distribuzione omogenea della popolazione nell'intero spazio delle sequenze) corrisponde a una replicazione con bassa fedeltà sopra la soglia di errore. Ciò ricorda un gas ideale che popola l'intero spazio con una densità molto bassa. La condensazione del 'gas' in una 'nube' localizzata può avvenire soltanto al di sotto della soglia di errore. Quindi la sequenza di consenso è identica al 'centro di gravità' della nube. Il passaggio dal gas alla nuvola mostra tutte le caratteristiche di una transizione di fase, come è stato confermato dalla teoria (Leuthiiusser, 1986). Il virus HlV, per esempio, è codificato da 9200 nucleotidi e perciò corrisponde a uno spazio delle sequenze comprendente 4⁹²⁰⁰ (circa 105500) punti, una complessità che non possiamo neppure immaginare. Come paragone, se dividessimo il nostro Universo, equivalente a una sfera con raggio dell'ordine di 10¹⁰ anni-luce, in celle della grandezza giusta per contenere un solo atomo di idrogeno (cioè con un volume di un Ångström cubico), vi sarebbero 'soltanto' 10¹⁰⁸ di tali celle. Da questo esempio si vede chiaramente che nelle condizioni ambientali attuali la selezione deve preferire alcuni mutanti nella quasispecie, mentre altri tipi di mutanti sono svantaggiati; quindi molti punti sono occupati molte volte, mentre altri restano vuoti. L'evoluzione di una quasispecie ricorda una nube di sequenze strettamente collegate che si muovono attraverso lo spazio delle sequenze.
Ciascuna sequenza mostra una certa fitness in date condizioni ambientali. Assegnando valori di fitness a ciascuna sequenza ore sente nello spazio delle sequenze si disegna un 'paesaggio' di fitrless che consiste di picchi (sequenze con una fitness alta), collegati da catene e separati da selle, vallate (sequenze con bassa fitness) e piani. La topografia di questo paesaggio di fitrless, espressa in termini di densità di popolazione, indirizza la nube vagante della quasispecie verso una fitrless massima.
L'evoluzione può essere riprodotta in provetta
Una volta esplorata la base teorica dell'evoluzione molecolare, la domanda che ci si pone è come questa teoria possa essere verificata sperimentalmente. Vi sono due quesiti fondamentali:
1) È possibile ottimizzare le funzioni biochimiche e le strutture applicando strategie evolutive in laboratorio?
2) Esistono applicazioni pratiche che siano basate sulla visualizzazione e sull'imitazione dell'evoluzione naturale? Il nostro interesse primario si focalizza quindi su dispositivi sperimentali che permettano di saggiare e imitare i meccanismi dell'evoluzione naturale. Un prerequisito necessario sta nel condurre i tre processi (mutazione, selezione e amplificazione) in condizioni definite con precisione e in maniera sequenziale. Secondo la teoria, individui molecolari e, similmente, virus e piccoli organismi come i batteri, dovrebbero funzionare come oggetti di una 'evoluzione in provetta'. Prima di esaminare più da vicino i vari esperimenti, dobbiamo distinguere due diverse strategie.
1) Selezione naturale e auto organizzata, dovuta alla crescita competitiva di una popolazione di individui.
2) Selezione artificiale e non autoorganizzata, senza competizione, nella quale mutanti con una qualsiasi proprietà desiderata possono essere favoriti da un'interferenza esterna.
La selezione naturale favorisce individui con sistemi riproduttivi più efficienti. La loro fitness è manifestata da un sostanziale vantaggio nella crescita. Mutanti con proprietà defmite emergono sotto l'influenza della pressione selettiva che è vantaggiosa alla crescita di questi mutanti. In una serie di esperimenti per dimostrare il potere di questo approccio, i replicatori molecolari e i batteriofagi sono stati esposti a varie costrizioni ambientali che hanno portato a specie con aumentata efficienza replicativa. A partire dal batteriofago Qβ, sono state studiate le infezioni virali sotto l'influenza di anticorpi che neutralizzano la proteina del rivestimento del fago (Lindemann, 1992). In un altro esperimento sono stati aggiunti piccoli RNA replicativi che potessero competere con il genoma virale per la replicazione da parte della polimerasi codificata dal fago (Schwienhorst, 1992). In entrambi gli esperimenti si sono evoluti dei mutanti fagici che, sfuggendo alla pressione selettiva, sono stati in grado di evitare l'attacco antivirale cui erano stati sottoposti. Similmente è stata investigata la fitness di replicatori molecolari. L'RNA di Qβ purificato è stato amplificato con la replicasi di Qβ (la polimerasi codificata dal fago) in un mezzo di reazione contenente un inibitore della replicazione (eparina) oppure un enzima che degrada l'RNA (Strunk, 1993). Come previsto, anche questi esperimenti hanno portato a specie che sfuggono alla pressione selettiva, e hanno permesso una iniziale comprensione dei meccanismi molecolari dell'evoluzione (per un maggiore approfondimento, v. oltre).
La selezione artificiale, al contrario della selezione naturale, è basata sulla conoscenza precisa della proprietà desiderata e perciò richiede un metodo per selezionare grandi popolazioni secondo le caratteristiche che sono state definite per la selezione. Una procedura di selezione tipica comincia con una estesa mutagenesi casuale di un clone che presenta somiglianze considerevoli alle caratteristiche desiderate. Il repertorio (library) di mutanti risultante viene poi selezionato mediante un saggio adatto a rivelare mutanti promettenti. Questo processo è reiterato sui mutanti nuovamente selezionati fino a quando non si ottengono uno o più individui ottimali. La caratteristica di questa procedura di selezione artificiale è la separazione tra crescita (o amplificazione) e selezione: in antitesi con il sistema naturale, dove il tasso di riproduzione ottimale gioca sempre un ruolo importante nella selezione, qui si può scegliere qualsiasi criterio di selezione. Uno dei primi esperimenti in cui è stata applicata questa strategia è quello ideato da C. Tuerk e L. Gold (1rvine et al., 1991). l loro tentativi di sviluppare il metodo SELEX (Systematic Evolution of Ligands by EXponential enrichment, evoluzione sistematica dei ligandi per arricchimento esponenziale) coinvolgevano la selezione da una library di sequenze casuali di acidi nucleici con varia attività di legame. Effettuando la selezione con un processo iterativo (utilizzando un saggio in grado di evidenziare l'affrnità di legame verso un dato ligando) seguito da amplificazione (per es., tramite PCR, Polymerase Chain Reaction, reazione a catena della polimerasi) sono stati arricchiti gli RNA con alta affinità per una varietà di ligandi. Modificando questa procedura alla ricerca di acidi nucleici con attività catalitiche, G.F. Joyce (Joyce, 1989) e J.W. Szostak (Bartel e Szostak, 1993) hanno prodotto ribozimi con nuove specificità catalitiche. In un successivo sviluppo della tecnica, R.A. Lerner e P.G. Schultz (Lerner et al., 1991) hanno applicato l'idea dell'arricchimento per mezzo di un saggio specifico per un certo ligando alla ricerca di peptidi con particolari proprietà catalitiche e di legame. Utilizzando un metodo nel quale library peptidiche sono presentate sulle superfici fagiche (Devlin et al., 1990; Scott e Smith, 1990), essi trovarono il primo anticorpo catalitico. La scelta di una particolare strategia di ottimizzazione, naturale o artificiale, dipende dall'obiettivo da raggiungere. Naturalmente, la selezione naturale e quella artificiale possono essere combinate. La amplificazione (o auto amplificazione) degli individui è un passaggio comune a tutti questi esperimenti.
I virus sono modelli naturali di individui molecolari che si replicano
Lo studio del meccanismo dell'evoluzione naturale dovrebbe essere preferibilmente condotto con specie semplici autoreplicantisi e che coinvolgono soltanto poche reazioni biochimiche. Invece gli organismi autonomi coinvolgono reti complesse di reazioni biochimiche e, quindi, presentano troppe variabili per interpretare esperimenti evolutivi. Per questo motivo, S. Spiegelman (Spiegelman, 1971) scelse i virus come primi semplici replicatori. I virus sono particelle infettive, consistenti di acidi nucleici racchiusi in un capside proteico. Essi non hanno un apparato metabolico proprio e perciò non sono organismi viventi autonomi. Dopo l'infezione di una cellula ospite e l'iniezione nella cellula di acido nucleico virale, l'informazione genetica virale è letta e tradotta preferenzialmente in competizione con il DNA cellulare. Spesso, tutto ciò è fatale alla cellula ospite perché tutte le sue risorse vengono devolute alla sintesi di nuove particelle virali.
I virus mostrano una efficienza di replicazione sorprendente, con rapidi tempi generazionali, alta densità di popolazione e, a causa dell'alto tasso di errore nella replicazione, rapido adattamento alle costrizioni selettive. I virus sono dunque idealmente adatti per esperimenti di evoluzione molecolare.
Dal punto di vista delle indagini evolutive, il batteriofago Qf3 è probabilmente il sistema virale meglio studiato. Il suo genoma di RNA a singolo filamento (4200 nucleotidi) è stato sequenziato (Mekler, 1981), la sua morfologia e le sue caratteristiche biochimiche ben comprese (Kuo et al., 1975), e il suo ciclo virale infettivo elucidato (Biebricher e Eigen, 1988). Poiché la replicasi di Qβ, l'enzima per la replicazione codificato dal fago, manca di un meccanismo di correzione degli errori, il tasso di errore di replicazione è molto alto; esso è pari a 3 ∙ 10-⁴, corrispondente in media a 1,3 errori per genoma (Batschelet et al., 1976). Perciò Qβ è capace di sfuggire alle costrizioni ambientali con un processo evolutivo rapido. Nella tabella (tab. I) sono riportati il tasso di errore, la superiorità e la lunghezza massima della catena calcolata e sperimentale per diversi tipi di organismi.
Virus in coltura continua: il sistema cellstat
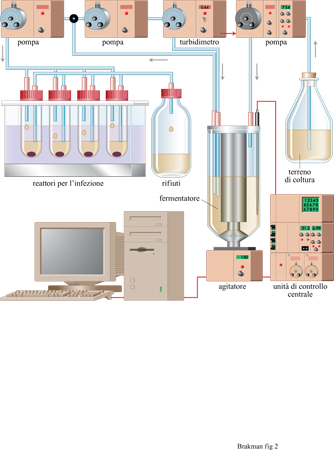
La competizione in una popolazione di individui emerge quando il numero totale di individui è mantenuto costante; questo è descritto dalle equazioni [3] e [4]. Una popolazione può essere mantenuta numericamente costante mantenendo costanti tutte le risorse necessarie. Comunque, appena nasce per caso una variante che è più adatta dell' originale, la nuova popolazione può sorpassare la prima raggiungendo una concentrazione di individui totale più alta. In effetti, gli eventi evolutivi sono di solito rivelati con l'emergenza di specie a replicazione più veloce. Mantenendo le condizioni costanti, l'applicazione di una qualsiasi nuova pressione selettiva aumenta la visibilità della fitness di alcuni individui. In linea di principio, è possibile avere delle colture continue di virus per studi evolutivi. Poiché i virus sono parassiti, essi non crescono da soli, ma hanno bisogno di un substrato di batteri ospiti per replicarsi. Queste cellule batteriche ospiti possono essere prodotte in un reattore a flusso continuo per la coltivazione dei batteri.
La produzione continua di virus può essere realizzata con un apparato detto cellstat (fig. 2) che può essere diviso in due unità principali (Husimi et al., 1982; Schwienhorst et al., 1996): il fermentatore, che è alimentato con un turbidimetro (cioè, controllato mediante una misurazione continua, on line, della torbidità della coltura), e che genera colture di cellule ospiti con una costante riproducibilità fisiologica nella fase di crescita esponenziale; i reattori cellstat, dove un flusso stazionario delle cellule ospiti è infettato con i virus, raggiungendo le condizioni di stato stazionario e producendo così quantità costanti di fago, di batteri infettati e non infettati. La separazione tra la coltura batterica e la coltura fagica evita arte fatti dovuti a coevoluzione. Infatti, la coltura parallela dell' ospite e del parassita in un singolo recipiente porterebbe inevitabilmente a una popolazione batterica resistente ai virus, rendendo impossibile l'interpretazione dell'esperimento. Invece, qualsiasi batterio potenzialmente resistente all'infezione viene rapidamente eliminato dal sistema cellstat.
Quando nei reattori si raggiunge uno stato stazionario, si possono cominciare esperimenti di evoluzione applicando nuove e definite pressioni selettive di tipo fisico, chimico o biologico. Le costrizioni applicate al sistema possono portare alla selezione naturale dei fagi che in queste condizioni si replicano più efficacemente, e sono quindi definiti come i più adatti. Così, con questo apparato si possono simulare infezioni virali di organismi superiori o di cellule in coltura e si possono verificare nuove strategie antivirali.
In un esperimento tipico (Lindemann, 1992), Qβ è stato esposto all'influenza di anticorpi monoclonali diretti contro la proteina del capside virale, 'mimando' quindi la risposta immunitaria di un organismo superiore a una infezione virale. Questo ha permesso ad alcuni fagi della popolazione di sfuggire alla pressione selettiva, mutando il gene che codifica la proteina del capside. L'analisi delle proteine ha mostrato che queste sostituzioni di basi erano espresse come epitopi antigenici alterati sulla superficie virale. Coltivando successivamente i mutanti resistenti all'anticorpo in assenza di pressione selettiva, il fago tornava a essere completamente di tipo selvatico. In un altro esperimento (Kettling, 1996) un fago fd, geneticamente modificato per l'espressione di peptidi sulla superficie virale (Zacher et al., 1980) e contenente una sequenza codificante per la resistenza alla tetraciclina, è stato inizialmente coltivato nel cellstat in assenza di questo antibiotico e ha perso di conseguenza il gene inserito codificante la resistenza alla tetraciclina. Quando, in un secondo esperimento, lo stesso fago è stato coltivato sotto la pressione selettiva dell'antibiotico, il gene aggiuntivo è stato ereditato stabilmente per diverse generazioni fagiche. Così, utilizzando coltivazioni continue dei fagi Qβ e fd, è stato possibile dimostrare qualitativamente e quantitativamente per la prima volta come funzionano i meccanismi utilizzati dai virus per superare una terapia antivirale. Similmente, è stato dimostrato come i virus reagiscono per ottimizzare il genoma, relativamente alla velocità di replicazione, rimuovendo tutti i carichi genetici inutili. In esperimenti futuri, diversi sistemi di espressione fagica potranno essere oggetto di esperimenti evolutivi nel cellstat.
La 'macchina per trasferimenti in serie': l'evoluzione dei replicatori molecolari
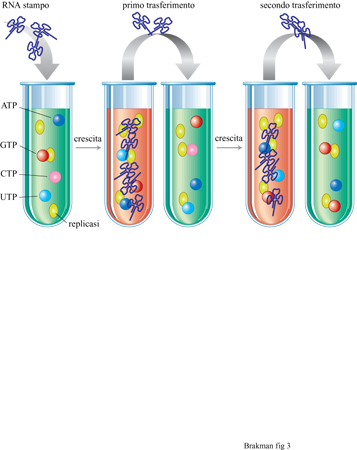
Con esperimenti utilizzanti il cellstat è stata studiata la crescita competitiva tra gli organismi viventi. Tale competizione può essere studiata anche in vitro, cioè con specie molecolari replicantisi. Il replicatore molecolare più ampiamente utilizzato negli esperimenti evolutivi è quello di Qβ costituito dalla replicasi di Qβ purificata, che utilizza RNA virale come stampo e nucleosidi trifosfati come substrati monomerici. Spiegelman (Spiegelman et al., 1965) fu il primo che, utilizzando tale sistema di replicazione, applicò condizioni costanti (simili al cellstat). Nel concetto dei trasferimenti in serie (fig. 3), una specie di RNA viene amplificata in vitro fino a una certa concentrazione massima a cui segue una diluizione della popolazione molecolare in una nuova soluzione. Così, il continuo alternarsi di fasi di amplificazione e di diluizione simula le caratteristiche descritte dal cellstat. I replicatori molecolari sono forzati a evolvere durante il periodo di crescita variando le condizioni di crescita (per esempio, aumentando la temperatura di reazione o aggiungendo sostanze che interferiscono con la crescita stessa). I prodotti dell'evoluzione possono essere analizzati dopo ciascun trasferimento, fornendo così informazioni sul meccanismo molecolare di questo processo. Se non vengono applicate costrizioni esterne, la velocità di replicazione determina la composizione della popolazione. Per esempio, sebbene la crescita dell'RNA di Qβ possa essere mantenuta senza pressione selettiva su un numero sostanzialmente indefinito di trasferimenti seriali, la sua infettività diminuisce con il tempo. Ciò deriva da una sostanziale perdita di nucleotidi, che permette ai genomi più corti rimasti di replicarsi più velocemente.
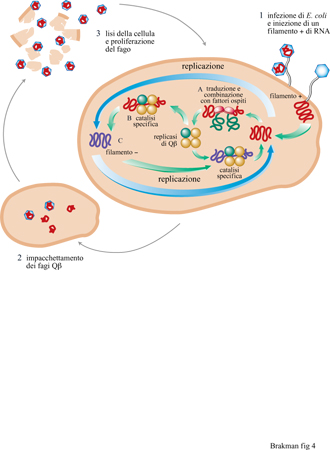
In generale, è importante eseguire esperimenti di evoluzione in vitro in condizioni ottimali, in particolare vicino alla soglia di errore, come suggerito dalla teoria. Gli esperimenti di Spiegelman furono condotti molto al di sotto dell'errore della replicasi di Qβ. Ne deriva che in questi esperimenti il processo evolutivo è risultato piuttosto lento. Il sistema di Spiegelman è stato nuovamente investigato in dettaglio da C.K. Biebricher e collaboratori (Biebricher, 1983; Biebricher et al., 1983; Biebricher et al., 1984). Questi ricercatori hanno risolto il meccanismo della replicazione di Qβ, hanno ricavato delle relazioni quantitative che descrivono il ciclo di reazione e hanno confermato queste leggi con alcuni esperimenti. Quando l'RNA stampo di Qj3 (cioè il filamento positivo) e i precursori dell'RNA vengono incubati con la replicasi di Qβ, la replicazione inizia con la produzione del filamento complementare negativo, che a sua volta serve come stampo per produrre altri filamenti positivi. Nel passaggio successivo, lo stampo e l'enzima sono riciclati per dissociazione dai loro complessi binari, per essere di nuovo soggetti allo stesso processo (fig. 4). Entrambi i filamenti, positivo e negativo, devono essere presenti in una forma a singolo filamento per poter essere utilizzati come stampi dalla replicasi; i doppi filamenti, risultanti dall' appaiamento di un filamento positivo e uno negativo, sono inattivi rispetto alla replicazione.
Guardando più da vicino il sistema della replicasi di Qβ, si possono distinguere quattro differenti tipi di andamento della reazione, a seconda dello stadio del processo. Nello stadio iniziale (diluito) della reazione di replicazione, la concentrazione di RNA aumenta costantemente mentre la quantità di enzima rimane costante. Fino a quando la replicasi di Qβ è disponibile in eccesso, la concentrazione dell'RNA aumenta esponenzialmente. Quando si raggiunge la saturazione di tutte le molecole di enzima con l'RNA stampo la crescita diventa lineare. A concentrazioni di RNA ancora maggiori, i filamenti positivi e negativi si combinano in una reazione bimolecolare per formare doppi filamenti inattivi e, come risultato, il tasso di crescita diminuisce ancora fino a diventare zero, quando la sintesi di nuovi filamenti è bilanciata esattamente dalla perdita di filamenti stampo a causa della formazione di doppi filamenti. Tempi di reazione più lunghi portano alla fine a una carenza di nucleotidi, alla completa inattivazione dell'RNA di nuova produzione e quindi all'interruzione della reazione. La comprensione del comportamento cinetico del sistema di replicazione di Qβ ha permesso di predire, in questo caso particolare con precisione a priori, l'esito degli esperimenti evolutivi. Determinando sperimentalmente alcuni parametri per specie isolate, si può simulare la loro crescita in una miscela idealmente diluita di specie differenti, integrando numericamente tutte le equazioni che defmiscono il completo meccanismo di reazione.
Per esempio, è stato calcolato teoricamente, e poi verificato sperimentalmente, che la crescita di una miscela di specie differenti non è semplicemente rallentata quando raggiunge l'inizio della fase di crescita lineare; piuttosto, la costrizione selettiva cambia così efficientemente che viene preferita una specie che mostra un basso o moderato tasso di replicazione. Questa è una diretta conseguenza delle condizioni di crescita lineari che sono caratterizzate dal fatto che una risorsa essenziale, la replicasi di Qβ, diviene l'elemento limitante. Così, la selezione favorisce non più la massima fecondità, ma il competitore che meglio sfrutta una risorsa comune.
L'interpretazione degli esperimenti evolutivi è particolarmente semplice nella fase di crescita esponenziale, perché in queste condizioni i valori di selezione sono identici ai tassi di replicazione. Le specie possono essere mantenute in una fase esponenziale compensando la crescita delle molecole che si replicano (organizzazione costante) oppure diluendo le specie in crescita in una nuova soluzione (trasferimento seriale). Tuttavia, quando avvengono dei cambiamenti evolutivi, cioè vengono selezionati mutanti con un certo vantaggio, i tassi di replicazione aumentano in maniera imprevedibile. Perciò sono necessarie misure in tempo reale del tasso di replicazione, per programmare in maniera accurata ulteriori esperimenti evolutivi.
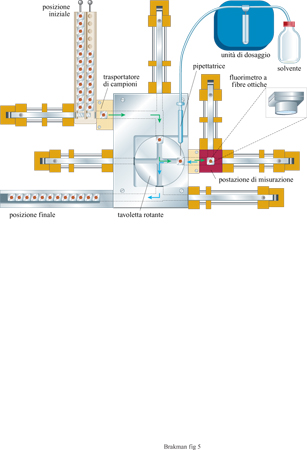
A Gottinga è stata costruita una macchina per eseguire automaticamente esperimenti di trasferimento in serie (fig. 5). I recipienti di reazione e i trasportatori di campioni sono costruiti in argento rivestito di materiale biologicamente inerte (oro). Cominciando dalla posizione di partenza i campioni sono trasportati alla postazione di misurazione, che è termostatata a una temperatura di crescita ottimale. La reazione di amplificazione viene innescata da un salto di temperatura. La concentrazione di acidi nucleici continuamente prodotti può essere seguita direttamente con un fluorimetro a fibre ottiche. Il bromuro di etidio, presente nella soluzione di reazione, si intercala negli acidi nucleici con un aumento concomitante di intensità della fluorescenza che viene misurata. Il numero di fotoni emessi è proporzionale alla concentrazione dell'RNA, per cui l'intensità della fluorescenza rivelata controlla il dispositivo automatico di pipettamento (attraverso un processore centrale). Appena viene superata la concentrazione massima fissata di RNA, la pipetta robot rimuove un'aliquota della soluzione e la trasferisce al vicino trasportatore di campioni che a sua volta è condotto alla postazione di misurazione quando il primo trasportatore la abbandona spostandosi nella posizione di stop.
L'evoluzione di una quasispecie di RNA sotto l'influenza selettiva delle ribonucleasi TI e A è stata uno dei primi fenomeni investigati sperimentalmente con questo strumento (Strunk, 1993). L'RNasi A è particolarmente interessante perché taglia l'RNA a livello delle pirimidine. Ogni regione pirimidinica nel filamento positivo richiede una regione purinica nel filamento negativo e viceversa. Una semplice sostituzione non offre una soluzione al problema dell'evoluzione. Aumentando progressivamente la concentrazione dell'RNasi A durante 91 trasferimenti in serie, si è evo Iuta una nuova quasispecie che è resistente a questo enzima. L'analisi del suo genotipo ha poi rivelato quali erano i motivi che conferivano resistenza. Poiché la endonucleasi RNasi A attacca specificamente l 'RNA a singolo filamento in corrispondenza delle basi pirimidiniche (U, C), la specie di RNA selezionata è riuscita a proteggersi dai tagli endonucleolitici riducendo lo stampo originale di 22 nucleotidi e così diminuendo al minimo le regioni a singolo filamento nella sua struttura secondaria. Inoltre, le basi pirimidiniche (bersagli per l'RNasi A) si sono spostate dal filamento + al filamento -, che si replica 100 volte più velocemente rispetto al filamento +, sfuggendo quindi agli attacchi dell'RNasi.
La macchina multicanale per i trasferimenti in serie: l'evoluzione di una 'piccola' quasispecie
Come è stato dimostrato, il cellstat e la macchina per trasferimenti in serie sono strumenti adatti per investigare l'evoluzione molecolare. Entrambe le macchine sono adatte a studiare le quasispecie come entità globali. In linea di principio, una quasispecie può essere diluita fino al livello di sottopopolazioni, o anche a quello di singole molecole, e ciascun clone può essere compartimentalizzato. In questo modo, gli adattamenti evolutivi di singole varianti possono essere studiati in parallelo. Inoltre, si può osservare l'effetto di una varietà di costrizioni esterne su mutanti di una quasispecie. Per questo scopo è stata costruita una macchina che permette di studiare in parallelo 96, o anche 960, cloni individuali che si replicano (Schober et al., 1995).
Precedentemente, è stata descritta la selezione naturale in vitro con la replicasi di Qβ purificata. Oltre a questa, esistono altre procedure di replicazione molecolare come la PCR, SDA (Strand Displacement Amplification, amplificazione per spostamento del filamento) o 3SR (Self-Sustained Sequence Replication, replicazione autosostenuta di sequenze). A differenza dell'amplificazione isoterma di Qβ, o delle reazioni isoterme SDA e 3 SR, la PCR dipende da una alternanza ciclica di tre temperature di reazione diverse. Il nuovo robot estende le caratteristiche della precedente macchina per il trasferimento in serie, rendendo possibili esperimenti evolutivi assistiti dalla PCR.
La macchina multicanale per i trasferimenti in serie descritta da A. Schober e collaboratori (Schober et al., 1995) consiste di tre blocchi di alluminio, ciascuno con 96 o 960 pozzetti nei quali sono posti i contenitori in cui avvengono le reazioni. Le temperature di questi tre blocchi metallici sono controllate singolarmente da un computer. Le soluzioni contenenti i sistemi molecolari sono caricate in contenitori di plastica con cavità che si adattano ai pozzetti del sostegno metallico. Queste celle sono coperte, a tenuta d'aria, con un altro foglio di plastica e possono essere spostate da un blocco all'altro a temperature differenti come definito dalla PCR. In questi blocchi si possono ottenere cambiamenti della temperatura in pochi secondi. Come negli esperimenti condotti nella macchina 'a singolo canale', le concentrazioni degli acidi nucleici prodotti dalla PCR possono essere seguite on line con un fluorimetro a fibre ottiche di vetro a 960 canali. Inoltre, viene installato un robot capace di eseguire le diluizioni necessane.
l primi esperimenti con questa macchina furono condotti per investigare l'influenza di varie sostanze chimiche sull'amplificazione del DNA mediante PCR. È inoltre possibile usare questa macchina automatica per esaminare la selezione artificiale in assenza di crescita competitiva. Selezioni di nuove varianti desiderate possono essere ottenute mediante la rivelazione simultanea di 960 varianti con saggi che collegano una proprietà nuova o funzionalmente ottimizzata a un segnale fluorescente, per esempio l'incorporazione di analoghi fluorescenti dei nucleotidi da parte di una nuova polimerasi mutante.
Immagini di eventi evolutivi: il reattore capillare
Una caratteristica di tutte le macchine descritte finora è che i sistemi di reazione investigati con questi strumenti esibiscono una omogeneità spaziale. Questa omogeneità permette l'osservazione della competizione tra gli individui. J.S. McCaskill ha descritto una macchina con un ambiente di reazione unidimensionale che, a differenza degli strumenti descritti finora, creerebbe una disomogeneità spaziale vantaggiosa per ottenere informazioni aggiuntive sul sistema che si replica (Bauer et al., 1989). Questo è dovuto all'effetto accoppiato della reazione e della diffusione, che determina una velocità di propagazione costante, a differenza della semplice diffusione descritta dalla tipica relazione distanza al quadrato/tempo (Fisher, 1994):
v ≈ 2√kD . [7]
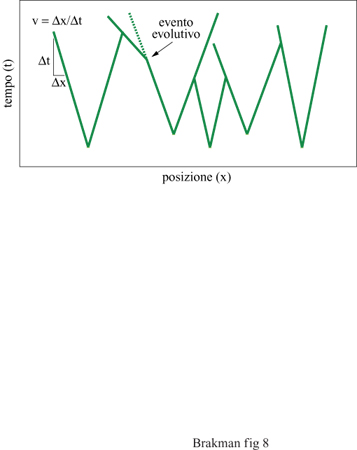
Secondo questa equazione, la velocità di espansione v di una specie che cresce lungo uno 'spazio vitale' unidimensionale dipende semplicemente dal suo tasso di replicazione k e dalla sua costante di diffusione D. Un replicatore molecolare quindi si diffonde come una 'nube vagante'. Lo strumento che realizza questa idea è stato introdotto da McCaskill (McCaskill e Bauer, 1993); si tratta di un reattore che consiste di 144 capillari paralleli riempiti con il sistema di replicazione insieme a un colorante intercalante per vi sualizzare le molecole di RNA di nuova sintesi; è così possibile controllare il progredire spaziale in questo ambiente. Le singole molecole di RNA sono la sorgente delle colonie in crescita esponenziale. Dopo un breve periodo di tempo, questi cloni possono espandersi mediante replicazione e diffusione solamente lungo il capillare, poiché la loro crescita è limitata nelle altre due dimensioni dalle pareti esterne. Questa costrizione determina dunque un'onda di concentrazione che procede con velocità costante. Una camera CCD installata al di sopra dell'area dei capillari può acquisire immagini a intervalli di tempo regolari. Serie di queste immagini possono poi essere combinate in immagini spazio/tempo (fig. 8) per documentare la diversità degli eventi evolutivi e, inoltre, la storia delle singole colonie nel reattore. Le colonie originate da molecole iniziali differenti possono essere distinte perché il loro tasso di replicazione è accoppiato alla loro velocità di espansione. Così, cloni con tassi di replicazione differenti presentano nelle immagini risultanti gradi enti differenti e caratteristici. Inoltre, ciascun evento evolutivo connesso con una variazione del tasso replicativo altera la velocità di avanzamento del fronte dell' onda che si propaga, risultando in un cambiamento della pendenza della curva spazio/ tempo.
L'ovvio vantaggio del reattore capillare sta nella semplicità con cui si possono seguire eventi mutazionali. Dopo avere terminato l'esperimento, i campioni possono essere sequenziati. In questo modo, le strategie molecolari utilizzate per sfuggire alle costrizioni della selezione possono essere seguite passo per passo.
Evoluzione molecolare applicata: ottimizzazione di biomolecole funzionali per mezzo di strategie evolutive
Ogni reazione di un sistema auto organizzante si che è 'inizializzata' e catalizzata da un biopolimero funzionale (acido nucleico o proteina) fa uso del principio della 'chiave' e 'serratura'. Quindi l'attività di qualsiasi macromolecola biologica è strettamente correlata alla sua struttura tridimensionale; per esempio, l'affinità di legame con molecole come anticorpi, RNA antisenso, acidi nucleici che bloccano la codificazione e aptameri (molecole polimeriche che si adattano), o le attività catalitiche con molecole come alcuni anticorpi catalitici artificiali, ribozimi e enzimi ubiquitari. Diversi tra questi biopolimeri naturali non sono al momento del tutto adatti per applicazioni mediche e tecnologiche. Essi spesso presentano bassa stabilità e basse attività catalitiche se usati in ambienti artificiali caratterizzati da alte temperature, valori di pH estremi o solventi non acquosi. Nella maggioranza dei casi, queste biomolecole interagiscono specificamente con i loro substrati naturali ma lasciano immodificati i substrati di interesse applicativo o artificiali. Vi è quindi molto interesse per nuovi metodi che permettano di modificare le proprietà dei biopolimeri.
l metodi per l'ottimizzazione di molecole effettrici includono progettazioni 'razionali' e'irrazionali'. l metodi razionali sono spesso utilizzati per ottimizzare gli enzimi: essi sono basati su una conoscenza perlomeno incompleta dei motivi strutturali che determinano alcune funzioni. Le strategie coinvolgono sostituzioni specifiche dei monomeri (amminoacidi nel caso degli enzimi) e lo studio delle attività alterate delle strutture che ne risultano. Con una informazione meno precisa delle relazioni struttura/funzione, mutazioni casuali devono essere introdotte nelle regioni selezionate. Le parti di una sequenza che si pensa siano importanti per alcune funzioni possono essere sostituite da oligonucleotidi sintetici a sequenza casuale, ottenendo numerose varianti che possono essere sottoposte a screening per evidenziare nuove proprietà. Tuttavia, per investigare sequenze più lunghe, fino alla grandezza di geni interi, può essere preferibile introdurre mutazioni puntiformi casuali lungo l'intero frammento, tipicamente con una frequenza di una o poche mutazioni per molecola. Ciò si può ottenere, per esempio, copiando con una polimerasi poco accurata una sequenza di acido nucleico. Le library di mutanti risultanti, create sia mediante oligonucleotidi casuali sia mediante mutagenesi puntiforme casuale, possono essere esposte a saggi per valutare le attività individuali, o ad ambienti artificiali per selezionare determinati individui. Con cicli sequenziali di mutagenesi casuale e selezione, l'evoluzione delle molecole può essere indirizzata verso nuove strutture funzionali e verso l'adattamento a nuove richieste. Poiché né la mutagenesi con oligonucleotidi casuali né la replicazione inaccurata sono combinatorie, la loro abilità di esplorazione dello spazio delle sequenze in cerca di nuove funzioni è limitata. Dunque, gli approcci evolutivi possono essere migliorati con procedure di ricombinazione (omologa) consistenti nel rapido accumulo di mutazioni vantaggiose. Inoltre, le differenti strategie (mutagenesi sito-specifica o casuale, inserzione di cassetta) possono essere combinate creando una varietà simile a quella esistente in natura.
Una connessione fisica o logica tra un gene e la corrispondente biomolecola funzionale (accoppiamento genotipo- fenotipo) è un prerequisito necessario per la selezione, e questa rimane la difficoltà principale delle ottimizzazioni evolutive in laboratorio. L'informazione per una certa funzione è codificata nell'acido nucleico di un gene. L'espressione di questo gene porta alla proteina funzionale corrispondente. La connessione tra geni e proteine può essere realizzata o con una compartimentazione spaziale di macromolecole isolate, dove l'identità di un composto è data o dalla sua localizzazione in una disposizione (micro )strutturata (sistema in vitro) o dalla tecnica recentemente sviluppata e detta phage display o bacterial display. La tecnica del phage display utilizza l'espressione di una proteina estranea, o un frammento proteico, e la sua presentazione sulla superficie di un batteriofago. Il fago contiene di solito l'informazione che codifica la proteina presentata sulla sua superficie e così garantisce l'accoppiamento genotipo/fenotipo. Similmente, proteine esogene possono essere espresse sulla superficie di un batterio. Tuttavia, la grandezza delle library di questi sistemi in vivo è limitata dalla necessità di trasformare l'insieme di varianti di DNA in cellule batteriche ospiti. In aggiunta, le proteine desiderate possono essere altamente tossiche per la cellula batterica e quindi difficilmente espresse. Per evitare questa limitazione, si possono utilizzare sistemi in vitro basati sull'accoppiamento trascrizione/traduzione. Con questo approccio, la costruzione di library di mutanti, l'espressione delle corrispondenti proteine e la selezione vengono fatte interamente in forma di estratti cellulari. Al contrario, gli RNA catalitici (ribozimi) posseggono sia un genotipo sia un fenotipo, e quindi sono molecole ideali da utilizzare in esperimenti in vitro sui modelli di evoluzione. Grandi insiemi di RNA genotipicamente eterogenei possono essere soggetti a ottimizzazioni evolutive che portano a nuovi e desiderati fenotipi. Con queste nuove strategie, cambiamenti genetici specifici possono essere correlati con alterazioni di specifiche proprietà catalitiche definite. G.F. Joyce ha descritto alcuni di questi approcci evolutivi diretti per molecole di RNA catalitico (Breaker e Joyce, 1994).
La scelta del metodo di selezione appropriato dipende da quali proprietà della proteina interessano. Una importante applicazione delle ottimizzazioni evolutive consiste nel miglioramento del legame di farmaci peptidici o proteici ai loro recettori. I recettori possono essere fissati su apposite superfici producendo così supporti d'affinità per applicazioni cromatografiche; soluzioni di library peptidiche (per esempio, esposte sulla superficie di fagi) possono essere applicate a colonne cromato grafiche preparate con questi supporti, fino a quando restano legati solo quei peptidi che si legano fortemente ai recettori immobilizzati. Naturalmente, questa procedura può essere reiterata fino al punto di ottenere molecole con proprietà ottimali.
Biotecnologia evolutiva: prospettive
L'evoluzione molecolare è un processo che può essere descritto con modelli matematici e verificato con esperimenti condotti in condizioni ben defrnite. Il meccanismo mediante il quale la natura ha generato la diversità della vita può così essere trasferito in laboratorio e lì riprodotto a una velocità molto accelerata. Substrati macromolecolari naturali possono essere fatti evolvere direttamente in sostanze attive altamente efficienti aventi le proprietà desiderate.
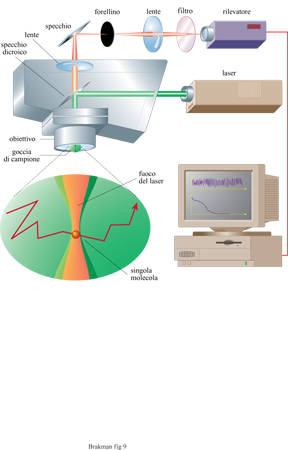
Vi è una costante ricerca di nuovi ed efficaci prodotti farmaceutici con alte affinità per i propri recettori. Poiché sia il DNA sia l'RNA, come pure i peptidi, possono servire a questo scopo e possono essere rapidamente sintetizzati in quantità dell'ordine delle nanomoli, sono state messe a punto tecniche che permettono una miniaturizzazione utilizzando supporti costituiti da multi strati di silicio e di plastica. Sintesi parallele delle distribuzioni dei mutanti macromolecolari possono essere realizzate con questo metodo. Inoltre, l'analisi delle attività richieste può essere anche eseguita abbinando questi apparati con le tecniche di spettroscopia a correlazione di fluorescenza (FCS) che sono state sviluppate recentemente (Eigen e Rigler, 1994). Questo metodo, che registra le correlazioni spazio-temporali tra segnali fluttuanti di luce, accoppiato a dispositivi in grado di intrappolare singole molecole in un campo elettrico (fig. 9), permette il monitoraggio di concentrazioni fino a 10-¹⁵M, o anche più basse, senza bisogno di amplificazione. Con questa tecnica, le 'cavità di luce laser' essenziali per il rilevamento di segnali fluorescenti sono rappresentate da elementi a forma cilindrica con un raggio di 200 nm e una lunghezza di 2000 nm. Come conseguenza di queste piccole dimensioni, circa 10⁸ varianti diverse possono essere determinate in una area di l cm²). L'FCS e i sistemi di espressione del fenotipo controllati geneticamente (phage display e bacterial display) aprono dunque nuovi orizzonti alla evoluzione molecolare diretta.
La comprensione del comportamento evolutivo dei virus fornisce nuove prospettive alle strategie antivirali. I virus, e specialmente l'HIV, possono sfuggire alla risposta immunitaria con l'aiuto dei loro enzimi di replicazione che introducono molte mutazioni. È stata così proposta la possibilità di influenzare la replicazione virale mediante l'aggiunta di alcuni composti chimici che possono diminuire sempre di più la fedeltà dell'enzima, fino a superare la soglia di errore. Come risultato, il genoma virale accumula errori fino a diventare una sequenza priva di informazione che non ha più somiglianza con la sequenza originale e non può replicarsi ulteriormente.
Con il progredire della ricerca sulla autoevoluzione, la varietà delle tecniche di selezione del 'più adatto' continuerà ad apportare sorprendenti miglioramenti alla biotecnologia evolutiva.
Bibliografia citata
BARTEL, D.P., SZOSTAK, J.W. (1993) Isolation of new ribozymes from a large pool of random sequences. Science, 261, 1411-1418.
BATSCHELET, E., DOMINGO, E., WEISSMANN, C. (1976) The proportion of revertant and mutant phage in a growing population as a function ofmutation and growth rate. Gene, 1, 27-32.
BAUER, G.J., MCCASKILL, J.S., OTTEN, H. (1989) Traveling waves of in vitro evolving RNA. Proc. Natl. Acad. Sci. USA, 86, 7937-7941.
BIEBRICHER, C.K. (1983) Darwinian selection of self-replicating RNA. In Evolutionary biology, voI. 16, a c. di Hecht B., Wallace M.K., Prance G.T., New York, Plenum Press, pp. 1-52.
BIEBRICHER, C.K., EIGEN, M. (1988) Kinetics of RNA replication of Qβ replicase. In RNA genetics, voI. 1, a c. di Domingo E., Holland J.J., Ahlquist P., Boca Raton, CRC Press, pp. 1-21.
BIEBRICHER, C.K., EIGEN, M., GARDINER, W.C. (1983) Kinetics of RNA replication. Biochemistry, 22, 2544-2559.
BIEBRICHER, C.K., EIGEN, M., GARDINER, W.C. (1984) Kinetics of RNA replication: plus-minus asymmetry and double strand formation. Biochemistry, 23, 3186-3194.
BOERLIJST, M.C., BONHOEFFER, S., NOWAK, M.A. (1996) ViraI quasispecies and recombination. Proc. Royal Soc. B (London) - Series B. Biological Sciences, 263, 1577-1584.
BREAKER, R.R., JOYCE, G.F. (1994) Inventing and improving ribozyme function: rational design versus iterative selection methods. Trends Biotechnol., 12, 268-275.
DEVLIN, U., PANGANIBAN, L.C., DEVLIN, P.E. (1990) Random peptide libraries: a source of specific protein binding molecules. Science, 249, 404-406.
DOMINGO, E. et al. (1985) The quasispecies (extremely heterogeneous) nature of viraI RNA genome populations: biological relevance - a review. Gene, 40, 1-8.
DOMINGO, E., SABO, D., TANIGUCHI, T., WEISSMANN, C. (1978) In vitro nucleotide sequence heterogeneity of an RNA phage population. CelI, 13, 735-744.
EIGEN, M., MCCASKILL, lS., SCHUSTER, P. (1989) The molecular quasispecies. Adv. Chem. Phys., 75, 149-263.
EIGEN, M., RIGLER, R. (1994) Sorting single molecules: application to diagnostics and evolutionary biotechnology. Proc. Natl. Acad. Sci. USA, 91, 5740-5747.
EIGEN, M., SCHUSTER, P. (1977) The hypercycle: a principle of natural self-organization. Part A: emergence ofthe hypercycle. Naturwissenschaften, 64, 541-565.
EIGEN, M., WINKLER-OSWATITSCH, R., DRESS, A. (1988) Statistical geometry in sequence space: a method of quantitative comparative sequence analysis. Proc. Natl. Acad. Sci. USA, 85, 5913-5917.
FISHER, R. (1994) The wave of advance of advantageous genes. Ann. Eugenics, 7, 355-369.
HAMMING, R.W. (1980) Coding and information theory. Englewood Cliffs, Prentice-Hall.
HUSIMI, Y., NISHIGAKI, K., KINOSHITA, Y., TANAKA, T. (1982) Cellstat - a continuous culture system of a bacteriophage for the study of the mutation rate and the selection process of the DNA leve l. Rev. Sci. Instrum., 53, 517-522.
IRVINE, D., TUERK, C., GOLD, L. (1991) Selection: systematic evolution of ligands by exponential enrichment with integrated optimization by non-linear analysis. J. Mo!. Biol., 222, 739-76l.
JONES, B.L., ENNS, R.H., RANGNEKAR, S.S. (1976) On the theory of selection of coupled macromolecular systems. Bull. Math. Biol., 38, 15-28.
JOYCE, G.F. (1989) Building the RNA world: evolution of catalytic RNA in the laboratory. In Molecular biology of RNA.- UCLA symposia on molecular and cellular biology, a c. di Cech T. R., New York, Alan R. Liss Inc., pp. 361-37l.
KETTLING, U. (1996) Untersuchungen = Evolution von F-spezifischen, filamentosen Bakteriophagen in kontinuierlicher Kultur. Diploma Thesis, Techuical University of Braunschwelg.
KUO, C.H., EOYANG, L., AUGUST, 1T. (1975) Protein factors required for the replication of phage Qβ RNA in vitro. In RNA phages, a c. di Zinder N. D., Cold Spring Harbor, Cold Spring Harbor Laboratory Press, pp. 259-278.
LERNER, R.A., BENKOVIC, S.A., SCHULTZ, P.G. (1991) At the crossroads of chemistry and immunology: catalytic antibodies. Science, 252, 659-668.
LEUTHAUSSER, I. (1986) An exact correspondence between Eigen's evolution model and a two-dimensional Ising system. J. Chem. Phys., 84, 1884-1885. LINDEMANN, B.F. (1992) Kontrollierte Evolution von RNA-Phagen in kontinuierlicher Kultur. Ph. D. Thesis, Technical University of Braunschweig.
McCASKILL, J.S., BAUER, G. (1993) Images of evolution: origin of spontaneous RNA replication waves. Proc. Natl. Acad. Sci. USA, 90, 4191-4195.
MEKLER, P. (1981) Determination of nucleotide sequences of the bacteriophage Qβ genome: organization and evolution of an RNA virus. Ph. D. Thesis, University of Zurich.
RECHENBERG, I. (1973) Evolutionsstrategie. Stuttgart-Bad Caunstadt, Problemata Frommaun-Holzboog.
SCHOBER, A., WALTER, N., TANGEN, U., STRUNK, G., EDERHOF, T., DAPPRICH, J.G., EIGEN, M. (1995) Multichaunel PCR and serial transfer machine as a future tool in evolutionary biotechnology. Biotechniques, 18, 652-667.
SCHWIENHORST, A (1992) Grundlagen einer evolutiven Biotechnologie. Ph. D. Thesis, Aachen, Shaker-Verlag.
SCHWIENHORST, A., LINDEMANN, B.F., EIGEN, M. (1996) Growth kinetics of a bacteriophage in continuous culture. Biotechnol. Bioengin., 50, 217-22l.
SCOTT, J.K., SMITH, G.P. (1990) Searching for peptide ligands with an epitope library. Science, 249, 386-390.
SPIEGELMAN, S. (1971) An approach to the experimental analysis of precellular evolution. Q. Rev. Biophys., 4, 213-253.
SPIEGELMAN, S., HARUNA, J., HOLLAND, J.B., BEAUDREAU, G., MILLS, D. (1965) The synthesis of a self-propagating and infectious nucleic acid with a purified enzyme. Proc. Natl. Acad. Sci. USA, 54, 919-927.
STRUNK, G. (1993) Automatisierte Evolutionsexperimente in vitro. Ph. D. Thesis, Aachen, Shaker-Verlag.
THOMPSON, C.J., McBRIDE, J.L. (1974) On Eigen's theory of the self-organization of matter and the evolution of biological macromolecules. Math. Biosci., 21, 127-142.
WIEHE, T., BAAKE, E., SCHUSTER, P. (1995) Error propagation in reproduction of diploid organisms. A case study on single peaked landscapes. J. Theor. Biol., 177, 1-15.
ZACHER, A.N., STOCK, C.A, GOLDEN, J.W., SMITH, G.P. (1980) A new filamentous phage cloning vector: fd-tet. Gene, 9, 127140.
Bibliografia generale
EIGEN, M. (1971) Self-organization of matter and the evolution of biological macromolecules. Naturwissenschaften, 58, 466-565.
EIGEN, M. Steps towards life: a perspective on evolution. Oxford, Oxford University Press, 1992