Il mondo a RNA
Il mondo a RNA
Per coloro che sono interessati ai primissimi stadi dell'evoluzione è una sfida capire come abbiano avuto origine gli organismi viventi primordiali, e come questi effettuassero il processo più essenziale: la replicazione. La scoperta delle proprietà catalitiche dell'RNA ha dimostrato che una singola specie chimica può funzionare sia come genoma sia come catalizzatore, e ha suggerito che ci sia stata un'era, prima dell'evoluzione delle attuali cellule, in cui genomi costituiti da RNA erano replicati da enzimi fatti di RNA: il mondo a RNA. Negli anni Ottanta abbiamo appreso molto sulle notevoli proprietà catalitiche dell'RNA e sulla replicazione di genomi a RNA in una straordinaria varietà di microrganismi contemporanei. Queste conoscenze ci forniscono indicazioni sulla vita nel mondo a RNA, e suggeriscono come il mondo a RNA sia diventato il mondo a DNA che noi oggi conosciamo.
Introduzione
Allora il Signore Iddio plasmò dal suolo ogni sorta di bestie selvatiche e tutti gli uccelli del cielo e li condusse all 'uomo, per vedere come li avrebbe chiamati: in qualunque modo l'uomo avesse chiamato ognuno degli esseri viventi, quello doveva essere il suo nome. (Genesi 2: 19)
Il mondo a RNA è comparso quando W. Gilbert (1986) usò per la prima volta questa espressione per descrivere i primordi della vita sulla Terra. Nel mondo a RNA gli organismi possedevano genomi fatti di RNA che erano replicati da enzimi costituiti da RNA.
La nozione che una singola entità chimica, l'RNA, potesse funzionare sia come genoma sia come catalizzatore era un'elegante risposta a una domanda molto problematica: quali forme di vita hanno preceduto quelle che noi osserviamo oggi nel mondo a DNA? Per molto tempo si è ritenuto che solo gli acidi nucleici fossero in grado di immagazzinare e replicare le informazioni genetiche, e che solo le proteine potessero funzionare da catalizzatori. Questo rendeva difficile immaginare come potevano essersi evo Iute le cellule odierne. Nel nostro mondo, il mondo a DNA, i genomi costituiti da DNA si replicano tramite enzimi costituiti da proteine, e il DNA fornisce l'informazione per la sintesi delle proteine. Un sistema vivente primordiale basato solo su DNA o solo su proteine era inconcepibile, ed era ancora più assurdo ipotizzare che il DNA e la sintesi delle proteine su stampo avessero avuto origine nello stesso momento.
Uno schema semplice per l'evoluzione molecolare
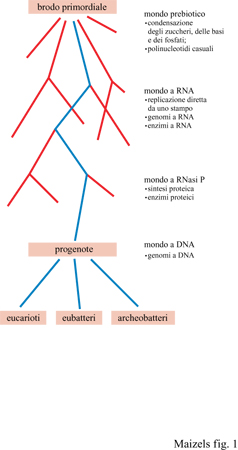
La dimostrazione che l'RNA potesse funzionare come catalizzatore (v. oltre) risolse il problema dell'uovo e della gallina che aveva afflitto la biologia evolutiva. Inoltre ciò richiamò l'attenzione dei biologi sul fatto che le attuali cellule costituiscono un archivio di storia evolutiva e consentì persino ai più stretti sperimentalisti di cominciare a meditare sulle implicazioni evolutive del proprio lavoro. Come risultato, in pochi anni le nostre conoscenze sull'evoluzione hanno fatto un gigantesco progresso. Adesso possiamo rappresentare le prime fasi dell'evoluzione con uno schema molto semplice: reazioni prebiotiche sulla Terra primordiale hanno generato l'RNA; l'RNA è divenuta la prima molecola in grado di autoreplicarsi; si sono sviluppati esseri viventi con genoma a RNA e sistemi biochimici relativamente semplici; questi si sono poi evoluti in organismi più complessi, in grado di sintetizzare proteine, per divenire infine le cellule moderne con genomi a DNA e apparati biochimici estremamente sofisticati che noi conosciamo (fig. 1).
L'RNA catalitico
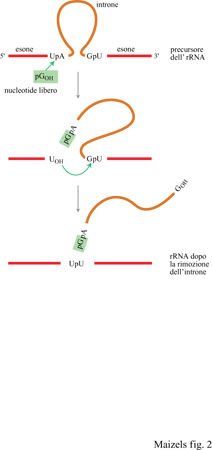
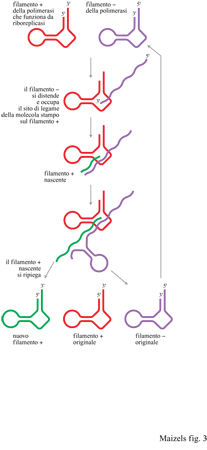
Nel 1981, Th. Cech stava studiando la biogenesi dell'RNA ribosomale (rRNA) nel ciliato Tetrahymena, quando fece una scoperta sorprendente (Cech et al., 1981): l'introne presente nel precursore dell'rRNA era in grado di rimuoversi autonomamente dalla molecola senza l'ausilio di una proteina catalizzatrice (fig. 2). La capacità dell'introne dell'rRNA di funzionare come un enzima implicava la possibilità che la primissima molecola della vita sia stata una RNA-replicasi in grado di catalizzare la sua stessa replicazione senza l'aiuto di una proteina. Questo ipotetico RNA avrebbe dovuto funzionare sia come materiale genetico sia come replicasi, cioè enzima della replicazione (fig. 3). La scoperta nel 1983 di un secondo tipo di RNA catalitico dette un'ulteriore conferma a questa osservazione (Guerrier-Takada et al., 1983). Da allora sono stati scoperti molti nuovi esempi di RNA catalitico, e le proprietà catalitiche dell'RNA sono state esaminate in grande dettaglio. Per differenziarli dagli enzimi proteici spesso gli RNA catalitici vengono definiti ribozimi (Cech e Bass, 1986).
Creare un passato virtuale: una riboreplicasi fatta di RNA copia l'RNA in RNA
Di recente, D. Bartel e collaboratori (Ekland et al., 1995), usando tecniche selettive per l'RNA note come SELEX hanno dimostrato che un piccolo RNA di circa 200 nucleotidi è in grado di usare ribonucleotidi-trifosfati come precursori per copiare una molecola-stampo di RNA (D. Bartel, comunicazione personale). L'esistenza di questo enzima prova un principio fondamentale del mondo a RNA: un enzima fatto di RNA può copiare l'RNA in RNA. Malgrado la versione attuale di questo enzima riproduca solo molecole di RNA di limitata lunghezza, in molti ritengono che non trascorrerà molto tempo prima che l'ingegneria genetica produca una riboreplicasi in grado di sintetizzare trascritti molto più estesi.
L'universo di tutte le possibili sequenze di RNA di una certa lunghezza - lo spazio delle sequenze dell'RNA - è veramente enorme. Perciò una riboreplicasi prodotta al giorno d'oggi con le tecniche dell'ingegneria genetica avrebbe sicuramente una sequenza diversa da quella del ribozima primordiale. Tuttavia, è noto come i prodotti di ripetute selezioni in vitro possano talvolta convergere su un'unica (o pochissime) soluzione strutturale che sia dotata di una certa attività enzimatica. Un'affascinante domanda è se l'analisi di questi campioni rivelerà l'emergere sistematico di somiglianze strutturali. Se così fosse, le ripetute selezioni avrebbero il potere di ricreare un passato virtuale, in cui sequenze moderne riprodurrebbero quelle antiche molecole che eseguivano le funzioni cruciali della vita primordiale.
Le cellule attuali come archivi viventi
Prima della scoperta dell'RNA catalitico, il punto di vista comune di biologi e chimici era che miliardi di anni di evoluzione avessero completamente cancellato la natura delle prime molecole in grado di autoreplicarsi e delle prime cellule viventi. Questa concezione era basata su un'intuizione assolutamente logica: poiché le cellule attuali sono complesse e sofisticate, devono avere scartato (o trasformato in modo irriconoscibile) le componenti macromolecolari e le strategie biochimiche usate nelle prime fasi dell'evoluzione. La scoperta dell'RNA catalitico modificò questo concetto: divenne possibile ipotizzare che la vita avesse avuto inizio con un RNA che funzionava sia come genoma sia come replicasi. Nello stesso tempo, poiché gli RNA catalitici esistono ancora nel mondo odierno a DNA, divenne lecito pensare agli organismi contemporanei come ad archivi viventi della storia dell'evoluzione.
Questa visione della biologia moderna come memoria storica, che risale fino all'origine della vita stessa, è caratterizzata da ciò che il filosofo Karl Popper definiva "potere esplicativo": riesce cioè a dare un significato ad alcune caratteristiche delle cellule moderne altrimenti incomprensibili. L'RNA è al centro di molte attuali reazioni essenziali ed esegue funzioni non facilmente spiegabili solo in termini chimici. Nella replicazione del DNA, per esempio, le basi del DNA sono prodotte come precursori ribonucleotidici poi convertiti dalla ribonucleotidereduttasi; inoltre il primo passo nella replicazione del DNA è la sintesi di un primer (innesco) di RNA. Un'origine primordiale dell'RNA spiega entrambe queste osservazioni: le basi dell'RNA sono precursori del DNA perché, quando si originarono le prime reazioni biosintetiche dei nucleotidi, queste producevano solamente RNA e l'uso vestigiale dell'RNA nella replicazione del DNA è un ricordo del periodo in cui esisteva soltanto l'RNA. La sintesi delle proteine fornisce altri esempi interessanti sul ruolo dell'RNA nelle attuali reazioni biochimiche essenziali. Sia la reazione della peptidiltransferasi ribosomale (Noller, 1993) che la maturazione del tRNA (RNA transfer o di trasporto) tramite l'RNasi p (Guerrier-Takada et al., 1983) sono reazioni catalizzate dall'RNA. La spiegazione più ovvia del ruolo fondamentale dell'RNA nella sintesi proteica è che il processo di sintesi proteica sia stato creato in un mondo a RNA e, di conseguenza, abbia utilizzato le specie macromolecolari a disposizione (principalmente o esclusivamente RNA) per adempiere alla nuova funzione. Quando venne perfezionato questo primitivo meccanismo della sintesi proteica basato sull'RNA, il ruolo centrale dell'RNA non fu, o non poté essere, sostituito. Una spiegazione alternativa del ruolo centrale dell'RNA nella sintesi proteica è che esso sia stato introdotto dall'evoluzione nel processo in un secondo tempo, rimpiazzando un meccanismo originario di natura totalmente differente. Questa spiegazione però non chiarisce perché venne scelto l'RNA come componente centrale dell'apparato di sintesi delle proteine, né perché l'apparato originale sia stato completamente scartato piuttosto che perfezionato.
La partecipazione dell'RNA a quasi tutti gli aspetti essenziali del flusso dell'informazione (replicazione, trascrizione, trascrizione inversa e traduzione) fa supporre che l'RNA sia comparso così precocemente nella storia della vita da rimanere di fatto implicato in tutti i processi molecolari fondamentali; inoltre la maggior parte delle reazioni basate sull'RNA o catalizzate dall'RNA nell' attuale mondo a DNA è, quasi sicuramente, superstite di un precedente mondo basato sull'RNA (Alberts, 1986). Senza dubbio, ci sono eccezioni: alcuni RNA catalitici attuali potrebbero essersi evoluti successivamente alla comparsa delle sofisticate cellule con genomi a DNA. Gli introni del Gruppo l, per esempio, molecole non essenziali che sono in grado di rimuoversi autonomamente dall'RNA messaggero (self-excising), in effetti potrebbero avere un'origine più recente.
Fossili molecolari
Sulla base dell' assunto che tracce determinanti del passato sono conservate nella biochimica delle cellule attuali, si può cominciare a descrivere un percorso evolutivo plausibile ragionando, a ritroso, su quanto si conosce degli attuali genomi ed enzimi. Useremo la locuzione fossili molecolari per indicare quelle molecole che sono cruciali per tale analisi. Nelle attuali cellule, qualsiasi molecola la cui struttura o funzione fornisca una traccia della sua storia evolutiva rappresenta un fossile molecolare. Si tratta di un'astrazione più che di oggetti tangibili: tali molecole registrano, incorporano e riflettono un'antica tappa dell'evoluzione ma non sono antiche. I fossili molecolari sono essenziali per capire l'evoluzione molecolare perché non c'è nessun reperto fossile tangibile delle forme di vita più antiche. Così come le ossa, le conchiglie o le pareti cellulari mineralizzate ci dicono qualcosa sull'evoluzione degli odierni organismi pluricellulari, le molecole biologiche attuali ci forniscono indizi sull'evoluzione delle prime forme di vita. Un esempio particolarmente eclatante di fossile molecolare è l'RNA catalitico.
Riprodurre un brodo primordiale
Se l'idea di un mondo a RNA è stata come un'illuminazione per i biologi, essa ha costituito un fardello oneroso per i chimici, come sarà discusso anche nel prossimo paragrafo. La condizione sine qua non di un mondo a RNA è l'esistenza di un brodo primordiale di oligonucleotidi con una concentrazione di lunghe molecole di RNA (o simili all'RNA) sufficiente per la produzione casuale di un RNA che si autoreplichi, il suo incontro con una molecola complementare e l'inizio della moltiplicazione. Per i chimici, concepire un tale brodo è uno scoglio insormontabile. Nelle presumibili condizioni primordiali il ribosio è difficile da sintetizzare, è instabile ed è in grado di esistere in molte forme isomere (Larralde et al., 1995); le basi stesse non sono particolarmente facili da sintetizzare, sebbene si stiano facendo progressi in questo senso (Robertson e Miller, 1995); il legame glicosidico si può formare sia nella configurazione α sia in quella β, ed è labile nelle condizioni in cui, plausibilmente, si sarebbero dovuti formare il ribosio e le basi; la condensazione del ribosio con il fosfato deve avvenire sull'idrossile 3' o 5' del ribosio, non sul 2'; il fosfato deve essere attivato senza distruggere altri gruppi; e infine lo scheletro fosfoesterico deve essere costruito tra i gruppi idrossilici 3' e 5' senza coinvolgere l'idrossile 2'. Perciò, anche se fosse possibile ottenere sufficienti quantità di unità monomeriche di RNA, nucleosidi o nucleotidi, la polimerizzazione non sarebbe banale, perché l'RNA è una molecola a elevata chiralità e con molti isomeri. E la presenza di entrambe le forme enantiomeriche dei monomeri avrebbe certamente compromesso un eventuale processo di sintesi degli oligoribonucleotidi su stampo di natura non enzimatica che avrebbe potuto innescare nel brodo la prima reazione della vita.
Per tutti i concreti motivi sopra elencati, l'idea che l'RNA sia stato la prima molecola in grado di autoreplicarsi si è consolidata di più tra i biologi molecolari che tra i chimici. Ma c'è anche un'altra differenza fondamentale tra le discipline della biologia e della chimica che influenza profondamente la discussione sulle prime fasi dell'evoluzione. Ci sono due modi diversi di pensare all'origine della vita: si può ragionare a ritroso, partendo dai meccanismi molecolari odierni, o in progressione, partendo dai principi chimici di base. I chimici tendono a ragionare 'in avanti' presupponendo che la vita debba avere avuto origine secondo le regole della chimica come le conosciamo ora; i biologi tendono ad andare 'a ritroso' perché sono umilmente consci delle notevoli trasformazioni elaborate dall' evoluzione, e sono quindi più a loro agio nel considerare la biologia contemporanea come una memoria storica. L'abisso intellettuale tra l'ottica sintetica così comune tra i chimici e quella analitica dei biologi non è facile da colmare. In effetti, come sarà discusso più approfonditamente in seguito, i chimici hanno fatto una tale fatica a ottenere piccole molecole di RNA tentando di ricreare in laboratorio le presunte condizioni primordiali, che sono quasi arrivati a dichiarare per iscritto che la vita è impossibile (Joyce e Orgel, 1993; Ferris et al., 1996). I biologi cercano di non fare questo tipo di errore.
La prima molecola capace di replicarsi era costituita da RNA o da un quasi-RNA?
I chimici, cercando nel brodo chimico primordiale un primo barlume di replicazione biotica, si sono trovati in difficoltà per la scarsità di schemi interessanti che simulassero la sintesi prebiotica dell'RNA. Inoltre, si è dimostrato molto difficile ipotizzare meccanismi plausibili per la sintesi preferenziale, la stabilizzazione e la polimerizzazione di polimeri simili all'RNA con la necessaria chiralità. Questa difficoltà ha guidato alcuni chimici a una visione radicale, espressa con forza, seppure ironicamente da Joyce e Orgel (1993), secondo cui l'RNA è una molecola relativamente avanzata che deve essere stata preceduta da qualcosa di più semplice, qualcosa di simile all'RNA, ma un quasi-RNA. In particolare, il fenomeno di inibizione causato dagli enantiomeri ha portato a supporre che il primo materiale genetico fosse una molecola achirale (o protochirale), simile all'RNA, basata sul glicerolo aciclico e non sul ribosio, che sarebbe diventata chirale solo nella forma a doppio filamento. La proposta originale di un quasi-RNA è andata così oltre da abbandonare persino l'architettura attuale dell'ossatura dell'RNA, invocando un legame pirofosfato invece del semplice legame fosfodiesterico delle successive unità monomeriche basate sul glicerolo.
I biologi molecolari, guardando a ritroso da un mondo in cui l'RNA funziona sia come genoma sia come enzima, sono stati in difficoltà ancora maggiori per le complicazioni che impone ammettere l'esistenza di un materiale genetico che non fosse RNA. Non c'è alcun problema a immaginare che un quasi-RNA, come l'RNA, possa aver funzionato sia come genoma sia come catalizzatore, dando forse anche origine a un iniziale mondo di quasi-RNA. Il problema con il quasi-RNA è un problema di transizione: sarebbe stato molto difficile, se non impossibile, per un qualsiasi mondo di quasi-RNA trasformarsi in un vero mondo a RNA, che potesse in seguito dare origine al mondo a DNA che conosciamo oggi.
La conversione dal mondo di quasi-RNA al mondo a RNA pone problemi in termini sia chimici sia biologici. Poiché non ci sono tracce del quasi-RNA nei sistemi viventi contemporanei, la transizione da quasi-RNA a RNA deve essere stata essenzialmente continua, di modo che l'RNA abbia potuto assumere progressivamente tutte le funzioni. Per fare questo, l'RNA dovrebbe essere stato una copia perfetta del quasi-RNA, nella struttura come nella funzione. Tuttavia, elementi chiave della struttura dell'RNA sembrano dipendere specificamente e, forse, esclusivamente dalla struttura dell'unità monomerica. Per esempio, i tetraloop (anse di 4 nucleotidi) costituiscono una struttura particolarmente stabile, ben impilata, con legami idrogeno, formata da determinate anse di RNA a 4 nucleotidi come GNRA, UNCG e CUUG, dove N indica un nucleotide qualsiasi e R una purina (Woese et al., 1990). Questi tetraloop sono strutture così precise che è difficile pensare che qualsiasi altra molecola diversa dall'RNA possa adottare una struttura del genere (Allain e Varani, 1995). Allo stesso modo, benché l'assenza di un gruppo idrossile 2' non comprometta affatto la funzione dal punto di vista strutturale, dato che una sequenza di tRNA fatta di DNA non modificato può essere amminoacilata (Khan e Roe, 1988), l'idrossile 2' può essere indispensabile per altre funzioni come la formazione di tetraloop (Allain e Varani, 1995), il legame coll'RNA a doppio filamento (Strobel e Cech, 1995) e anche la catalisi (Abramovitz et al., 1996). Inoltre, i monomeri o i tratti di quasi-RNA inibirebbero quasi sicuramente un enzima a RNA, proprio come si ritiene che gli enantiomeri possano inibire la polimerizzazione non enzimatica (Joyce e Orgel, 1993). Perciò, anche se in teoria un quasi-RNA potrebbe anche essere esistito, esso sarebbe stato un ramo morto nell'evoluzione di un mondo a RNA (v. figura 1).
Per tutti questi motivi, siamo assolutamente favorevoli a ipotizzare che le prime molecole in grado di autoreplicarsi fossero costituite da RNA, come lo conosciamo oggi. Per fortuna, i chimici stanno iniziando a credere a un mondo a RNA, in quanto in nuove condizioni, su superfici minerali, la polimerizzazione di monomeri dell'RNA ha dato luogo alla formazione di polimeri più lunghi con il corretto legame fosfoesterico (Ferris et al., 1996).
I differenti ruoli dell'RNA
L'RNA come genoma
Anche prima che venissero scoperte le proprietà catalitiche dell'RNA, era ben conosciuta la sua capacità di funzionare come genoma, visto che in molti virus esso è costituito interamente da RNA. Gli studi su questi virus hanno fornito parte dello schema concettuale sui genomi a RNA del mondo a RNA. L'RNA è chimicamente in grado di funzionare come genoma, perché due filamenti complementari possono formare una struttura duplex antiparallela, essenzialmente con le stesse coppie di basi usate nel DNA. Sia nell'RNA sia nel DNA, la struttura complementare della doppia elica è perfettamente adatta alla funzione della replicazione. Ciascun filamento può servire da stampo per dirigere la polimerizzazione di una progenie di nuovi filamenti per appaiamento di basi complementari.
L'RNA come catalizzatore
Alcune proprietà chimiche dell'RNA mancano nel DNA. La differenza tra RNA e DNA è che negli anelli di desossiribosio del DNA mancano i gruppi idrossili 2'. Gli enzimi a RNA, come gli enzimi proteici, creano una superficie catalitica con il ripiegamento in strutture secondarie e terziarie complesse. L'assenza dei gruppi idrossili 2' rende il DNA meno adatto dell'RNA a ripiegarsi nelle complesse forme tridimensionali necessarie all'attività catalitica. Inoltre, in alcuni enzimi a RNA, l'idrossile 2' del ribosio svolge anche un ruolo catalitico diretto. Poiché il DNA manca di un idrossile 2', gli enzimi a DNA potrebbero non essere altrettanto versatili quanto gli enzimi a RNA. Alcune delle attività catalitiche dell'introne dell'rRNA di Tetrahymena sono senza dubbio dovute alla sua capacità di formare una struttura chimica complessa.
Dopo la scoperta della funzione catalitica dell'RNA, sono state formulate molte ipotesi su come l'RNA avesse potuto svolgere il compito della catalisi. Una scuola di pensiero sosteneva che gli enzimi a RNA si sarebbero rivelati molto simili agli enzimi proteici: il ripiegamento dell'ossatura dell 'RNA (così come quello di un polipeptide negli enzimi proteici convenzionali) creerebbe una superficie provvista di gruppi chimicamente attivi, precisamente localizzati, che potrebbero effettuare il processo di catalisi. Il problema principale di questa ipotesi era che nell 'RNA la varietà dei gruppi interessanti dal punto di vista chimico (quattro basi fortemente aromatiche e non particolarmente reattive) è estremamente limitata in confronto ai numerosi gruppi funzionali che hanno gli amminoacidi delle attuali proteine (imidazolici, sulfidrilici, idrossilici, indolici, fenolici, ammidici, amminici, guanidinici, carbossilici e tioeterici). L'altra scuola di pensiero, all'inizio più prudente, ma fondamentalmente più radicale, sosteneva che gli enzimi a RNA potevano funzionare con un meccanismo assolutamente nuovo. E così è effettivamente.
l cationi bivalenti si sono dimostrati la chiave dell'attività catalitica dell'RNA. Tutti gli enzimi a RNA noti si sono rivelati essere non ribozimi, ma metalloribozimi (Yarus, 1993). Il ruolo principale del ripiegamento dell' ossatura di RNA non è quello di esporre le basi, ma piuttosto di fornire una superficie che possa chelare (e quindi collocare con precisione) metalli bivalenti nel sito attivo. l cationi bivalenti chelati (in genere Mg²+, ma anche altri metalli) e l'acqua di idratazione che li circonda funzionano quindi come catalizzatori acido/base per estrarre un protone dal gruppo entrante e aggiungerlo a quello uscente. Anche se sarebbe prematuro concludere che tutti gli enzimi a RNA sono metalloenzimi (ciò potrebbe semplicemente riflettere il fatto che tutti i ribozimi esistenti in natura e finora scoperti catalizzano il trasferimento di legami fosfoesterici), è interessante notare che anche molti enzimi proteici che catalizzano i trasferimenti di legami fosfoesterici, e in particolare le polimerasi, sono metalloenzimi e usano i gruppi carbossilici per chelare e collocare con precisione i cationi bivalenti (Steitz e Steitz, 1993; Steitz et al., 1994). È importante osservare che il gruppo idrossile 2' in più che distingue il ribosio (nell'RNA) dal desossiribosio (nel DNA) costringe l'RNA a doppio filamento ad assumere una configurazione a elica di forma A, invece della configurazione di forma B tipica del DNA della maggior parte dei sistemi biologici. Come risultato gli RNA a doppio filamento sono più corti e più tozzi del DNA, le coppie di basi sono inclinate rispetto all'asse dell'elica, l'incremento della distanza per ogni coppia di basi risulta molto più piccolo e le proporzioni relative del solco maggiore (stretto, ma profondo) e di quello minore (largo, ma poco profondo) sono quasi l'inverso rispetto al DNA. Le notevoli differenze stereochimiche tra l'RNA a doppio filamento e i genomi a DNA complicano qualsiasi scenario nel quale i genomi di un mondo a RNA possano aver dato origine ai genomi a DNA; gli RNA o le proteine che riconoscono la doppia elica di RNA e agiscono su di essa non necessariamente sono in grado di riconoscere la doppia elica di DNA.
Gli organismi nel mondo a RNA
Dal momento che non esiste nessun reperto fossile del mondo a RNA, potremmo non avere speranza di conoscere quale genere di organismi vivesse in quell'era. Tuttavia, se si accetta la nozione che la biochimica degli organismi contemporanei contiene fossili molecolari che molto probabilmente risalgono all'inizio della vita sulla Terra, allora uno sguardo attento alla biologia molecolare degli organismi moderni può fornire qualche spunto illuminante su uno degli aspetti più affascinanti dell'evoluzione molecolare: come queste molecole primordiali effettuavano la replicazione.
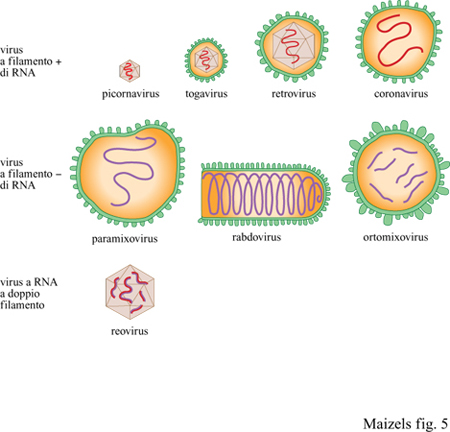
l genomi di nuclei, mitocondri e cloroplasti di tutte le cellule attuali sono costituiti da DNA; tuttavia nella straordinaria diversità dei virus contemporanei, ve ne sono molti che contengono genomi fatti di RNA. Vi sono virus a doppio o a singolo filamento di RNA, virus con filamenti di RNA di segno positivo (+) o di segno negativo (-), virus con RNA circolare o lineare (fig. 5). Vi sono virus a RNA che si replicano tramite intermedi di DNA, virus a DNA che si replicano tramite intermedi di RNA, ma anche virus costituiti sia da RNA sia da DNA. Parte di questa diversità dei genomi (che qualcuno potrebbe chiamare 'baraonda') potrebbe riflettere la varietà di strategie di replicazione presente prima che i grandi genomi a DNA divenissero la norma cellulare?
Virus semplici con genomi a RNA
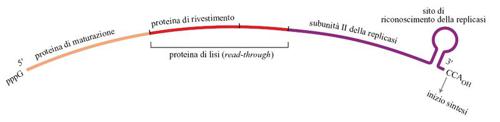
Alcuni dei virus a RNA più semplici infettano il batterio Escherichia coli; uno di questi, Qβ, è stato studiato approfonditamente. Il genoma di Qβ è una molecola di RNA lunga 4,2 kb ed è uno dei genomi più piccoli che si conoscano (fig. 6). All'estremità 3' del Qβ c'è una struttura tRNA-simile che termina con la sequenza CCA. La struttura tRNA-simile all'estremità del filamento (+) serve come elemento di riconoscimento per la replicasi, che inizia la sintesi del filamento (-) a livello del penultimo C della terminazione CCA. Così la struttura tRNA-simile assicura la specificità della replicazione. Il CCA 3' -terminale del Qβ può anche funzionare, almeno in linea di principio, come un moderno telomero; la perdita di parte o di tutta la sequenza CCA può essere ripristinata dall'enzima che aggiunge il CCA, la nucleotidiltransferasi del tRNA.
Virus con genomi molto simili infettano anche le piante. Dai nomi di questi virus risulta che vi sono inclusi alcuni terribili patogeni vegetali, come il virus delraggrinzimento delle rape o il bromovirus. Anche i genomi di questi virus delle piante recano all'estremità 3' strutture tRNA -simili. Addirittura esse possono essere riconosciute da specifiche amminoacil-tRNAsintetasi che legano alla loro estremità un amminoacido. In diversi casi esse vengono riconosciute e vagliate dalla RNasi P di E. coli. Pertanto non vi può essere alcun dubbio che queste strutture abbiano una reale somiglianza con i veri e propri tRNA. Piante e batteri appartengono a due regni distinti, Eucarioti e Eubatteri, che divergono molto presto nella storia evolutiva del mondo a DNA. Sebbene i virus possano avere, come i plasmidi (Sprague, 1991), la potenzialità di passare da un regno a un altro, l'esistenza di strutture genomiche conservate in virus che infettano due diversi regni rafforza l'ipotesi che questi virus possano essere altrettanto antichi, o ancora più vecchi, delle forme di vita cellulari. Questi virus possono quindi essere fatti risalire almeno ai tempi del progenitore di tutte le forme di vita, o a tempi addirittura precedenti.
Il ruolo del tRNA nella replicazione
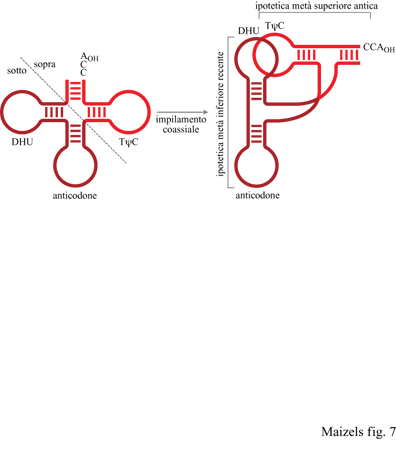
Le osservazioni sulla replicazione dei più semplici genomi a RNA contemporanei ci hanno permesso di ipotizzare che le prime strutture tRNA-simili abbiano avuto origine come marcatori genomici i quali contrassegnavano le estremità 3' degli antichi genomi a RNA per la replicazione da parte di enzimi a RNA in un mondo a RNA (Weiner e Maizels, 1987). I più semplici di tali marcatori sarebbero stati i predecessori della metà superiore del moderno tRNA, che consiste in un impilaggio coassiale del braccio TtC sullo stelo accettore (fig. 7). In quest'ottica, il tRNA dovrebbe essersi inizialmente evoluto per funzionare nella replicazione ma non nella traduzione, e solo successivamente sarebbe stato introdotto nell'apparato di traduzione. L'ansa dell'anticodone potrebbe essere stata un'aggiunta successiva sulle prime strutture tRNA-simili, avvenuta con lo sviluppo della sintesi proteica diretta da stampo.
L'evoluzione dei genomi a DNA
Genomi di transizione
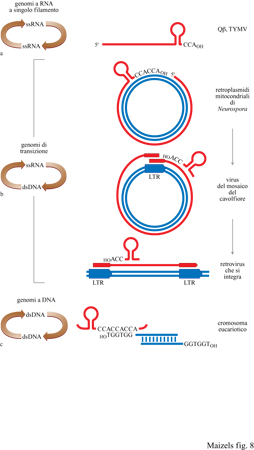
Se le strutture tRNA-simili funzionarono dapprima nella replicazione, ci dovremmo aspettare che queste strutture siano di importanza critica anche per la replicazione di genomi diversi dai semplici virus a RNA. Infatti le strategie di replicazione di numerosi genomi attuali, molto differenti tra loro, utilizzano strutture tRNA-simili. In ciascuno di questi casi, un RNA genomico si replica tramite un intermedio di DNA; li abbiamo chiamati genomi di transizione in quanto possono essere considerati come la riproduzione della transizione da un mondo a RNA all'attuale mondo a DNA (fig. 8). Una filogenesi di questi genomi basata sulla funzione del tRNA nella replicazione sembra legare la replicazione dei virus a singolo filamento di RNA con la sintesi dei moderni cromosomi (Maizels e Weiner, 1994).
Il plasmide Mauriceville dei mitocondri di Neurospora è l'esempio più simile ai semplici virus a RNA (Wang e Lambowitz, 1993): in uno stadio del suo ciclo vitale, questo plasmide è formato da un doppio filamento di DNA. Il DNA è replicato tramite il meccanismo di trascrizione a rolling circle per generare un RNA multirnerico che viene tagliato fino a produrre trascritti monomerici completi, ciascuno con una struttura tRNA -simile al 3' - terminale che finisce con un CCACCA. Questi RNA rappresentano l'altro stadio del ciclo di vita genomico. Gli RNA codificano una trascrittasi inversa, che copia l'RNA in un cDNA (DNA complementare), iniziando a livello marcatore genomico 3'-terminale. Il plasmide Mauriceville è dotato di un genoma a RNA o a DNA, a seconda della fase del suo ciclo: in questo senso esso può essere chiamato anche retroplasmide, in cui il plasmide a DNA svolge la stessa funzione del provirus nel ciclo della vita di un moderno retrovirus.
Il genoma del virus del mosaico del cavolfiore (CaMV) è un DNA circolare a doppia elica che si replica come elemento extra-cromosomico senza mai integrarsi nel DNA cromosomico. La trascrizione del genoma virale inizia a livello di una particolare regione regolatrice che, analogamente all'LTR (Long Terminai Repeat, lungo elemento ripetuto terminale) retrovirale, è costituita da un promotore della RNA-polimerasi II, posto subito a monte di un segnale di poliadenilazione. La trascrizione del genoma genera un trascritto ridondante in posizione terminale, con lunghezza leggermente maggiore di quella definitiva. Questo RNA genomico è poi convertito in cDNA da una trascrittasi inversa codificata da CaMV, utilizzando tRNA come primer. Questo è uno schema di replicazione molto simile a quello usato dai retrovirus, con la differenza che l'integrazione del DNA del retrovirus nel cromosoma ospite genera un provirus a DNA lineare con una copia della regione regolatrice a ogni estremità terminale, e che la trascrizione, a partire dal promotore del provirus a monte fino al segnale distale di poliadenilazione, genera l'RNA genomico con la zona ridondante nella parte terminale.
Questa filogenesi può essere estesa ai cromosomi cellulari. La telomerasi, l'enzima che replica le estremità degli attuali cromosomi, aggiunge sequenze ripetute TnGm, un nucleotide alla volta, a un determinato primer TnGm (Blackbum, 1992).
La telomerasi è una ribonucleoproteina e la sua componente di RNA serve da stampo interno per l'aggiunta delle sequenze da parte della subunità proteica.
Il meccanismo d'azione della telomerasi è analogo a quello della trascrittasi inversa del plasmide Mauriceville che addiziona il tratto finale ripetuto CCACCA al marcatore genomico tRNA-simile; in particolare, sia la trascrittasi inversa del retroplasmide sia la telomerasi possono iniziare a livello di una sequenza CCA interna, usando un primer di DNA (Wang e Lambowitz, 1993). Se le strutture primitive tRNA-simili fossero predecessori degli stampi incorporati delle telomerasi contemporanee, si spiegherebbe perché le sequenze degli attuali telomeri siano variazioni del motivo ripetuto CnAm.
Scenari semplici ma filogenesi complesse
È improbabile che ci sia stata una linea evolutiva continua di discendenza da un genoma a RNA a singolo filamento, tipo Qβ, fino a un attuale genoma a DNA a doppia elica. Date la natura modulare di molti virus e la frequenza di ricombinazione, l'evoluzione virale è complessa da ricostruire. L'abbondanza di pseudotipi virali prova che le proteine di rivestimento sono spesso intercambiabili. L'intercambiabilità di moduli virali con funzioni potenzialmente simili può causare disastri nel tentativo di sviluppare filogenesi virali basate su una singola molecola come la trascrittasi inversa (Xiong e Eickbush, 1990). Con la proposta di una filogenesi funzionale basata sulle strategie di replicazione, noi suggeriamo che queste strategie sono più durature dei moduli che le eseguono: i moduli possono essere cambiati o associati in nuove combinazioni, mentre la strategia di replicazione tende a mantenersi. L'analisi delle strategie di replicazione, sebbene non sia quantitativa, evita i rischi delle filogenesi basate sulle singole molecole.
Il batteriofago Qβ potrebbe essere una combinazione evolutiva recente, con una proteina di rivestimento proveniente da un virus, una replicasi da un altro e una struttura 3' -terminale tRNA-simile di origine ancora diversa, che interagisce con l'EF-Tu (Elongation Factor Tu) che già interagisce con la replicasi. La notevole diversità dei virus costituiti da RNA a singolo filamento potrebbe avere avuto origine esattamente in questo modo. Così componenti cellulari e altri virus rappresentano una riserva (o un repertorio) di parti: anche se Qβ dovesse scomparire, potrebbe essere inventato di nuovo a partire da queste parti di scorta. Se si accetta questo, viene di conseguenza che nessun singolo stadio della filogenesi funzionaIe delle strategie di replicazione dipende da una precisa continuità di una linea di discendenza dai primordi a oggi. Ricombinazione e creazione sono realtà in un mondo biologico, ma non fanno necessariamente scomparire tutte le relazioni evolutive.
L'evoluzione della sintesi proteica
La biochimica di tutte le cellule moderne è così simile che ci sono pochi dubbi che tutta la vita sulla Terra possa essere fatta risalire a un unico progenitore comune, il progenote (Woese, 1990). Inoltre, poiché l'apparato di traduzione è essenzialmente simile in tutti gli organismi viventi, questo progenote deve essere stato in grado di effettuare sintesi proteica su stampo, sostanzialmente in modo analogo al nostro. Come già discusso in dettaglio altrove (Weiner e Maizels, 1987), uno scenario plausibile per l'evoluzione dell'apparato di traduzione può essere costruito su un percorso che parte con l'inizio dell'evoluzione del tRNA. Questo scenario fornisce una spiegazione convincente del vantaggio selettivo portato da ciascun nuovo componente, man mano che si presentava: prima i tRNA, come etichette genomiche, che funzionavano nella replicazione; più tardi le tRNA-sintetasi, per caricare queste etichette con amminoacidi basici e facilitare così l'interazione con la riboreplicasi fortemente acida; quindi i primi ribosomi per sintetizzare polimeri misti con un meccanismo che era essenzialmente senza stampo; infine l 'RNA messaggero e il codice genetico.
Il mondo a RNA non potrebbe durare a lungo senza membrane e metabolismo
Qui ci siamo soffermati, come avrebbe fatto la maggior parte dei genetisti molecolari, su quello che Francis Crick ha chiamato in modo memorabile il "dogma centrale", cioè il flusso dell'informazione genetica dal DNA all'RNA alla proteina. Tuttavia, qualsiasi biochimico o biologo cellulare, e forse anche un brillante studente delle scuole superiori, potrebbe far rilevare che un genoma primitivo non avrebbe potuto esistere a lungo isolato, vagando libero in un enorme mare primordiale. Il problema della diluizione sarebbe semplicemente insolubile; i filamenti complementari non si sarebbero potuti incontrare e, a meno che la concentrazione chimica delle molecole dei precursori nel brodo primordiale non fosse stata davvero astronomica, la riboreplicasi non avrebbe potuto trovare i nucleotidi precursori con cui costruire nuovi genomi. Dunque qualunque protocellula degna di questo nome deve avere avuto un interno e un esterno, e i componenti di quella protocellula dovevano essere stati sintetizzati a partire da composti reperibili nelle immediate vicinanze. Sebbene i primissimi genomi possano aver vagato liberi, prima della comparsa delle membrane, presto o tardi si devono essere chiusi in compartimenti. Gli organismi primitivi risultanti hanno dovuto sviluppare un metabolismo per costruire composti biologicamente utili con quelli disponibili nel brodo primordiale, e i loro genomi sono divenuti le unità di selezione evolutiva.
Le origini della compartimentazione e del metabolismo sono questioni ancor più difficili da affrontare dell'origine dei genomi e della replicazione, perché è molto più difficile trovare e identificare i loro fossili molecolari. Qualche progresso è stato fatto con una simulazione della sintesi prebiotica di lipidi (de Graaf et al., 1995), ma il problema della compartimentazione è lontano dall'essere risolto, e non c'è alcuna indicazione se siano più antichi i lipidi legati con legame esterico, come negli Eubatteri ed Eucarioti, o quelli legati con legame eterico, come negli Archaea. Si è ampiamente concordi, comunque, nel considerare la struttura di molti attuali cofattori metabolici come fossili molecolari del metabolismo del mondo a RNA, secondo quanto proposto da H.E. White (1982). In alcuni di questi cofattori (NAD e FAD), un residuo di adenosina serve da 'manico' per un anello aromatico chimicamente re attivo simile a un nucleotide (nicotinamide o flavina). In altri cofattori (acilCoA e coenzima B12), il 'manico' di adenosina è attaccato a una struttura relativamente semplice ma chimicamente reattiva, costituita da amminoacidi e loro derivati. In tutti i casi, la scelta dell' adenosina come 'manico' fa davvero pensare che i primi enzimi a usare questi cofattori fossero ribozimi. Una volta entrati stabilmente a far parte del metabolismo cellulare intermedio, questi cofattori sarebbero poi stati conservati e utilizzati dagli enzimi proteici che si stavano evolvendo e che gradualmente sostituivano i ribozimi originari.
Possiamo perciò affermare con una certa sicurezza che un tempo il mondo a RNA possedeva un assortimento di ribozimi in grado di effettuare un metabolismo intermedio rudimentale (o forse anche complesso). Comunque, nessuno di questi ribozimi sembra essere sopravvissuto, forse perché le proteine sono di gran lunga migliori dell'RNA nello svolgimento della maggior parte delle reazioni metaboliche (Alberts, 1986). Al contrario, molti ribozimi che eseguono il trasferimento del legame fosfoesterico (in primo luogo il ribosoma e l'RNasi P) sono sopravvissuti, forse perché l'RNA è più efficiente delle proteine nello svolgere le mansioni chimiche cruciali della sintesi proteica. Possiamo sperare che il progresso ottenuto recentemente con l'uso del SELEX per selezionare ribozimi con attività metabolica possa creare (o ricreare) un metabolismo virtuale non molto dissimile da quello che esisteva approssimativamente 4 miliardi di anni fa.
Il codice genetico: l'ultima grande frontiera
Rimangono ancora grandi misteri sulle fasi iniziali dell' evoluzione: uno dei più stimolanti è l'origine della sintesi proteica diretta da molecole-stampo e del codice genetico. Tutti gli studenti di biologia molecolare hanno esaminato attentamente la tavola delle triplette del codice cercando di trovare il filo logico che li aiutasse a impararlo a memoria. l teorici sono stati ispirati dalla magia delle triplette, ma anche scoraggiati dalle idiosincrasie delle degenerazioni del codice. Nessuno sa quando o come avverrà il passo in avanti decisivo nella comprensione dell'origine del codice, ma è lecito supporre che la chiave, quasi certamente, risieda in una conoscenza ancor più dettagliata di quella che a prima vista sembra essere una piccola molecola senza pretese: il tRNA.
Ringraziamenti
Siamo riconoscenti al dott. Alessandro Senes per la gentile collaborazione offerta alla revisione delle bozze della traduzione italiana del manoscritto.
Bibliografia citata
ABRAMOVITZ, D.L., FRIEDMAN, R.A., FYLE, A.M. (1996) Catalytic role of 2'-hydroxyl groups within a group II intron active site. Science, 271, 1410-1413.
ALBERTS, B.M. (1986) The function of the hereditary materials: biological catalyses reflect the cell's evolutionary history. Amer. Zoologist, 26, 781-796.
ALLAIN, F.H., VARANI, G. (1995) Structure of the PI helix from group I self-splicing introns. J. Mol. Biol., 250, 333-353.
BLACKBURN, E.H. (1992) Telomerases. Ann. Rev. Biochem., 61, 113-129.
CECH, T.R., BASS, B. (1986) Biological catalysis by RNA. Ann. Rev. Biochem., 55, 599-629.
CECH, T.R., ZAUG, A.J., GRABOWSKI, P.I. (1981) In vitro splicing of the ribosomal RNA precursor of Tetrahymena: involvement of a guanosine nucleotide in the excision of the intervening sequence. CelI, 27, 487-496.
DE GRAAF, R.M., VISSCHER, J., SCHWARTZ, A.W. (1995) A plausibly prebiotic synthesis of phosphonic acids. Nature, 378, 474-477.
EKLAND, E.H., SZOSTAK, lW., BARTEL D.P. (1995) Structuraly complex and highly active RNA ligases derived from random RNA sequences. Science, 269, 364-370.
FERRIS, J.P., HILL, A.R.JR., Lm, R., ORGEL, L.E. (1996) Synthesis of longprebiotic oligomers onmineral surfaces. Nature, 381, 59-61.
GILBERT, W. (1986) The RNA world. Nature, 319, 618.
GUERRIER-TAKADA, C., GARDINER, K., MARSH, T., PACE, N., ALTMAN, S. (1983) The RNA moiety of ribonuclease P is the catalytic subunit of the enzyme. CelI, 35, 267-272.
JOYCE, G.F., ORGEL, L.E. (1993) Prospects for understanding the origin of the RNA world. In The RNA world, a c. di Gesteland R. F., Atkins J., New York, Cold Spring Harbor Press, pp. 1-25.
KHAN, A.S., ROE, B.A. (1988) Aminoacylation of synthetic DNAs corresponding to Escherichia coli phenylalanine and Iysine tRNAs. Science, 241, 74-79.
LARRALDE, R., ROBERTSON, M.P., MILLER, S.L. (1995) Rates of decomposition of ribose and other sugars: implications for chemical evolution. Proc. Nat. Acad. Sci. USA, 92, 8158-8160.
MAIZELS, N., WEINER, A.M. (1994) Phylogeny from function: evidence from the molecular fossil record that tRNA originated in replication, not translation. Proc. Nat. Acad. Sci. USA, 91, 6729-6734.
NOLLER H.F. (1993) Peptidyl transferase: protein, ribonucleoprotein, or RNA? J. Bacteriol., 175, 5297-5300.
ROBERTSON, M.P., MILLER, S.L. (1995) An efficient prebiotic synthesis of cytosine and uracil. Nature, 375, 772-774.
SPRAGUE, G.F.JR. (1991) Genetic exchange between kingdoms. Curr. Opin. Genet. Dev., l, 530-533.
STEITZ, T.A., SMERDON, S.I., JAGER, J., JOYCE, C.M., STEITZ, J.A. (1994) A unified polymerase mechanism for nonhomologous DNA and RNA polymerases. Science, 266, 2022-2025.
STEITZ, T.A., STEITZ, lA. (1993) A generaI two-metal-ion mechanism for catalytic RNA. Proc. Nat. Acad. Sci. USA, 90, 6498-6502.
STROBEL, S.A., CECH, T.R. (1995) Minor groove recognition of the conserved G.U pair at the Tetrahymena ribozyme reaction site. Science, 267, 675-679.
WANG, H., LAMBOWITZ, A.M. (1993) The Mauriceville plasmid reverse transcriptase can initiate cDNA synthesis de novo and may be related to reverse transcriptase and DNA polymerase progenitor. CelI, 75, 1071-1081.
WEINER, A.M., MAIZELS, N. (1987) tRNA-like structures tag the 3' ends of genomic RNA molecules for replication: implications for the origin of protein synthesis. Proc. Nat. Acad. Sci. USA, 84, 7383-7387.
WHITE, H. B., III (1982) Evolution of coenzimes and the origin of pyridine nucleotides. In The pyridine nucleotide coenzymes, a c. di Everse, J., Anderson, B., Yu, K.S., New York, Academic Press, pp. 1-17.
WOESE, C. (1990) Evolutionary questions: the 'progenote'. Science, 247, 789.
WOESE, C.R., KANDLER, O., WHEELIS, M.L. (1990) Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya. Proc. Nat. Acad. Sci. USA, 87, 4576-4579.
XIONG, Y., EICKBUSH, T.H. (1990) Origin and evolution of retroelements based upon their reverse transcriptase sequences. EMBO J., 9, 3353-3362.
YARUS, M. (1993) How many catalytic RNAs? lons and the Cheshire cat conjecture. FASEB J., 7, 31-39.
Bibliografia generale
GESTELAND, R.F., ATKINS, J.F., a c. di, The RNA world. New York, Cold Spring Harbor Press, 1993.
WATSON, J.D., HOPKINS, N., ROBERTS, J.W., STEITZ, J.A., WEINER, A.M. The origins of life. Molecular biology of the gene. 4a ed., cap. 28, In Menlo Park, Benjamin Cummings, 1987