Proteine. Degradazione delle proteine
Proteine. Degradazione delle proteine
Le cellule spesso passano da uno stato all'altro, sia in risposta a stimoli ambientali sia come parte di una via di sviluppo finemente regolata. Questi passaggi richiedono, generalmente, che venga rapidamente smantellata la rete di regolazione preesistente, e ciò dipende, nella maggior parte dei casi, dalla degradazione delle proteine. Tale processo deve essere altamente specifico, allo scopo di evitare che siano coinvolte proteine essenziali o che la degradazione avvenga in tempi inappropriati. Il turnover proteico selettivo offre numerosi vantaggi rispetto ad altri tipi di meccanismi di regolazione. Un primo vantaggio è la velocità: minore è l'emivita di una proteina, inferiore è il tempo necessario per raggiungere lo stato stazionario in seguito a cambiamenti nella sua velocità di sintesi. Un secondo vantaggio è l'irreversibilità: l'eliminazione di una proteina evita ogni possibilità che essa sia riattivata in modo inappropriato.
Queste caratteristiche aiutano a spiegare perché la degradazione selettiva delle proteine sia quasi sempre una componente dei meccanismi di regolazione che coinvolgono precisi controlli temporali. Alcuni esempi sono la progressione del ciclo cellulare, i ritmi circadiani e le varie vie di trasduzione del segnale che conducono alla specificazione del tipo cellulare, al controllo metabolico e all'embriogenesi. Il prezzo da pagare per questo rapido meccanismo di regolazione del livello intracellulare delle proteine è la quantità di energia relativamente alta che viene consumata, soprattutto per risintetizzare le proteine distrutte.
Uno studio iniziato alla fine degli anni Settanta del Novecento ha dimostrato che una singola via proteolitica estremamente complessa è responsabile delle fasi principali della degradazione selettiva delle proteine cellulari solubili: la via ubiquitina-proteasoma. Lo sviluppo di un metodo che permettesse di osservare, in estratti cellulari, la degradazione ATP-dipendente di specifici substrati proteici ha consentito di scoprire che una modificazione covalente del substrato dovuta al legame molecolare di un peptide chiamato 'ubiquitina', rappresenta un punto critico del processo di degradazione; è stato inoltre possibile determinare il meccanismo molecolare della marcatura delle proteine con ubiquitina (detta 'ubiquitinilazione') e della loro degradazione finale.
Sebbene i lisosomi nelle cellule animali e i vacuoli nelle cellule vegetali e nei lieviti siano importanti organuli degradativi, la maggior parte dei fenomeni di demolizione di proteine citoplasmatiche che avviene in essi è relativamente aspecifica. Il contatto con gli enzimi idrolitici all'interno dei lisosomi avviene, infatti, in seguito a processi autofagici, che 'inghiottono' porzioni casuali di citoplasma. Vi sono, ovviamente, molti altri enzimi proteolitici nelle cellule e sono in corso approfonditi studi su alcuni di essi, come le calpaine e le proteasi tipo ICE (Interleukin converting enzyme), cruciali per l'apoptosi. Si porrà qui l'accento sulla logica della via ubiquitina-proteasoma, utilizzando come esempi alcuni dei sistemi modello maggiormente sviluppati ed esaminando da vicino la natura complessa e a più stadi della proteolisi ubiquitina-dipendente.
L'enzima attivatore dell'ubiquitina (E1)
L'attivazione del gruppo α-carbossilico terminale dell'ubiquitina da parte di E1 è un passaggio obbligatorio in tutti i processi dipendenti dall'ubiquitinilazione. Nello stesso modo in cui l'ubiquitina è indispensabile per la vitalità della cellula, non è stato sorprendente apprendere che E1 è essenziale per l'attività dell'ubiquitina. Vi sono due geni trovati nel lievito che codificano due polipeptidi, piuttosto divergenti, di tipo simile a E1. Queste proteine condividono una sequenza di consenso per il legame a nucleotidi (ATP) e conservano un residuo di cisteina che si pensa possa formare un tioestere con l'ubiquitina.
L'enzima che coniuga l'ubiquitina (E2)
In presenza di ubiquitina e ATP, l'enzima E1 forma un tioestere con l'ubiquitina. Al contrario, le proteine E2 non formano legami tioestere con l'ubiquitina in assenza di E1. Avram Hershko e collaboratori hanno dimostrato (1983) che l'ubiquitina è trasferita da E1 a E2 mediante transtiolazione. Il frazionamento, sia di reticolociti di coniglio sia di estratti di lievito, ha rivelato la presenza di diverse isoforme di E2, codificate da una famiglia di geni correlati tra loro. Nel lievito si possono riconoscere tredici enzimi simili a E2 (chiamati anche Ubc). Mentre i dati genetici del lievito indicano che la presenza di enzimi E2 multipli può essere necessaria per la massima degradazione di un substrato, l'analisi dell'ubiquitinilazione in vitro ha suggerito che diversi enzimi E2 potrebbero, a volte, avere funzioni ridondanti nell'ubiquitinilazione di particolari substrati. Tre differenti enzimi E2, che per sequenza non sembrano appartenere tutti alla stessa sottofamiglia, sono ciascuno in grado di ubiquitinilare la proteina antioncogene p53 in vitro. È interessante l'esempio delle cicline, proteine di regolazione che svolgono un ruolo fondamentale nella progressione del ciclo cellulare. L'improvvisa scomparsa di alcune cicline in momenti precisi del ciclo è alla base di questa progressione. La loro rapida rimozione è garantita dall'ubiquitinilazione seguita da proteolisi nel proteasoma. Le cicline mitotiche possono essere ubiquitinilate, in vitro, da due diversi enzimi E2: Ubc4 o UBCx/E2-C. L'ubiquitinilazione operata da questi due enzimi è cineticamente simile, e dipende dal fatto che il substrato possiede intatto un dominio, detto 'dominio di distruzione della ciclina', che costituisce un elemento di sequenza necessario per la degradazione regolata dal ciclo cellulare.
La ligasi ubiquitina-proteina (E3)
Si ritiene generalmente che il componente del sistema di coniugazione dell'ubiquitina più direttamente coinvolto nel riconoscimento del substrato sia l'enzima E3 o ligasi E3. Gli enzimi E3 sono tuttavia i meno conosciuti tra i fattori coinvolti nel legame ubiquitina-proteina, e quelli di cui si è, finora, compreso meno il funzionamento. Innanzitutto, le ligasi E3 possono essere raggruppate in classi distinte, in termini di sequenza amminoacidica e, probabilmente, anche per quanto riguarda il meccanismo d'azione. Negli ultimi anni, la famiglia degli enzimi E3 conosciuti o 'sospetti' ha iniziato a espandersi in maniera significativa. La regione all'estremità carbossiterminale, composta di circa 350 residui amminoacidici, è conservata in almeno una dozzina di questi enzimi, in organismi che vanno dal lievito ai Mammiferi, ed è stata chiamata 'dominio hect' (Homologous to E6-AP carboxyl-terminus). Il dominio hect comprende un residuo di cisteina sempre conservato, che nel caso di E6-AP (uno degli enzimi E3 più caratterizzati) è essenziale per la formazione del tioestere-ubiquitina e per l'ubiquitinilazione del substrato. È stato dimostrato che anche altre proteine di questa classe formano tioesteri con l'ubiquitina e sono implicate nell'ubiquitinilazione di proteine specifiche. Si potrebbe scoprire che anche altre proteine, magari molto diverse da quelle prima descritte, possono facilitare l'ubiquitinilazione delle proteine ed essere dunque considerate fattori tipo E3, almeno fino a quando non sarà chiarito pienamente il funzionamento di E3.
3. I meccanismi di ubiquitinilazione delle proteine e la loro regolazione
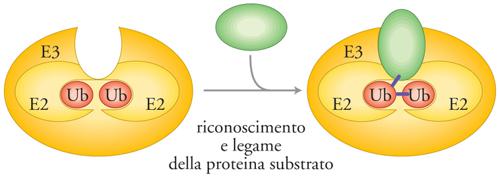
Mentre è ormai chiaro che per ubiquitinilare le proteine substrato sono necessari molti enzimi differenti, rimane ancora da chiarire come il substrato sia riconosciuto con precisione e come venga multiubiquitinilato. Potrebbe essere più appropriato considerare come fattore di riconoscimento l'intero complesso di ubiquitinilazione, che comprende le proteine tipo E2 ed E3 (e forse anche E1), piuttosto che le sole proteine E3 (fig. 3). Il complesso di ubiquitinilazione potrebbe assemblare una o più catene di ubiquitina direttamente sul substrato, mediante ripetute aggiunte di singole molecole di ubiquitina, oppure trasferendo una catena preassemblata sulla proteina bersaglio. Cosa distingue una proteina substrato per l'ubiquitinilazione da proteine che vengono raramente ubiquitinilate o che non lo sono mai? Questo è un quesito fondamentale, benché in gran parte irrisolto. Nella tab. 1 sono elencate alcune delle più importanti proteine la cui degradazione è soggetta a regolazione.
Gli esperimenti in corso stanno chiarendo che i substrati del sistema di degradazione mediato dall'ubiquitina contengono degli elementi di sequenza o 'segnali' che li indirizzano verso un rapido turnover e che questi segnali differiscono da substrato a substrato. L'analisi di substrati modello artificiali ha rivelato che alcuni segnali di degradazione contengono elementi separabili: uno che funziona come sito di legame per un fattore che riconosce il substrato e l'altro che serve da sito di legame per l'ubiquitina. L'ubiquitina viene quindi attaccata a residui di lisina, ma qual è l'elemento di riconoscimento che rende una lisina ubiquitinilabile? Sono in corso di definizione siti putativi di riconoscimento da parte degli enzimi ubiquitinilanti. Il primo esempio è stato la regione amminoterminale di substrati proteici artificiali. Gli studi su questi substrati hanno suggerito che l'amminoacido all'estremità amminoterminale determini il riconoscimento della proteina da parte di un enzima E3 (regola dell'amminoterminale).
La regolazione della degradazione ubiquitina-dipendente è di grande interesse. Vediamo, per esempio, cosa accade nelle cicline alle quali abbiamo già accennato. Molte cicline di tipo B sono degradate solo durante la mitosi e, almeno nel lievito, nella fase precoce G1 del ciclo cellulare. Una sequenza moderatamente conservata di nove residui, posta all'estremità amminoterminale, costituisce il segnale di degradazione, il cosiddetto 'dominio di distruzione'. È probabile che esistano dei controlli addizionali anche a livello del substrato stesso. Questo è suggerito dal fatto che la degradazione delle cicline di tipo A dipende dal dominio di distruzione, ma mostra nello stesso tempo una diversa dipendenza dal ciclo cellulare rispetto a quella delle cicline B. In base alle prove disponibili, sembra possibile che proteine contenenti domini di distruzione differenti siano il bersaglio di diversi fattori di ubiquitinilazione, i quali potrebbero o meno avere alcuni componenti comuni.
4. Le catene di ubiquitina
La poliubiquitinilazione del substrato proteolitico, di solito per aggiunta di uno o più oligomeri di ubiquitina, sembra stimolare la proteolisi anche se, almeno in vitro, la monoubiquitinilazione è chiaramente sufficiente per avere velocità apprezzabili di degradazione proteasomiale di alcune proteine. D'altra parte, alcune proteine dipendono chiaramente dalla poliubiquitinilazione per avere un rapido turnover in vitro. Sebbene non vi siano molti dati disponibili a questo riguardo, è stato anche suggerito che l'ubiquitina potrebbe innescare la denaturazione del substrato piuttosto che essere semplicemente un segnale passivo per il proteasoma. È probabile che le condizioni richieste per la denaturazione dipendente dall'ubiquitina siano variabili da substrato a substrato. L'ubiquitina potrebbe anche facilitare la denaturazione del substrato in maniera indiretta. Si ritiene che complessi regolatori (19S) all'interno del proteasoma 26S denaturino i substrati, forse mediante l'azione delle sei o più subunità ATPasiche trovate nel complesso, allo scopo di far penetrare la proteina all'interno del proteasoma 20S vero e proprio. Le catene di ubiquitina attaccate al substrato potrebbero servire da ancore per il proteasoma, per cui le proteine difficili da denaturare dovrebbero richiedere un'ubiquitinilazione maggiore. Molecole di ubiquitina sono, generalmente, legate insieme tramite legami isopeptidici tra una lisina in posizione 48 di un'ubiquitina e il gruppo carbossiterminale dell'altra ubiquitina. Recenti studi molecolari e genetici suggeriscono che si possano formare anche altri tipi di legame che coinvolgono, forse, forme varianti di ubiquitina. È possibile quindi pensare che l'aggiunta a una proteina di catene di ubiquitina alternative sia un segnale per destini metabolici diversi.
5. La deubiquitinilazione delle proteine
L'ubiquitina è rimossa dai substrati a opera di enzimi noti come 'enzimi deubiquitinilanti': idrolasi carbossiterminale dell'ubiquitina oppure isopeptidasi dell'ubiquitina. Questi enzimi appartengono a due distinte famiglie: un gruppo di proteine relativamente piccole, che potrebbero rimuovere preferenzialmente l'ubiquitina da peptidi e da piccoli addotti, e un gruppo di proteine più grandi, che generalmente rimuovono l'ubiquitina da una gamma di substrati proteici, almeno in vitro. Quest'ultima famiglia, la cosiddetta 'classe Ubp' (Ubiquitinspecific processing protease), è estremamente diversificata, ma tutti i membri contengono differenti sequenze di consenso brevi, i 'domini Cys e His', che probabilmente aiutano a formare il sito attivo dell'enzima. Cambiare la velocità di deubiquitinilazione del substrato vuol dire alterare la probabilità che un intermedio poliubiquitinilato venga riconosciuto dal proteasoma 26S. Il numero sorprendentemente alto di enzimi deubiquitinilanti che si sta via via scoprendo aumenta la probabilità che la velocità del turnover di una specifica proteina possa essere regolata da questi enzimi in modo differenziale. Questa regolazione implicherebbe che gli enzimi Ubp possiedano un considerevole grado di specificità per il substrato o che siano differentemente compartimentalizzati all'interno della cellula.
Mentre è ragionevole supporre che, per la maggior parte, gli enzimi deubiquitinilanti lavorino come regolatori negativi del sistema dell'ubiquitina, vi sono anche modalità in cui gli enzimi deubiquitinilanti potrebbero aumentare l'ubiquitinilazione delle proteine o la loro degradazione. Prima di tutto, l'ubiquitina è sempre sintetizzata sotto forma di precursore e richiede la rimozione di peptidi o amminoacidi all'estremità carbossiterminale. Gli enzimi deubiquitinilanti sono, quindi, necessari per generare monomeri di ubiquitina. In secondo luogo, l'ubiquitina attivata può velocemente formare addotti con nucleofili cellulari particolarmente abbondanti come, per esempio, le ammine o il glutatione. Un terzo aspetto importante è costituito dal fatto che le lunghe catene di ubiquitina sono spesso assemblate insieme a proteine intracellulari e, in alcuni casi, anche con proteine che non dovrebbero essere bersaglio del sistema di degradazione.
Si stanno acquisendo prove sui diversi enzimi deubiquitinilanti che indicano che, in vivo, il loro ruolo sia effettivamente quello di facilitare la degradazione ubiquitina-dipendente, impedendo l'eccessivo accumulo di oligomeri inibitori di ubiquitina. Altri dati, che dimostrano che gli enzimi deubiquitinilanti possono avere una funzione di regolazione, derivano da lavori recenti condotti su cellule di mammifero. È stato osservato che diverse proteine implicate nella genesi dei tumori o nel controllo della crescita sono enzimi deubiquitinilanti. È interessante inoltre considerare che, per quanto riguarda l'oncogene umano tre-2, sembra che il prodotto proteico responsabile dell'attività tumorigenica sia la forma inattiva di un enzima deubiquitinilante. Nel loro insieme, queste scoperte indicano che gli enzimi deubiquitinilanti giocano un ruolo importante nella regolazione della crescita e dello sviluppo degli organismi eucariotici.
6. I proteasomi 20S e 26S
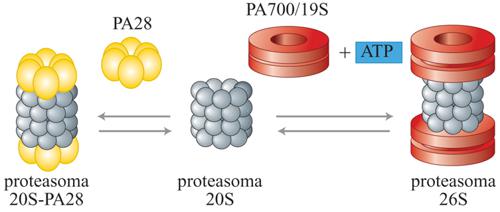
Il proteasoma 26S è un grosso complesso multimerico (fig. 4), composto da un core proteolitico, noto come 'proteasoma 20S', e da un paio di complessi regolatori 19S, disposti simmetricamente. Questi ultimi complessi sono strettamente imparentati con una proteina nota come PA700 (Proteasome activator di 700 kDa), anche se la loro esatta composizione e funzione e la loro precisa relazione con PA700 devono essere ancora chiarite. Quattro anelli, ciascuno composto da sette subunità, sono impilati in un cilindro a formare il proteasoma 20S. Queste subunità sono codificate da una famiglia di geni correlati ma distinti. La struttura del proteasoma 20S di Thermoplasma acidophilum, rivelata con risoluzione di 0,34 nm, ha permesso di far luce sul suo meccanismo d'azione. Dall'analisi della struttura del co-cristallo del proteasoma con un peptide inibitore, è stato possibile localizzare il sito attivo all'interno delle subunità β sulla superficie interna di una camera centrale del complesso del proteasoma. Le pareti del cilindro mostrano di non possedere larghe aperture, limitando così l'ingresso e l'uscita dei substrati e dei prodotti a una serie di canali che danno accesso alle due estremità del cilindro. Il proteasoma segrega quindi i suoi gruppi catalitici dal citoplasma circostante: caratteristica probabilmente vantaggiosa per una proteasi intracellulare.
Il riconoscimento di questi gruppi catalitici nella struttura tridimensionale del proteasoma ha rivelato un'altra sorpresa: l'attività catalitica del proteasoma è basata sulla presenza di un residuo dell'amminoacido treonina nel sito attivo; quindi, il proteasoma è una proteasi a treonina, la prima descritta finora. Una distinzione significativa tra il proteasoma di T. acidophilum e il proteasoma 20S degli eucarioti è che l'enzima procariotico ha un singolo sito attivo, mentre il più complesso proteasoma eucariotico ne ha almeno tre. Non è immediatamente comprensibile perché il proteasoma eucariotico 20S debba avere tante distinte subunità né è evidente quale sia la loro relazione con i diversi tipi di siti catalitici. Tuttavia, dato che il proteasoma 20S eucariotico deve associarsi con molti tipi distinti di fattori di regolazione alle due estremità del cilindro proteasomico (fig. 4), si può supporre che un'ulteriore variabilità nelle subunità α possa portare a un più ampio spettro di capacità di legame di fattori di regolazione. La presenza di molteplici subunità β, d'altro canto, potrebbe permettere sia una fine regolazione delle specificità di taglio di proteine (cleavage), sia una più efficiente degradazione di uno spettro più ampio di substrati. Ad aggiungere ulteriore complessità al rompicapo costituito dall'eterogeneità delle subunità del proteasoma 20S è intervenuta la scoperta che nei Mammiferi coesistono, all'interno di una stessa cellula, popolazioni proteasomiali composte da subunità tra loro diverse.
7. Il ruolo del proteasoma nella degradazione delle proteine
Denaturazione delle proteine come prerequisito per la loro degradazione
A differenza del proteasoma 26S, il proteasoma 20S non è capace di degradare proteine che siano in una conformazione tridimensionale stabile, ma può frammentarle completamente, in assenza di ATP, se sono preventivamente denaturate. Pertanto una possibile funzione della particella 19S/PA700 sarebbe quella di fungere da 'srotolatore' di proteine. Una proteina chaperon dipendente dall'ATP può quindi alterare lo stato conformazionale di una proteina substrato in modo tale da cambiarne l'attività, oppure può, se è accoppiata a un enzima proteolitico, causarne la distruzione. Almeno sei delle subunità PA700 risultano essere membri di una famiglia molto conservata ad attività ATPasica ed è noto che anche PA700, in toto, possiede un'attività ATPasica. Il numero delle diverse ATPasi di PA700 è sorprendente. Esse potrebbero fornire siti di legame alternativi, che favoriscono il legame e quindi la denaturazione di particolari proteine, o potrebbero avere funzioni alternative rispetto alla denaturazione, per esempio la traslocazione del substrato dai siti di legame di PA700 verso l'interno del proteasoma.
Rilascio dell'ubiquitina dalle proteine ubiquitinilate
Solitamente l'ubiquitina è riciclata dopo aver indirizzato la proteina ubiquitinilata al proteasoma e ciò indica che il proteasoma contiene, o può reclutare, enzimi deubiquitinilanti. Un quesito importante è relativo al momento in cui l'ubiquitina viene rilasciata dal substrato nel ciclo degradativo. Un meccanismo che rimuova l'ubiquitina solo dopo che la maggior parte del substrato è stata frammentata in peptidi avrebbe il vantaggio di impedire che peptidi non completamente digeriti si dissocino prematuramente dal proteasoma. Un possibile svantaggio di questo meccanismo sarebbe la complicanza topologica dell'esistenza di parti della proteina legate ai complessi 19S, mentre la proteina stessa viene denaturata e inviata al core del proteasoma. Il punto in cui un substrato è distaccato dalla sua catena (o catene) di ubiquitina potrebbe anche essere substrato-dipendente. Per esempio, il rilascio precoce potrebbe permettere a una proteina parzialmente degradata di essere rilasciata. Al momento vi sono poche prove che sostengono questa idea.
Degradazione delle proteine indipendente dall'ubiquitina
Un'eccezione alla generale importanza dell'ubiquitina nella degradazione a opera del proteasoma è data da un enzima di mammifero, l'ornitinadecarbossilasi (ODC), la cui degradazione (tab. 1) mediante proteasoma è ubiquitina-indipendente. Per essere degradata, ODC deve associarsi a un'altra proteina, l'antizima, che la indirizza al proteasoma 26S perché sia proteolizzata in modo ATP-dipendente. Non è ancora chiaro come l'azione dell'antizima possa essere analoga a quella dell'ubiquitina. Entrambe queste specie sembrano facilitare l'interazione tra le proteine a cui sono legate e il proteasoma 26S, o fornendo siti di legame per il proteasoma o alterando le proteine a loro associate, in modo da promuovere il legame al proteasoma, oppure in entrambi i modi.
8. Le altre funzioni cellulari dell'ubiquitina
Recentemente sono stati descritti diversi nuovi meccanismi di regolazione cellulare che coinvolgono l'ubiquitina. In molti casi è stato dimostrato che l'ubiquitinilazione funziona indipendentemente dall'azione del proteasoma, confermando un ruolo di primo piano per l'ubiquitina nella modificazione della funzione delle proteine, come da lungo tempo si sospettava. Un altro importante studio ha coinvolto il proteasoma nella degradazione di una proteina indirizzata, nelle fasi iniziali del processo, al reticolo endoplasmatico, apparentemente in seguito alla fuoriuscita della proteina dal reticolo stesso. Queste stimolanti scoperte suggeriscono che le nostre conoscenze sui processi cellulari regolati dal sistema dell'ubiquitina continueranno a espandersi in direzioni che ancora non possiamo prevedere.
L'endocitosi ubiquitina-dipendente di proteine della superficie cellulare
Molte proteine della superficie cellulare sono rapidamente internalizzate per endocitosi e, se non riciclate dalla membrana plasmatica, indirizzate ai vacuoli e ai lisosomi, dove sono distrutte da proteasi lisosomiali e vacuolari. Sembra molto probabile che l'ubiquitinilazione sia usata come segnale per l'endocitosi e come guida verso i vacuoli di molte proteine di membrana. Tuttavia, molti quesiti interessanti non sono stati ancora risolti. Prima di tutto, come si arriva dall'ubiquitinilazione delle proteine di membrana alla loro endocitosi? L'ubiquitina (o le catene di ubiquitina) potrebbe fornire un sito di legame per qualche componente del sistema endocitotico, oppure potrebbe alterare la struttura delle proteine in modo da facilitare questo legame. In alternativa, l'ubiquitina potrebbe promuovere il movimento delle proteine in regioni della membrana che compiono attivamente l'endocitosi. Un'altra questione è la ragione per la quale queste proteine ubiquitinilate non sono indirizzate al proteasoma. Cosa determini il destino metabolico alternativo delle proteine cellulari ubiquitinilate è, chiaramente, un quesito di considerevole importanza, a cui studi futuri dovranno dare una risposta.
La via di trasduzione del segnale di NFκB
Il fattore di trascrizione NFκB o membri correlati della famiglia di proteine Rel vengono attivati a seguito di una notevole varietà di segnali extracellulari. In condizioni non indotte, NFκB e i suoi simili risiedono nel citoplasma in un complesso eteromerico inattivo. NFκB è reso inattivo o perché le sue subunità sono legate a un membro della famiglia IκB o perché una delle sue subunità è in forma di un precursore (p105) che previene la traslocazione del complesso nel nucleo: entrambe funzionano nascondendo il segnale di localizzazione nucleare di NFκB. Questo è attivato o dalla maturazione proteolitica di p105 da parte della via ubiquitina-proteasoma, che porta alla rimozione dell'estremità carbossiterminale inibitoria, oppure dalla distruzione di IκB da parte della stessa via proteolitica (fig. 5). La degradazione di IκB, in vivo, richiede la fosforilazione di due serine vicine all'estremità amminoterminale che, a sua volta, innesca la poliubiquitinilazione del residuo di lisina adiacente e quindi la successiva degradazione di IκB. È stata descritta una proteina-chinasi con l'appropriata specificità verso queste serine critiche all'estremità amminoterminale. È interessante osservare che la fosforilazione di IκB è dipendente dall'ubiquitinilazione della chinasi o di un fattore regolatore che copurifica con la chinasi.
Inizialmente la degradazione delle proteine dipendente dall'ubiquitina era stata descritta sulla base di studi biochimici, ma ora la nostra conoscenza di questo sistema si è arricchita, grazie a un notevole numero di lavori sulla genetica e sulle strutture. Nonostante ciò, non siamo ancora in grado di rispondere a molte domande chiave in questo campo: qual è il segnale di degradazione proteica? Quali meccanismi regolano l'ubiquitinilazione e la degradazione delle proteine? Quali meccanismi di regolazione cellulare includono elementi del sistema dell'ubiquitina? Come sono regolate le dinamiche di addizione dell'ubiquitina al substrato e di rilascio da esso? Quali sono le funzioni delle diverse forme del proteasoma? Come è frammentata la proteina in piccoli peptidi e come viene regolato questo processo? Gli studi del sistema dell'ubiquitina portano sempre più a scoperte stimolanti e sorprendenti. Grazie alla sua fondamentale importanza biologica, la ricerca medica e biotecnologica su questo sistema è notevolmente aumentata. Molto vi è, però, ancora da scoprire prima di poter affermare di avere un'accettabile comprensione di questo meccanismo di modificazione e metabolismo proteico e di come esso funzioni nei circuiti regolatori delle cellule eucariotiche.
Bibliografia
Alberts 1994: Alberts, Bruce e altri, Molecular biology of the cell, 3. ed., New York, Garland, 1994.
Chen 1996: Chen, Zhijian J. - Parent, Lana - Maniatis, Tom, Site-specific phosphorylation of IkBalpha by a novel ubiquination-dependent protein kinase activity, "Cell", 84, 1996, pp. 853-862.
Deshaies 1995: Deshaies, Raymond J., The self-destructive personality of a cell cycle in transition, "Current opinion in cell biology", 7, 1995, pp. 781-789.
Gottesman, Maurizi 1992: Gottesman, Susan - Maurizi, Michael R., Regulation by proteolysis: energy-dependent proteases and their targets, "Microbiology and molecular biology reviews", 56, 1992, pp. 592-621.
Hershko 1983: Hershko, Avram e altri, Components of ubiquitin-protein ligase system. Resolution, affinity purification, and role in protein breakdown, "Journal of biological chemistry", 258, 1983, pp. 8206-8214.
Hershko, Ciechanover 1992: Hershko, Avram - Ciechanover, Aaron, The ubiquitin system for protein degradation, "Annual review of biochemistry", 61, 1992, pp. 761-807.
Hochstrasser 1996: Hochstrasser, Mark, Ubiquitin-dependent protein degradation, "Annual review of genetics", 30, 1996, pp. 405-439.
Huibregtse 1995: Huibregtse, John M. e altri, A family of proteins structurally and functionally related to the E6-AP ubiquitin-protein ligase, "Proceedings of the National Academy Sciences USA", 92, 1995, pp. 2563-2567.
Murakami 1992: Murakami, Yasuko, Ornithine decarboxylase is degraded by the 26S proteasome without ubiquitination, "Nature", 360, 1992, pp. 597-599.
Orlowski 1990: Orlowski, Mark, The multicatalytic proteinase complex, a major extralysosomal proteolytic system, "Biochemistry", 29, 1990, pp. 10289-10297.
Papa, Hochstrasser 1993: Papa, Feroz R. - Hochstrasser, Mark, The yeast DOA4 gene encodes a deubiquitinating enzyme related to a product of the human tre-2 oncogene, "Nature", 366, 1993, pp. 313-319.
Pickart 1988: Pickart, Cecile M., Ubiquitin activation and ligation, in: Ubiquitin, edited by Martin Rechsteiner, New York, Plenum, 1988, pp. 77-99.
Rubin, Finley 1995: Rubin, David M. - Finley, Daniel, Proteolysis. The proteasome: a protein-degrading organelle?, "Current biology", 5, 1995, pp. 854-858.
Thanos, Maniatis 1995: Thanos, Dimitris - Maniatis, Tom, NF-kB: a lesson in family values, "Cell", 80, 1995, pp. 529-532.
Varshavsky 1992: Varshavsky, Alexander, The N-end rule, "Cell", 69, 1992, pp. 725-735.
Wickner 1994: Wickner e altri, A molecular chaperon, ClpA, functions like DnaK and DnaJ, "Proceedings of the National Academy of Sciences USA", 91, 1994, pp. 12218-12222.