Trasposoni
Trasposoni
I trasposoni, o elementi genetici trasponibili, sono sequenze di DNA definite 'mobili' in quanto capaci di traslocare ('trasporre') da una localizzazione genica a un'altra senza apparentemente rispettare regole predeterminate. Vi sono trasposoni in una vastissima varietà di organismi, da quelli unicellulari (batteri e lieviti) fino all'uomo. La trasposizione può avvenire da un sito a un altro di uno stesso cromosoma, oppure da un cromosoma a un altro (in organismi eucariotici il cui genoma è organizzato in più cromosomi), o ancora da un sito cromosomico a un sito plasmidico (in organismi contenenti plasmidi, elementi genetici indipendenti dal cromosoma e capaci di replicazione autonoma), o infine dal cromosoma di una cellula a quello di un'altra.
La precoce osservazione pubblicata negli anni Quaranta del XX sec. a opera di Barbara McClintock, dalla quale risultava che gli elementi regolativi responsabili dell'espressione di alcuni geni sembrano spostarsi da un sito all'altro del genoma di mais, poté essere consolidata e sviluppata soltanto negli anni Settanta, quando la disponibilità di nuove tecniche di manipolazione del DNA consentì l'accumulo di numerose informazioni sulla presenza di trasposoni prima nei batteri e poi anche in organismi più complessi. Tali scoperte rendevano necessario rivedere almeno in parte uno dei concetti fondamentali della biologia, secondo il quale l'organizzazione dei cromosomi è molto stabile e i geni mantengono rigorosamente la loro posizione di generazione in generazione.
Al contrario, la diffusa presenza di trasposoni ‒ pur senza sconvolgere in poche generazioni l'organizzazione dei geni nei cromosomi, data la relativamente bassa frequenza di trasposizione (10−3÷10−7) ‒ fornisce un sostanziale contributo alla variabilità genetica e al trasferimento di materiale ereditario fra individui della stessa specie o di specie diverse, causando una diversificazione graduale dei genomi che nei tempi lunghi molto probabilmente modifica in maniera sostanziale il corredo genetico degli esseri viventi. Difatti gli effetti della trasposizione vanno dalla traslocazione di geni o gruppi di geni all'interruzione di altri geni per inserzione in essi di sequenze estranee, alla duplicazione di regioni anche grandi di DNA o alla loro inversione rispetto alla polarità del cromosoma. Tutti questi effetti, a loro volta, causano variazioni notevoli nelle proprietà della cellula, che vanno dalla perdita o dall'acquisizione di una o più funzioni fino alla morte della cellula stessa.
Nei batteri gli elementi trasponibili hanno dimensioni che variano da meno di 1000 a oltre 20.000 coppie di nucleotidi. La trasposizione avviene con un meccanismo di ricombinazione del DNA del trasposone con quello del sito in cui esso traspone detto, che viene detto 'ricombinazione sito-specifica'. Il trasposone si può inserire soltanto in regioni di DNA in cui si trovano brevi sequenze nucleotidiche che fanno da bersaglio per l'inserzione ('siti di inserzione'). La sequenza nucleotidica di tali siti sembra irrilevante e non omologa (cioè non somigliante) ad alcuna specifica sequenza del trasposone. Non occorre quindi affinità tra sequenze del trasposone e sito di inserzione, come invece nella ricombinazione omologa, che avviene tra geni aventi le stesse sequenze nucleotidiche.
Sequenze IS e trasposoni
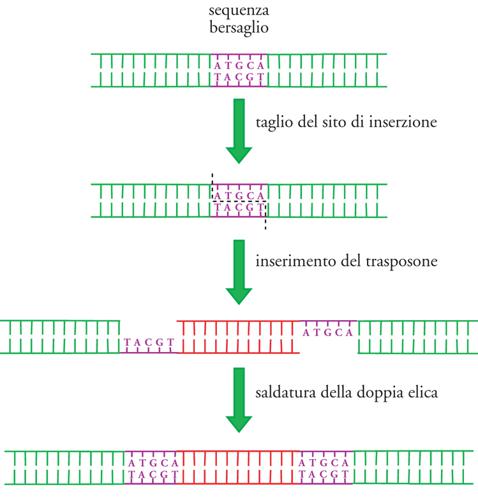
I più semplici elementi trasponibili sono le cosiddette 'sequenze IS' (Insertion sequences), presenti nei batteri. Si tratta di elementi genetici di piccole dimensioni, normalmente dalle 700 alle 1500 coppie di nucleotidi, caratterizzati dalla presenza alle loro estremità di brevi sequenze (generalmente intorno alle 10÷40 coppie di nucleotidi) uguali e disposte con orientamento opposto ('sequenze ripetute invertite'). L'unica funzione genica codificata dalle IS è quella relativa alla trasposasi, l'enzima che dirige il processo di trasposizione. I siti in cui avviene la ricombinazione sito-specifica delle IS non hanno sequenze ben definite e possono essere diversi anche per una stessa IS. Generalmente si tratta di brevi sequenze che vanno dalle 5 alle 10-12 coppie di nucleotidi. All'atto della trasposizione il sito di inserzione viene tagliato, lasciando a ognuna delle due estremità prodotte un breve tratto a singolo filamento che, a completamento dell'inserzione, verrà riempito con le basi complementari, creando pertanto due sequenze ripetute dirette ai due lati della IS (fig. 2). La presenza in un genoma batterico, a una distanza intorno alle 1000 coppie di nucleotidi, di due sequenze ripetute invertite, fiancheggiate da due brevi sequenze ripetute dirette, è un significativo segnale che in quel sito c'è una IS. Ogni IS è presente nelle cellule in un numero ben definito di copie, intorno alle 5÷10; tuttavia, in ogni organismo vi possono essere numerose IS diverse, designate con i simboli IS1, IS2, IS3 e così via.
Sono più grandi i trasposoni veri e propri, frammenti di DNA indicati con la sigla Tn, che possono avere dimensioni anche di oltre 20.000 coppie di nucleotidi (per es., le 24.000 di Tn4). In aggiunta al gene per la trasposasi, indispensabile per dirigere il processo di trasposizione, i trasposoni contengono uno o più geni che conferiscono alle cellule resistenze ad antibiotici o ad altre sostanze tossiche per i batteri. Tn1, Tn2 e Tn3 (circa 5000 coppie di nucleotidi ciascuno) contengono geni per la sola resistenza all'ampicillina, mentre Tn7 (14.000 coppie di nucleotidi) conferisce resistenza a tre antibiotici e Tn2603 a quattro antibiotici e al mercurio.
Le sequenze ripetute invertite presenti alle estremità degli elementi trasponibili possono in alcuni casi dar luogo, oltre che alla ricombinazione sito-specifica tipica della trasposizione, anche a ricombinazione omologa con uguali sequenze nucleotidiche situate in altre regioni dello stesso cromosoma o in un plasmide. Un esempio di tale fenomeno viene fornito dal fattore di fertilità F di Escherichia coli. Si tratta di un plasmide coniugativo, cioè che può essere trasferito da cellule che lo posseggono (definite 'cellule F+') a cellule che ne sono prive ('cellule F−') mediante la coniugazione, un particolare tipo di processo sessuale che avviene nei microrganismi. Occasionalmente, con frequenza di 1/106, il fattore F si integra nel cromosoma, diventando parte di esso. Tale integrazione avviene per ricombinazione omologa tra IS presenti nel fattore F e identiche sequenze presenti in varie copie lungo il cromosoma batterico.
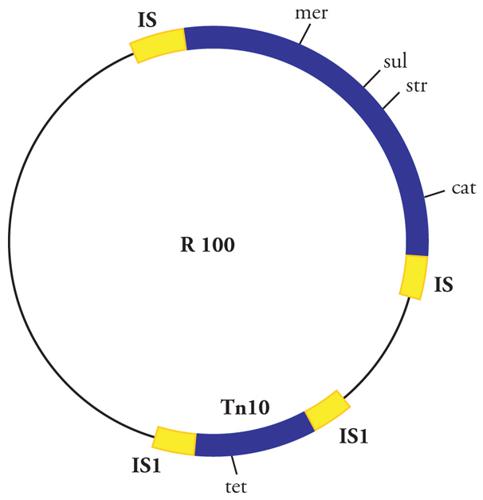
Numerosi trasposoni fanno parte di plasmidi (come il plasmide R100 rappresentato nella fig. 3). Alcuni plasmidi possono trasferirsi da una cellula all'altra mediante coniugazione (plasmidi coniugativi), veicolando con sé anche i trasposoni che contengono. Questi aggiungeranno quindi la mobilità plasmidica alla propria, incrementando in tal modo il proprio potere di trasferire da un organismo a un altro le funzioni che sono loro associate, quali per esempio le resistenze ad antibiotici. Ciò, se in tempi lunghi contribuisce alla variabilità genetica, che è notevolmente importante dal punto di visto evolutivo, rappresenta tuttavia un problema pratico, perché la diffusione rapida di resistenze agli antibiotici fra organismi diversi causa un incremento delle popolazioni microbiche resistenti, con la conseguente riduzione dell'efficacia delle terapie antibiotiche che vengono utilizzate in medicina.
Trasposoni composti e trasposoni complessi
I trasposoni batterici appartengono a due classi: trasposoni composti (detti anche di 'classe I') e trasposoni complessi (di 'classe II').
Un trasposone di classe I contiene a ognuna delle due estremità una copia di una sequenza IS. Fra di esse è situato un gene che codifica per la resistenza a un antibiotico. Normalmente, benché non sempre, le due IS terminali sono identiche e possono essere disposte con lo stesso orientamento oppure con orientamento opposto. La presenza delle due IS terminali, caratterizzate da sequenze ripetute e invertite ad ambedue le estremità, fornisce anche due trasposasi, ognuna delle quali è potenzialmente in grado di dirigere la ricombinazione sito-specifica. In alcuni casi (per es., Tn903, contenente due IS identiche alle estremità) ognuna delle due può promuovere la trasposizione, mentre in altri soltanto una di esse è attiva (per es., Tn5, che possiede a destra e a sinistra due IS identiche, delle quali solo quella di sinistra è tuttavia in grado di promuovere la trasposizione). Le IS terminali di un trasposone composto possono anche trasporre solamente sé stesse.
Un trasposone di classe II possiede alle sue estremità, anziché vere e proprie sequenze IS, due sequenze uguali di 30÷40 coppie di nucleotidi ciascuna, disposte con orientamento opposto. Fra queste due sequenze ripetute invertite si trovano il gene per la trasposasi e almeno un gene per la resistenza a un antibiotico. Il Tn3, che è il più studiato fra i trasposoni di classe II, codifica anche per una proteina regolatrice, la quale, mediante repressione, controlla la sintesi della trasposasi, evitandone in tal modo una produzione eccessiva, che non sarebbe utile per un evento quale la trasposizione che avviene con bassa frequenza.
Tipi di trasposizione
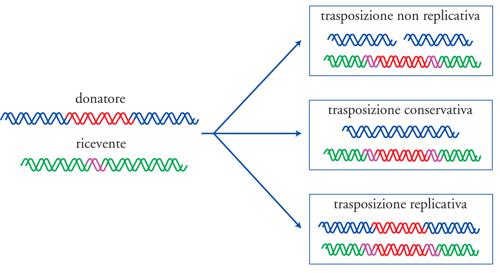
La trasposizione può essere replicativa o non replicativa. Per semplificare il discorso, immaginiamo che il sito di inserzione si trovi sul cromosoma e il trasposone su un plasmide. Nella trasposizione non replicativa si verifica un taglio della doppia elica al sito originale prima che il trasposone traslochi. Si otterrà quindi un'unica copia del trasposone nel sito di inserzione, mentre il plasmide, trasformato in una molecola di DNA non circolare, sarà perso, in quanto incapace di replicarsi nella cellula in forma lineare, salvo il caso in cui la doppia elica sia subito richiusa da una ligasi (enzima che catalizza la saldatura di molecole di DNA).
In alcuni casi il trasferimento avviene invece senza taglio della doppia elica al sito di origine (si parla di 'trasposizione conservativa'), in maniera analoga ai meccanismi con cui il DNA di virus quali il batteriofago λ si integra nel cromosoma batterico (fenomeno detto lisogenia). Nella trasposizione replicativa, come conseguenza della replicazione si avrà una copia del trasposone nel sito di inserzione e un'altra nel sito di origine, senza alcun taglio della doppia elica. Tale processo porterà alla formazione di un intermedio denominato 'cointegrato', nel quale il plasmide è rimasto temporaneamente inserito nel cromosoma. A tal punto interviene un enzima chiamato 'resolvasi' che, mediante ricombinazione omologa fra i due trasposoni, separerà il plasmide dal cromosoma. La fig. 4 illustra schematicamente i tre tipi di trasposizione descritti. Mentre alcuni trasposoni si trasferiscono mediante un solo tipo di meccanismo (per es., Tn5 e Tn10 si trasferiscono per trasposizione non replicativa, mentre la famiglia di trasposoni TnA attua il meccanismo replicativo), altri, quali alcuni elementi IS, usano sia la trasposizione replicativa sia quella non replicativa.
Il fago Mu, un trasposone particolare
Un esempio particolare di trasposone è il fago Mu, un virus specifico per Escherichia coli appartenente alla classe dei batteriofagi temperati, in quanto può svolgere all'interno della cellula un ciclo sia litico, duplicandosi numerose volte all'interno della cellula fino a distruggerla, sia lisogenico, ricombinando il proprio genoma all'interno del cromosoma batterico. La lisogenizzazione del fago Mu avviene mediante trasposizione non replicativa. Il ciclo litico si verifica invece per ripetute trasposizioni replicative. Diversamente dagli altri trasposoni, il fago Mu non possiede sequenze ripetute alle estremità, ma sequenze batteriche di diversa natura e lunghezza.
Trasposoni coniugativi
Alcuni trasposoni, detti 'coniugativi', possono passare da una cellula all'altra, anche di batteri di generi diversi, mediante coniugazione. In ciò essi ricordano i plasmidi, i quali tuttavia sono in grado di replicarsi autonomamente nella cellula, mentre i trasposoni possono essere trasferiti alla progenie solo se inseriti in elementi genetici capaci di replicazione autonoma all'interno della cellula, quali un cromosoma o un plasmide. Tali elementi sono stati individuati principalmente negli streptococchi, nei quali il trasferimento avviene con frequenze molto basse (ca. 10-8). Un elemento di questo genere è Tn916, un trasposone di circa 16.000 coppie di nucleotidi, che si trasferisce mediante trasposizione conservativa.
Trasposoni eucariotici
I trasposoni eucariotici non differiscono significativamente da quelli procariotici. L'elemento Ty del lievito Saccharomyces cerevisiae è composto da circa 6000 coppie di nucleotidi e presenta alle due estremità lunghe sequenze ripetute dirette denominate LTR (Long terminal repeats) che tuttavia non vengono trasposte. Ty traspone mediante ricombinazione sito-specifica e, in seguito alla sua inserzione, appare affiancato da due sequenze di cinque coppie di nucleotidi. La trasposizione di Ty non è diretta, ma il DNA del trasposone viene prima trascritto in RNA, da cui si forma il DNA che va a inserirsi nel sito bersaglio. Caratteristiche simili hanno i retrovirus, virus tumorali a RNA il cui genoma, all'interno della cellula animale, viene trasformato in DNA a doppia elica che si inserisce nel cromosoma cellulare mediante un processo di trasposizione formando il provirus, da cui viene sintetizzato l'RNA virale. Interessanti sono anche i trasposoni Ac di mais e gli elementi P del moscerino Drosophila melanogaster, che si trasferiscono con meccanismo non replicativo.
Bibliografia
Alberts 1998: Alberts, Bruce e altri, Essential cell biology, New York-London, Garland, 1998 (trad. it.: L'essenziale di biologia molecolare della cellula, 2. ed., Bologna, Zanichelli, 2005).
Brock 1998: Brock, Thomas D., Biology of microorganisms, edited by Michael T. Madigan, John M. Martink, Jack Parker, 8. ed., Needham Heights (Mass.), Simon & Schuster, 1998 (trad. it.: Biologia dei microrganismi, a cura di Bianca Colonna e Stefania Stefani, Milano, CEA, 2003).
Lewin 1997: Lewin, Benjamin, Gene VI, Oxford, Oxford University Press, 1997 (trad. it.: Il gene VI, Bologna, Zanichelli, 1999).
Polsinelli 1993: Polsinelli, Mario e altri, Microbiologia, Torino, Bollati Boringhieri, 1993.
Watson 1965: Watson James D. e altri, Molecular biology of the gene, New York, Benjamin, 1965 (trad. it.: Biologia molecolare del gene, trad. e rev. di Gianfranco Badaracco e altri, 5. ed., Bologna, Zanichelli, 2005).