Biologia dei sistemi
Biologia dei sistemi
La biologia dei sistemi è un approccio interdisciplinare volto alla comprensione del funzionamento dei sistemi biologici: integrando informazioni di diversa natura e studiando la rete di connessioni dinamiche esistenti tra geni, proteine, metaboliti e altre molecole, essa si propone di sviluppare un modello esplicativo dei sistemi viventi. La biologia dei sistemi, che tenta di fornire un'unica chiave di lettura del fenomeno vita, di fatto non è un concetto nuovo, poiché il tentativo di guardare a un sistema vivente in maniera globale per comprenderne appieno il funzionamento è insito nella ricerca scientifica degli ultimi decenni. Tuttavia, esso non è stato ancora sviluppato pienamente per la mancanza di strumenti computazionali e tecnologici adeguati che consentano di valutare simultaneamente più variabili dello stesso sistema.
A causa della sua natura, la biologia dei sistemi attrae diverse aree scientifiche e diverse competenze, cercando di elaborare un linguaggio comune che superi le barriere esistenti fra di esse. Le discipline che contribuiscono al raggiungimento di questo traguardo sono state ampiamente sviluppate nel corso del Novecento con risultati di grandissimo rilievo che ci consentono, per esempio, di esplorare lo spazio, di comunicare attraverso un telefono cellulare, navigare in internet ed estendere sensibilmente la vita media dell'uomo. Ciononostante, ci si ammala ancora di diabete, cancro, malattie neurodegenerative e altre patologie complesse, poiché la medicina contemporanea deve ancora sviluppare strategie efficaci per prevedere e prevenire il verificarsi di tali patologie. La precisa eziologia delle malattie complesse e le ragioni per cui alcuni individui mostrano una maggiore suscettibilità verso di esse, non sono ancora note nel dettaglio. In altre parole, ciò che risulta ancora carente è la nostra conoscenza di come ogni singolo dettaglio intervenga per modificare il sistema biologico.
La biologia dei sistemi affronta sistematicamente questi quesiti. Questa nuova forma di scienza integrata è sorretta in parte dal desiderio di conoscere i segreti dell'universo biologico, ma ancora di più dalla ricchezza delle sue applicazioni. Se da un lato, infatti, essa dovrebbe fornire indicazioni per personalizzare la medicina, dall'altro, potrebbe avere un impatto significativo in campi quali lo sviluppo di nuovi farmaci, la sperimentazione preclinica e clinica necessaria per la loro valutazione, la diagnostica e la predizione dei possibili effetti indesiderati di una terapia. A ciò si aggiunga la possibilità di focalizzare la ricerca sui microrganismi, forzando la loro evoluzione verso uno stato adattativo ottimale, utile, per esempio, a risolvere il problema della resistenza agli antibiotici.
La piramide della complessità
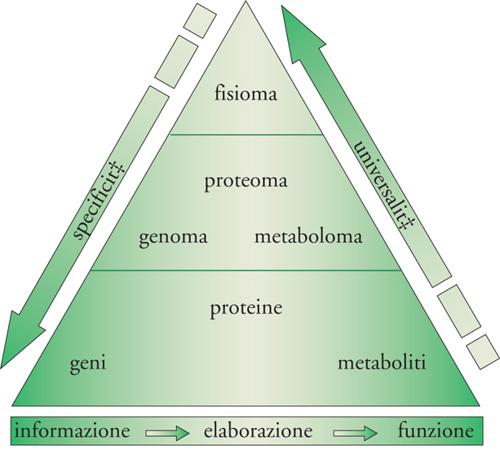
Il riduzionismo della biologia tradizionale (approccio dal basso verso l'alto), che ha dominato la ricerca scientifica per oltre un secolo, ha generato una mole indescrivibile di informazioni sui singoli componenti cellulari e sulla loro funzione, culminando nel tanto discusso tentativo di facilitare la comprensione della vita attraverso la creazione di un 'organismo minimo': un individuo unicellulare, parzialmente generato dall'uomo, contenente il minor numero possibile di geni compatibili con la vita. Sebbene in alcuni casi siano stati ottenuti risultati interessanti, nessuno di essi ha potuto contribuire alla conoscenza globale degli organismi viventi. Di qui la nascita del nuovo modello di ricerca denominato 'biologia dei sistemi' (approccio dall'alto verso il basso). "Gli organismi funzionano in modo integrato: i nostri sensi, i nostri muscoli, il nostro metabolismo e le nostre menti lavorano insieme in perfetta coordinazione. Ma i biologi hanno storicamente studiato gli organismi una parte alla volta e hanno celebrato la moderna abilità di studiarli molecola per molecola, gene per gene. Assistiamo ora alla nascita di una nuova scienza, una scienza fondamentale per il futuro, che cerca di svelare come l'integrazione delle singole parti possa costituire i sistemi biologici" (David Baltimore ‒ premio Nobel per la medicina ‒ 1975). Anziché analizzare i singoli componenti o gli aspetti individuali di ciascun organismo, la biologia dei sistemi si focalizza su tutti i costituenti e sulle interazioni fra di essi, come parti di una rete integrata e interattiva. Tali rapporti sono in ultima analisi responsabili della forma stessa di quell'organismo e di come esso vive e funziona (fig. 2). Come analogia, si consideri un'automobile con il suo motore, i suoi sedili e i suoi fari, e si studi la loro specifica funzione: ciò fornirà informazioni soddisfacenti su queste singole parti, ma non potrà dare origine alla conoscenza del meccanismo operativo dell'automobile, né tantomeno delle modalità da seguire per ripararla in caso di malfunzionamento.
Con la parola 'sistema' si definisce un gruppo di componenti che insieme interagiscono e contribuiscono a formare un'entità più complessa: ciò implica, ed è questo un requisito essenziale per un sistema, che l'entità prodotta dall'interazione delle parti è più grande della somma delle parti stesse. Queste possono infatti combinarsi in modi inaspettati generando proprietà o funzioni riconducibili unicamente al sistema (per es., il movimento controllato dell'automobile) e non ai suoi costituenti (motore, fari, ecc.). Tali proprietà o funzioni vengono definite 'proprietà emergenti'. Poiché le proprietà emergenti sono il risultato delle interazioni fra le parti, il sistema che le comprende viene detto 'irriducibile', ovvero non riconducibile alle sue componenti prese individualmente. In altri termini, il senso di un libro non può essere compreso analizzando una parola alla volta, così come la bellezza di una foresta non può essere apprezzata soffermandosi sui singoli alberi. Un sistema può infine essere definito 'complesso' se le sue proprietà emergenti non sono prevedibili, e i sistemi biologici costituiscono un perfetto esempio di complessità: diversamente da molti altri sistemi assimilabili, infatti, essi possiedono anche la straordinaria capacità di adattarsi in risposta agli stimoli ambientali, allo stress, alla mancanza di cibo o allo stadio dello sviluppo, nonché l'affascinante abilità di correggere i propri errori come le mutazioni geniche.
Tecnologia ad alto flusso
Il XXI sec. è iniziato con il completamento del progetto di sequenziamento del genoma umano. Sebbene questo abbia segnato il raggiungimento di uno straordinario traguardo, appare oggi evidente che l'inventario, seppur dettagliato, di geni, così come di proteine e metaboliti, costituisce solo l'inizio di una nuova e più ambiziosa avventura, il cui obiettivo è la comprensione delle interazioni fra ciascuno degli elementi identificati. La moderna tecnologia ad alto flusso di dati (high throughput), frutto dell'integrazione fra biologia e ingegneria, interviene a questo livello fornendo un supporto indispensabile. Per tecnologia ad alto flusso si intende lo sviluppo di metodi di laboratorio in grado di interrogare simultaneamente un sistema biologico come la cellula in ogni momento della sua vita, e di collezionare tutte le informazioni che ne scaturiscono, generando una banca dati successivamente analizzabile. Un esempio di tecnologia ad alto flusso oggi molto diffusa è il microarray a DNA, basato sull'uso di supporti di vetro di pochi centimetri che, facendo ricorso alla robotica, possono contenere il genoma di un intero organismo. Questi chip a DNA possono poi essere ibridati, ovvero messi a confronto, con il DNA derivante dal medesimo organismo preso in una condizione diversa. Un esperimento con un singolo chip a DNA può produrre informazioni su migliaia di geni contemporaneamente. Al fine di studiare, per esempio, l'effetto di un farmaco su un tipo di cellule, si dovranno porre a confronto i geni di quelle cellule sul microarray, prima e dopo il trattamento con il farmaco. Per individuare tutti i geni responsabili dello sviluppo di un embrione, si metteranno a confronto le molecole di quell'embrione estratte a un particolare stadio dello sviluppo con quelle dello stadio successivo.
Altre metodologie semiautomatiche ad alto rendimento affiancano oggi i microarray a DNA, permettendo, per esempio, l'analisi di RNA o di proteine, ed espandendo le nostre capacità interrogative nei confronti dei sistemi biologici. Dalla somma dei dati così generati è stato possibile identificare diverse tipologie di interazioni, organizzate in reti, nessuna delle quali sembra essere indipendente, ma piuttosto partecipa alla formazione di una 'rete di reti', responsabile della variabilità e del comportamento del sistema biologico. Lo sforzo maggiore richiesto alla biologia contemporanea consiste nell'individuare e realizzare un programma di studio integrato, sia teorico che sperimentale, in grado di delucidare e quantificare le proprietà topologiche e dinamiche delle diverse reti che controllano il comportamento di una cellula e, in ultima analisi, dell'organismo di cui essa fa parte. La scienza che si occupa di far questo, la bioinformatica, attinge le sue informazioni sia dalle moderne tecnologie ad alto flusso, che dalla letteratura scientifica prodotta sino a oggi, mettendo in relazione le diverse discipline afferenti allo studio della biologia dei sistemi.
Teoria delle reti complesse
Un sostanziale supporto viene fornito dalla matematica con la emergente 'teoria delle reti complesse': negli ultimi anni essa ha percorso a grandi passi la strada della comprensione dei principî che sovraintendono alla formazione ed evoluzione di numerose reti, siano esse tecnologiche o sociali. Probabilmente la scoperta più interessante della teoria delle reti complesse consiste nell'aver capito che le caratteristiche strutturali di una rete di interazioni molecolari all'interno di una cellula sono condivise con quelle di altri sistemi complessi, come internet, o i microprocessori di un computer, o addirittura la realtà sociale. Nonostante la notevole diversità dei sistemi in natura, essi sono governati da principî universali, il che permette di utilizzare tutte le conoscenze acquisite dai grandi e ben noti sistemi non-biologici, per caratterizzare il dedalo intricato di relazioni che sussiste in un organismo vivente.
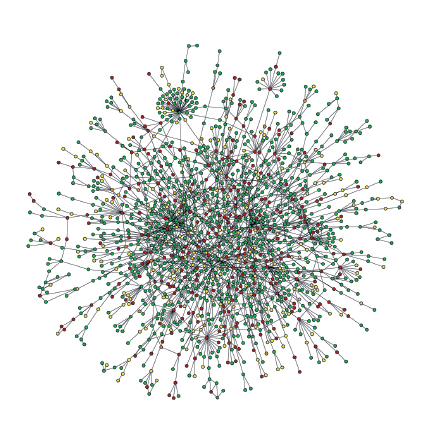
Il comportamento di molti sistemi complessi, dalla cellula a internet, emerge dall'attività concertata di numerosi elementi che interagiscono fra loro attraverso relazioni, la più piccola delle quali è quella esistente fra due soli elementi. In maniera astratta e semplificativa, ogni costituente di un sistema può essere considerato un nodo, connesso ad altri nodi mediante legami: ogni legame si riferisce all'interazione fra due sole parti. Nodi e legami insieme formano una rete o, in linguaggio più strettamente matematico, un grafico. Stabilire anche solo l'identità delle diverse e numerose reti operanti in un sistema biologico non è affatto un compito facile: una proteina, per esempio, può essere considerata un nodo quando si pensa al processo metabolico che la produce, ma è essa stessa un legame quando, agendo come enzima, catalizza una reazione chimica permettendone l'attuazione.
Le reti possono essere definite 'dirette' o 'indirette' a seconda della natura delle interazioni che in esse si manifestano. Nelle reti dirette, le interazioni fra due nodi qualsiasi hanno una direzione definita: per esempio, il flusso di materiale necessario nella produzione di un'automobile, per passare da motore, sedili e fari all'automobile funzionante, o il flusso di informazione che va da un evento di cronaca all'articolo che lo racconta in un quotidiano. Nelle reti indirette invece, i legami non hanno una direzione univoca: per esempio, nelle interazioni fra proteine, un legame rappresenta un'interazione reciproca: se la proteina X lega la proteina Y, di conseguenza la proteina Y lega la proteina X.
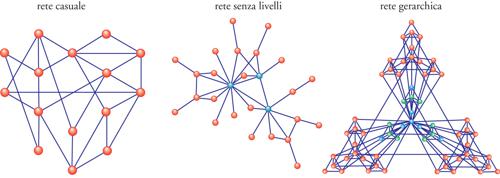
Nello studio delle reti complesse la matematica ha per molti anni considerato tutti i nodi e i legami egualmente possibili e con lo stesso grado di importanza, dando vita cioè a quella che oggi definiamo una 'rete casuale', random network (fig. 4A). In una rete così concepita, giacché i legami vengono posizionati in maniera casuale tra i nodi, ci si aspetta che mediamente i nodi abbiano lo stesso numero di collegamenti con i nodi circostanti. Una serie di scoperte recenti indica invece che il modello casuale non può spiegare le caratteristiche topologiche delle reti reali. Esse infatti sono caratterizzate dall'esistenza di pochi nodi definiti hub (supernodi), con un numero molto elevato di connessioni, i quali tengono insieme i numerosi altri nodi presenti. Reti caratterizzate da questo tipo di distribuzione vengono chiamate scale-free (senza livelli): in netto contrasto con le reti casuali, dove il grado di importanza di ogni nodo è simile alla media e come tale può essere definito un 'nodotipo', le reti scale-free non possiedono noditipo da utilizzare per la caratterizzazione degli altri (fig. 4B). Anche nei sistemi biologici la maggior parte delle reti approssima la topologia scale-free.
La ubiquitarietà delle reti scale-free e dell'esistenza di hub nei network tecnologici così come in quelli biologici e sociali è riconducibile a due fenomeni fondamentali. Il primo fenomeno è il risultato di un processo di crescita graduale cui sono sottoposte le reti, durante il quale nuovi nodi vengono aggiunti al sistema in un periodo di tempo relativamente lungo: è il caso, per esempio, del World wide web, che è stato protagonista di un'espansione da una a più di tre miliardi di pagine in un periodo di dieci anni. Secondariamente, giacché ogni nodo preferisce connettersi con altri nodi che hanno già numerosi legami, si verifica un processo denominato 'attacco preferenziale': quando si naviga in internet si tende a collegarsi con alcuni particolari e ben noti siti, quelli dei motori di ricerca per esempio, contribuendo così al loro rafforzamento e promuovendoli al ruolo di hub.
Una volta identificati i sistemi e le reti in essi operanti, è divenuto evidente che alcune funzioni cellulari non possono essere ricondotte alle teorie sin qui descritte, poiché vengono espletate in maniera modulare. In generale, la modularità si riferisce a un gruppo di molecole (nodi) fisicamente e/o funzionalmente correlate che lavorano insieme per generare una funzione. I moduli possono essere identificati nei sistemi sociali (circoli di amici), così come in quelli tecnologici (siti internet dedicati a un particolare argomento). Similmente, in molti sistemi di ingegneria, da un moderno aereo a un microprocessore, la struttura fortemente modulare è una caratteristica progettuale. L'esistenza di moduli implica che ci siano gruppi di nodi interconnessi che sono relativamente isolati dal resto del sistema. Ecco dunque che la struttura scale-free non riesce in qualche caso a definire i sistemi: in essa infatti gli hub sono sempre in contatto con una grande percentuale dei nodi e non esistono isole di interazioni riconoscibili. La coesistenza del raggruppamento in moduli e della presenza di hub in alcune reti reali, può essere resa compatibile con la topologia della rete gerarchica, in cui i moduli non sono totalmente indipendenti, ma sono piuttosto organizzati in ordine di importanza. La fig. 4C mostra una rete in cui moduli di 4 nodi ciascuno sono integrati in moduli più grandi costituiti da 16 nodi, ognuno dei quali si combina in modo gerarchico in moduli ancora più grandi con 64 nodi.
La modularità gerarchica, presente in tutte le reti cellulari finora studiate, pone un nuovo interessante problema: come e cosa fare per individuare i moduli in maniera automatica e oggettiva. Numerosi metodi di raggruppamento (clustering) sono stati recentemente introdotti per identificare i moduli nelle diverse reti esistenti e ognuno di essi fornisce risultati diversi; questa ambiguità non è frutto solo dell'inadeguatezza dei metodi utilizzati, ma è anche una proprietà intrinseca delle reti gerarchiche: i moduli infatti non hanno una dimensione ben definita e non hanno limiti riconoscibili; dunque i modelli matematici possono solo tentare di visualizzare le relazioni gerarchiche fra moduli di diversa grandezza, relativamente alla funzione che si propongono di studiare.
Dal modello alla realtà
Appare evidente che le dimensioni di questa materia vanno ben oltre le nostre capacità di comprensione e l'immensità delle informazioni ancora da svelare non può essere districata con approcci globali. Due sono pertanto i concetti fondamentali cui si attiene la biologia dei sistemi: da un lato l'importanza della semplificazione che si traduce nella costruzione di modelli, dall'altro l'esigenza di integrare i modelli in un sistema completo. La complessità biologica costringe a frammentare i sistemi in componenti più facilmente comprensibili, ma richiede anche che questi componenti vengano riassociati per delucidare il comportamento che da essi si genera solo quando sono ricostituiti nel sistema biologico. Fortunatamente, molti processi biologici operano nello stesso modo in quasi tutti i sistemi. Gli scienziati pertanto usano gli organismi più semplici, detti 'sistemi modello', sia per gli studi iniziali che per la validazione delle ipotesi.
Dopo l'applicazione degli algoritmi della bioinformatica ai quesiti biologici, infatti, i dati generati necessitano di conferme sperimentali, più facilmente ottenibili con l'uso di organismi meno complessi e che, riproducendosi rapidamente, consentono lo studio di diverse generazioni in un periodo di tempo relativamente breve. Nasce così il tentativo di creare dei simulatori: programmi sofisticati lanciati parallelamente su potenti computer che cercano di simulare le diverse reti che costituiscono i sistemi biologici. I primi simulatori prodotti, non riescono neanche lontanamente a imitare un sistema reale; quel che manca è la simulazione della capacità dei sistemi biologici di adattarsi ai cambiamenti esterni e interni, mantenendo un comportamento pressoché normale: ovvero la loro robustezza.
La robustezza è una delle caratteristiche essenziali dei sistemi biologici. Comprendere il meccanismo che la determina è particolarmente importante poiché fornisce informazioni su come un organismo operi per mantenersi funzionale malgrado cambiamenti di temperatura, carenze di cibo, mutazioni geniche, metabolismi alterati, infezioni, dandoci la possibilità di aiutare il sistema, per esempio nel corso di una malattia. La robustezza dei sistemi biologici è determinata da quattro fattori chiave: il controllo del sistema, cioè l'esistenza di campanelli di allarme che modificano la risposta biologica quando se ne verifica la necessità; la ridondanza, ovvero l'esistenza di più meccanismi parzialmente sovrapposti che raggiungono il medesimo obiettivo, nel caso in cui il meccanismo principale sia stato compromesso; la modularità, che limita i danni al modulo colpito, non permettendone l'espansione; la stabilità strutturale, che consente la variazione di alcuni parametri senza compromettere la funzionalità del sistema. Ovviamente non tutti i sistemi sono egualmente robusti, ma è di fondamentale importanza conoscere quali parti del sistema non lo sono e perché. La robustezza inoltre, è inevitabilmente associata anche alla vulnerabilità: sebbene infatti molte reti cellulari siano perfettamente adattate per compensare la maggior parte delle perturbazioni, esse collassano quando alcune componenti ben selezionate vengono distrutte.
Una sfida per il futuro
La malattia è la condizione di un organismo vivente, sia esso animale o vegetale, che si origina dal malfunzionamento di uno o più dei suoi costituenti. Parlando di sistemi, si dovrebbe dire che una malattia rappresenta la rottura di una o più parti del sistema stesso che, compromettendo le interazioni con altri componenti, inficia una o più proprietà emergenti. Anche un comune mal di testa può essere considerato una compromissione, seppure passeggera, del sistema. Studiare i meccanismi che generano le malattie è sempre stato uno degli obiettivi principali della biologia, al fine di modificare la medicina, storicamente reattiva, in medicina preventiva. Attualmente infatti, è necessario aspettare che un problema si verifichi prima di effettuare una cura, mentre la medicina del futuro si propone di essere predittiva: esaminando ogni individuo nella sua totalità si dovrebbe poter stabilire quale malattia è propenso a contrarre, la probabilità con cui la svilupperà e quando ciò dovrebbe verificarsi. Inoltre dovrebbe poter fare affidamento su metodologie di indagine più sofisticate e accurate per distinguere tra individui e tra forme anche simili della medesima patologia, determinando con precisione il trattamento più adatto.
Paradossalmente la biologia dei sistemi soffre del fatto che possiede troppe informazioni da decifrare e allo stesso tempo tali informazioni non sono sufficienti per gli obiettivi che essa si prefigge di raggiungere. Di qui la necessità sia di potenziare il livello di automazione delle procedure sperimentali esistenti (come i microarray), sia di sviluppare nuove e sofisticate metodologie di analisi. La miniaturizzazione, l'automazione e l'integrazione dei diversi componenti, così come l'aumento del flusso di dati e la riduzione dei costi di analisi si stanno concretizzando oggi nello sviluppo della micro-fluidica, della nano-tecnologia e della femto-chimica, in cui migliaia di misurazioni possono essere effettuate nello stesso momento su un numero enorme di campioni. L'uso di queste tecnologie permetterà di osservare il sistema biologico in un modo che oggi sembra impossibile, migliorando sensibilmente la nostra comprensione di esso e di conseguenza il suo controllo. Alla fine di questo percorso si potrebbe essere in grado di disegnare ex novo sistemi biologici, crescere organi di un paziente partendo dai suoi stessi tessuti, utilizzare materiale biologico per la robotica o per costruire computer. L'uso di materiale capace di autoripararsi e autocontrollarsi potrebbe rivoluzionare tutto il sistema industriale.
Bibliografia
Albert 1999: Albert, Réka - Jeong, Hawoong - Barabási, Albert-László, Diameter of the World Wide Web, "Nature", 401, 1999, pp. 130-131.
Albert 2000: Albert, Réka - Jeong, Hawoong - Barabási, Albert-László, Error and attack tolerance in complex networks, "Nature", 406, 2000, pp. 387-482.
Albert, Barabási 2000: Albert, Réka - Barabási, Albert-László, Topology of evolving networks: local events and universality, "Physical review letters", 85, 2000, pp. 5234-5237.
Albert, Barabási 2002: Albert, Réka - Barabási, Albert-László, Statistical mechanics of complex networks, "Review of modern physics", 74, 2002, pp. 47-97.
Barabási 1999: Barabási, Albert-László - Albert, Réka - Jeong, Hawoong, Mean-field theory for scale-free random networks, "Physica A", 272, 1999, pp. 173-189.
Barabási 2004: Barabási, Albert-László, Link. La nuova scienza delle reti, Torino, Einaudi, 2004.
Barabási, Albert 1999: Barabási, Albert-László - Albert, Réka, Emergence of scaling in random networks, "Science", 286, 1999, pp. 509-512.
Barabási, Oltvai 2004: Barabási, Albert-László - Oltvai, Zoltán N., Network biology: understanding the cells's functional organization, "Nature reviews. Genetics", 5, 2004, pp. 101-113.
Cusick 2005: Cusick, Michael E. e altri, Interactome: gateway into systems biology, "Human molecular genetics", 2005, 14 (suppl. 2), pp. 171-181.
Grimm 2005: Grimm, Volker e altri, Pattern-oriented modeling of agent-based complex systems: lessons from ecology, "Science", 310, 2005, pp. 987-991.
Kelly 1994: Kelly, Kevin, Out of control: the new biology of machines, social systems, and the economic world, London, Fourth Estate, 1994.
Kitano 2002: Kitano, Hiroaki, Systems biology: a brief overview, "Science", 295, 2002, pp. 1662-1664.
Oltvai, Barabási 2002: Oltvai, Zoltán N. - Barabási, Albert-László, Life's complexity pyramid, "Science", 298, 2002, pp. 763-764.
Ravasz 2002: Ravasz, Erzsébet e altri, Hierarchical organization of modularity in metabolic networks, "Science", 297, 2002, pp. 1551-1555.
Sweetlove, Fernie 2005: Sweetlove, Lee J. - Fernie, Alisdair R., Regulation of metabolic networks: understanding metabolic complexity in the systems biology era, "New phytologist", 168, 2005, pp. 9-24.
Siti internet
http://www.sorcerer2expedition.org/version1/HTML/main.htm
http://www.msr-unitn.unitn.it/
http://www.tigem.it/Research/Personal%20Web%20Page_files/dibernardo/Website/Group_Frame_blue.htm
http://cgr.harvard.edu/research/systems_biology.html