RNA. Apparati per la maturazione dell'RNA
RNA. Apparati per la maturazione dell'RNA
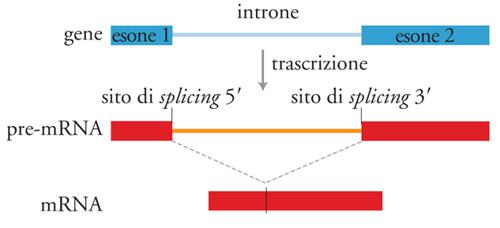
La maggior parte dei geni degli eucarioti è interrotta da sequenze che non codificano alcuna sequenza amminoacidica. Dopo essere state trascritte, queste sequenze non codificanti, gli introni, vengono rimosse e quelle codificanti, gli esoni, ricongiunte tra loro mediante un processo chiamato splicing. Ci sono quattro tipi differenti di splicing: lo splicing del pre-tRNA, catalizzato da enzimi proteici, gli autosplicing di gruppo I e gruppo II, osservati negli RNA catalitici, e lo splicing del pre-mRNA, che avviene in una grossa particella ribonucleoproteica chiamata 'spliceosoma'. Cinque piccoli RNA nucleari (snRNA), insieme a molte proteine, si assemblano sul substrato del pre-mRNA in un processo a più stadi. Si ritiene che il sito attivo dello spliceosoma sia composto da RNA (gli snRNA, evolutivamente conservati), mentre si reputa che alcune proteine giochino un ruolo strutturale o regolativo, mentre altre siano ATPasi o RNA-elicasi.
In seguito alla scoperta, avvenuta nel 1953, della struttura del DNA e della sua capacità di conservare l'informazione genetica, l'idea che vi fosse una corrispondenza lineare (colinearità) tra la sequenza nucleotidica di un gene e il suo prodotto polipeptidico era diventata un principio centrale della biologia. Per i biologi è stato quindi uno shock quando i primi confronti tra le sequenze di mRNA eucariotici e quelle dei geni corrispondenti dimostrarono che tali geni sono spesso interrotti da sequenze che non codificano il prodotto polipeptidico corrispondente del gene. Per esempio, il gene per la subunità β dell'emoglobina possiede due introni. Dato che queste sequenze non si trovano nell'mRNA per la β-emoglobina, esse devono essere rimosse dall'RNA precursore dell'mRNA, dopo la trascrizione del gene. Gli introni vengono eliminati mediante due tagli endonucleolitici e le estremità delle due sequenze codificanti, o esoni, vengono risaldate tra loro per formare l'RNA maturo (fig. 1).
Con l'avanzamento delle tecniche di clonaggio e sequenziamento del DNA, ci si è subito resi conto che alcuni geni sono di gran lunga più complicati del gene della β-emoglobina e, pertanto, nel processo di maturazione ci deve essere un'accuratezza estrema. Un errore, anche di un singolo nucleotide, creerebbe uno spostamento del modulo di lettura nell'mRNA e la traduzione determinerebbe un prodotto senza senso. Approssimativamente, il 15% delle mutazioni che causano malattie genetiche colpiscono lo splicing del pre-mRNA. Si possono avere vie alternative di splicing: lo splicing alternativo fa aumentare la capacità codificante di un singolo gene, permettendo la sintesi di isoforme proteiche strutturalmente e funzionalmente distinte. Esoni alternativi hanno particolari segnali di splicing e la loro inclusione è modulata da fattori che, agendo in trans, riconoscono elementi di sequenza cis, agenti positivi (splicing enhancers) e/o negativi (splicing silencers) che possono essere sia esonici che intronici. Lo splicing alternativo crea una diversificazione del trascrittoma, forse legata alla speciazione. Un gran numero di geni di organismi pluricelluari subisce splicing alternativo, al contrario di ciò che avviene negli eucarioti unicellulari. Un'analisi comparativa dei siti di splicing al 5′ in eucarioti uni- e multicellulari ha suggerito l'ipotesi che lo splicing alternativo sia il risultato di un 'rilassamento' del riconoscimento del sito di splicing al 5′ negli organismi che originariamente sopportavano solo uno splicing costitutivo.
Splicing e autosplicing
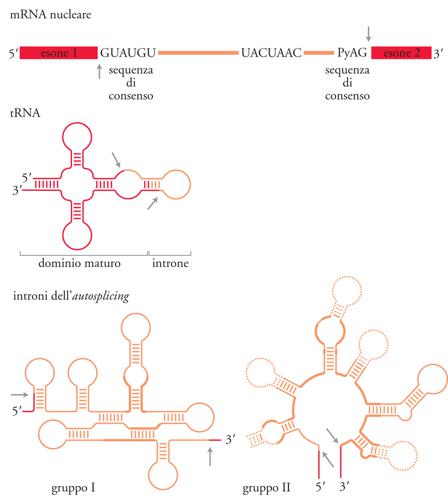
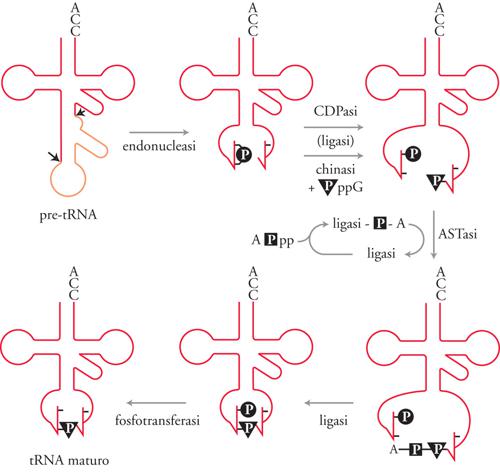
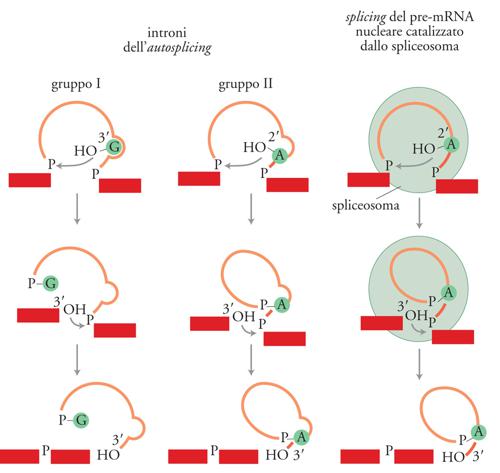
Quando nel 1977 vennero scoperti gli introni, sembrò plausibile che ci fosse un unico meccanismo di splicing. Successivamente si è trovato che esistono invece perlomeno quattro meccanismi diversi (fig. 2), in ciascuno dei quali la struttura dell'RNA precursore è diversa. Per lo splicing degli introni dai pre-mRNA non esiste una struttura intronica evolutivamente conservata. Invece, come mostrato nella fig. 2, le estremità introniche sono identificate da sequenze consensus minime. Alcuni geni codificanti i tRNA, sia negli eucarioti sia negli archeobatteri, contengono piccoli introni che di solito interrompono l'ansa dell'anticodone. In questo caso nei siti di splicing non ci sono sequenze conservate e l'introne viene riconosciuto per la sua posizione nella struttura terziaria del pre-tRNA. Scoperta sorprendente e inaspettata è stata trovare che esistono due differenti classi di introni che sono in grado di compiere autosplicing. Questi RNA catalitici hanno la capacità di rimuovere se stessi dal trascritto. In questo caso è importante la struttura secondaria e terziaria dell'introne stesso; i due gruppi di introni in grado di compiere autosplicing, gli introni di gruppo I (o classe I) e di gruppo II (o classe II), hanno strutture secondarie e terziarie distinte. I meccanismi chimici di ciascuno dei quattro tipi di splicing sono oggi conosciuti. Nello splicing dei tRNA degli eucarioti, la reazione è catalizzata da tre enzimi: una endonucleasi, una ligasi e una fosfotransferasi (fig. 3). La endonucleasi taglia il pre-tRNA ai due siti di splicing 5′ e 3′ (così indicati perché si trovano alle estremità 5′ e 3′ dell'introne) rilasciando due 'mezze molecole' di tRNA con una estremità 2′-3′/ fosfato ciclico e una estremità 5′-OH. La tRNA ligasi, attraverso una serie complessa di reazioni ATP- e GTP-dipendenti, unisce covalentemente le due mezze molecole. Dopo la reazione della ligasi, il 2′-fosfato rimasto è rimosso dalla fosfotransferasi. La rimozione degli introni dal pre-mRNA nucleare avviene in una struttura complessa chiamata spliceosoma, che viene assemblato ex novo per ciascun evento di splicing. Lo spliceosoma, una volta assemblato, catalizza due reazioni di transesterificazione sequenziali che rimuovono un introne dal pre-mRNA e riuniscono gli esoni (fig. 4). Nella prima reazione, il 2′-OH di un particolare residuo adenosinico prossimo al sito 3′ di splicing attacca il legame fosfodiesterico al sito 5′ di splicing producendo due intermedi: l'esone 1 e l'introne-esone 2 che assume una struttura ramificata a forma di lazo, detto 'lariat', nella quale l'estremità 5′ dell'introne è connessa alla adenosina del sito di ramificazione tramite un legame fosfodiesterico 2′-5′. Nella seconda reazione, l'estremità 3′-OH dell'esone 1 attacca il legame fosfodiesterico al sito 3′ di splicing, dando come risultato la formazione dell'mRNA maturo e la liberazione dell'introne a forma di lazo.
Nella fig. 4 sono messi a confronto il meccanismo di splicing degli introni ad autosplicing e quello dei pre-mRNA. In entrambi i casi il processo consiste di due reazioni fosfotransferasiche. Nello splicing degli introni del gruppo I, la reazione richiede un cofattore guanosinico. Nella prima reazione fosfotransferasica, il 3′-OH della guanosina (G) attacca il legame fosfodiesterico al sito di splicing 5′ trasferendo la G all'estremità 5′ dell'introne con un legame fosfodiesterico 3′-5′ e dando, come intermedi della reazione, l'esone 5′ e l'introne-esone 3′ contenente il cofattore guanosinico all'estremità 5′. Nella seconda reazione, il 3′-OH dell'esone 5′ attacca il legame fosfodiesterico alla giunzione di splicing 3′ dando, come prodotti di reazione, l'RNA maturo e un introne che termina con una Guanina. In una serie di esperimenti, Thomas Cech e collaboratori hanno dimostrato che l'introne stesso continua a essere cataliticamente attivo e può portare avanti un certo numero di altre reazioni fosfotransferasiche. Inoltre Craig Peebles e Renske Van der Veen hanno dimostrato che gli introni ad autosplicing che appartengono al gruppo II vengono rimossi con un processo identico a quello dello splicing del pre-mRNA. Questa scoperta ha portato a stabilire due principî importanti: lo splicing del pre-mRNA è una reazione catalizzata dall'RNA e i due processi, in termini evolutivi, discendono da un antenato comune.
Accanto a questa ipotesi, è stato anche proposto che gli introni nucleari discendano dagli introni batterici di gruppo II. Questi ultimi sarebbero stati inglobati nelle cellule eucariotiche perché erano contenuti nei batteri che invasero i precursori delle cellule eucariotiche moderne e che divennero poi i mitocondri. Le due reazioni, sebbene abbiano un meccanismo simile, richiedono molecole differenti. In generale gli introni ad autosplicing non richiedono fattori, sebbene per alcuni introni del gruppo II sia necessaria una proteina, probabilmente per stabilizzare la struttura terziaria dell'introne. La maturazione del pre-mRNA ha bisogno invece di cinque molecole di RNA aggiuntive, gli snRNA (Small nuclear RNA), di oltre 100 proteine e dell'idrolisi dell'ATP. Oggi si ritiene che gli snRNA, interagendo con il substrato di pre-mRNA, formino una struttura di RNA che catalizza la reazione.
Spliceosoma
La maggior parte delle informazioni sui componenti dello spliceosoma proviene da studi sul lievito Saccharomyces cerevisiae e sull'uomo. Le informazioni provenienti dai due sistemi convergono e sono ora noti molti esempi di componenti inizialmente scoperti nel lievito e successivamente identificati nell'uomo e viceversa. È chiaro, sebbene si osservino alcune interessanti differenze, che il meccanismo di splicing del pre-mRNA è conservato nell'intero regno eucariotico. Per lo splicing del pre-mRNA sono necessari cinque RNA, gli snRNA U1, U2, U4, U5 e U6. Gli RNA U1, U2, U4 e U5 hanno, alla loro estremità 5′, una tipica struttura a cappuccio ipermetilato, m3G, mentre U6 ha un γ-fosforilmetilestere. Inoltre, tutti gli RNA U contengono una varietà di altre basi modificate, come la pseudouridina e vari nucleosidi metilati. Gli snRNA si trovano nella cellula come particelle ribonucleoproteiche, RNP, dette snRNP (Small nuclear ribonucleoproteins). Quattro diverse snRNP, U1, U2, U5 e U4/U6 funzionano nel processo di splicing. U4 e U6, unite da 21 coppie di basi, si trovano nella stessa particella. Tutte le snRNP hanno un gruppo di proteine in comune, le proteine Sm, che si legano alla sequenza AU4‒6G che si trova negli RNA U1, U2, U4 e U5. Queste proteine sono anche bersagli comuni di anticorpi che compaiono in pazienti affetti da malattie autoimmuni sistemiche. Inoltre, ciascuna snRNP ha un gruppo di proteine uniche. Oltre a queste proteine associate stabilmente con le snRNP ve ne sono altre, necessarie per lo splicing, che non si trovano nelle snRNP. Molte delle proteine che fanno parte dello spliceososma sono state identificate per la prima volta nel lievito mediante selezione genetica. Uno degli approcci più efficaci è stato la selezione di banche di mutanti di lievito termosensibili per difetti nello splicing del pre-mRNA. La prima banca di questo tipo è stata costruita negli anni Sessanta del Novecento da Leland H. Hartwell. Questi e Calvin S. McLauglin hanno analizzato questa banca per difetti nella sintesi dell'RNA, che poi si sono rivelati interessare l'RNA ribosomale, dato che nel lievito la maggior parte dell'RNA è costituito da rRNA. Un'ulteriore analisi di un'altra banca di oltre mille mutanti termosensibili ha portato all'isolamento di altri geni, e da allora il numero ha continuato a crescere. Questi geni vengono ora chiamati PRP (Pre-mRNA processing).
Assemblaggio dello spliceosoma
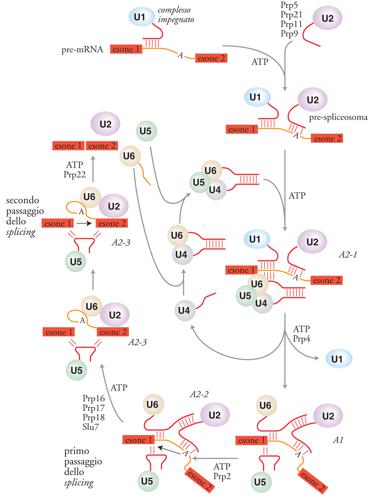
L'assemblaggio dello spliceosoma, schematizzato nella fig. 5, inizia con l'interazione della snRNP U1 con il pre-mRNA. La sequenza all'estremità 5′ dell'RNA U1 è complementare alla sequenza intronica conservata che si trova al sito 5′ di splicing, e l'appaiamento di queste regioni è essenziale. Il complesso U1-pre-mRNA è chiamato 'complesso impegnato' (CC, Commitment complex) perché, una volta formatosi, il pre-mRNA è destinato a completare la reazione di splicing, anche se l'appaiamento tra il sito di splicing 5′ e U1 non è sufficiente per uno splicing ottimale. Questo è il solo passaggio nell'assemblaggio dello spliceosoma che non richiede idrolisi di ATP. Nello stadio seguente, la particella snRNP U2 si unisce al complesso impegnato per formare un complesso stabile chiamato 'pre-spliceosoma'. In questo passaggio, una sequenza dell'RNA U2, vicina alla sua estremità 5′, si appaia con la sequenza intronica conservata che costituisce il sito di ramificazione, vicino al sito 3′ di splicing. La formazione del pre-spliceosoma richiede almeno quattro proteine Prp: Prp 5, 9, 11 e 21.
Negli estratti di cellule di mammifero questo passaggio richiede l'azione di una proteina ben studiata, chiamata U2AF, che sembra faciliti l'appaiamento tra l'RNA U2 e la sequenza del sito di ramificazione. Gli RNA U4 e U6 si trovano nella stessa snRNP, che contiene un RNA duplex U4/U6 che comporta l'appaiamento di 21 basi in due eliche e che si lega alla snRNP U5 per formare una tripla snRNP. Quando entrambe le eliche degli RNA U4/U6 si dissociano, l'RNA U4 lascia lo spliceosoma o, perlomeno, la sua interazione risulta fortemente destabilizzata. La presenza di U4 non è necessaria per nessuna delle reazioni catalitiche. Presumibilmente, a questo punto, il centro catalitico dello spliceosoma è quasi completo. Tuttavia, l'attivazione richiede un ulteriore evento ATP-dipendente, mediato dalla proteina di lievito Prp2. L'attivazione dello spliceosoma per il secondo passaggio dello splicing richiede ancora un'altra reazione ATP-dipendente e presumibilmente un altro cambiamento strutturale. In questa fase sono necessarie almeno quattro proteine di lievito: Prp 16, 17, 18 e Slu7. Dopo il completamento della reazione di splicing è necessaria almeno un'altra reazione ATP-dipendente per il rilascio dell'mRNA dallo spliceosoma, mediato da Prp22. L'introne resta associato allo spliceosoma e, in seguito, viene degradato e le snRNP riciclate.
Interazioni degli RNA nella formazione dello spliceosoma: l'assemblaggio progressivo del centro attivo
Il primo evento dello splicing consiste nell'identificazione dell'introne e dei siti di giunzione dello splicing. Nella successiva formazione dello spliceosoma i siti di giunzione si avvicinano per formare un sito attivo. Due dei cinque snRNA, U1 e U4, funzionano soltanto durante l'assemblaggio, mentre U2, U5 e U6 sono essenziali sia nell'assemblaggio sia nella catalisi. I segnali che identificano gli introni nel trascritto sono minimi. Nel lievito essi sono la sequenza del sito di splicing all'estremità 5′ GUAUGU, la sequenza del sito di ramificazione UACUAAC e la sequenza del sito 3′ di splicing PyAG. Nei Mammiferi le sequenze di consenso sono le stesse, anche se sono meno conservate, specialmente la sequenza del sito di ramificazione; inoltre, nell'introne, un ruolo importante è svolto da un tratto di polipirimidine adiacente al sito 3′ di splicing, di solito presente anche nel lievito.
L'introne viene riconosciuto inizialmente da snRNP U1 mediante un appaiamento di basi di una sequenza prossima alla sua estremità 5′ con il sito 5′ di splicing. Riguardo al sito 5′ di splicing, c'è una differenza molto interessante tra gli introni di lievito e quelli di mammifero. Nei Mammiferi, nei quali i geni hanno molti introni con sequenze esoniche corte e introniche lunghe, il sito 5′ di splicing nell'introne a valle è riconosciuto dalla snRNP U1, e nei successivi stadi dell'assemblaggio le snRNP U2 e U5-U4/U6 si associano al sito 3′ dell'introne a monte. Questo sistema di riconoscimento è chiamato 'definizione dell'esone'. Al contrario, nel lievito, i cui geni in generale hanno introni singoli e piccoli, il riconoscimento è operato mediante 'definizione dell'introne'. Sebbene la snRNP U1 si appai soltanto con il sito 5′ di splicing, ci sono prove che già a questo stadio precoce venga riconosciuta anche la regione del sito di ramificazione. Presumibilmente questo riconoscimento sembra essere determinato da interazioni proteina-proteina. Nel passaggio successivo la particella snRNP U2 si unisce al complesso impegnato e si forma il complesso stabile chiamato pre-spliceosoma. In questo passaggio, quindi, con l'appaiamento tra la sequenza prossima all'estremità 5′ dell'RNA U2 e quella intronica conservata di ramificazione vicina al sito 3′ di splicing, la penultima Adenina nella sequenza UACUAAC, destinata a formare il punto di ramificazione, risulta sporgere dall'elica.
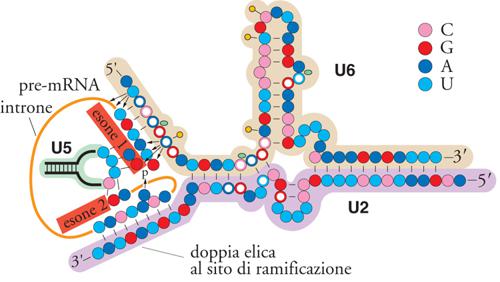
Negli estratti, la particella snRNP U5 e la particella snRNP U4/U6 si uniscono a formare una tripla snRNP e, apparentemente, è in questa forma che le particelle si uniscono allo spliceosoma. Durante questo evento si hanno varie interazioni cruciali RNA-RNA, alcune nuove, altre in sostituzione di interazioni già preesistenti. La prima di queste coinvolge l'appaiamento U1-sito 5′ con l'interazione U6-sito 5′. L'estremità 5′ della sequenza altamente conservata ACAGAGA di U6 si appaia al sito 5′ di splicing, sostituendo le interazioni U1-introne e destabilizzando le interazioni tra U1 e lo spliceosoma. Oltre alle interazioni tra U6 e il sito 5′ di splicing, due basi che si trovano nell'ansa centrale molto conservata dell'snRNA U5 interagiscono con le ultime due basi dell'esone a monte. Mentre la localizzazione del sito di splicing 5′ è definita dalle interazioni iniziali U1-sito 5′, le interazioni tra U6 e U5 con il sito 5′ avvengono in una fase successiva dell'assemblaggio dello spliceosoma e hanno entrambe un ruolo nel definire il preciso legame fosfodiesterico che deve essere attaccato durante il primo stadio dello splicing. Come vedremo anche per i passaggi successivi, si osserva una certa molteplicità dei meccanismi di identificazione dei siti di splicing, che comunque è senza dubbio essenziale per ottenere la necessaria accuratezza della reazione.
Nello stadio successivo dell'assemblaggio gli RNA U4/U6 si dissociano, U4 lascia lo spliceosoma oppure il suo legame con questo si destabilizza e la dissociazione delle eliche U4/U6 permette a U6 di formare un'elica U2/U6 e un'ansa intramolecolare prossima all'estremità 3′ di U6. Inoltre, una sequenza vicina all'estremità del suo 3′ si appaia con l'estremità 5′ di U2 per formare un'elica che è importante nello splicing nelle cellule di mammifero, ma forse non in quelle del lievito. L'insieme delle interazioni che avvengono tra U6 e il sito 5′ di splicing, tra U6 e U2 e tra U2 e la sequenza nel sito di ramificazione nell'introne servono per posizionare il pre-mRNA in modo che il sito 5′ di splicing si trovi in prossimità della sequenza del sito di ramificazione (fig. 6). Presumibilmente, a questo punto il centro catalitico dello spliceosoma è pronto per catalizzare il primo passaggio dello splicing. Prima che questo accada, è necessario che avvenga un rimodellamento ATP-dipendente dello spliceosoma, catalizzato da Prp2. Dopo di ciò si verifica la prima delle due reazioni fosfotransferasiche. Nella reazione di autosplicing degli introni del gruppo I, il primo e il secondo passaggio consistono in due reazioni quasi esattamente inverse. La reazione di transesterificazione SN2 richiede l'inversione della stereochimica del carbonio nel suo legame con l'ossigeno e, come ci si aspetta, soltanto il secondo passaggio è inibito dalla sostituzione del fosfato reattivo con un RP del fosfotioato diastereoisomero. Tra le interazioni che definiscono il secondo passaggio e l'identificazione del sito 3′ di splicing, ci può essere un'interazione diretta tra i residui 5′G e 3′G dell'introne.
L'eccezione conferma la regola: introni con la sequenza di consenso AT-AC
Una classe minore di introni, trovata finora soltanto in pochi geni di Drosophila e di altri Metazoi, possiede la sequenza AT invece che GT al sito 5′ di splicing e la sequenza AC invece che AG al sito 3′. Questi introni contengono peculiari sequenze di consenso a entrambi i siti 5′ e 3′ di splicing e al posto della sequenza di ramificazione contengono la sequenza TCCTTAAC, 8÷11 nucleotidi a monte del sito 3′ di splicing. Lo splicing di questi introni richiede l'assemblaggio di uno spliceosoma speciale ('spliceosoma atac'). Lo spliceosoma atac ha soltanto l'snRNA U5 in comune con il normale spliceosoma GUAG e quattro nuove snRNP. U11 prende il posto di U1 e riconosce il sito di splicing 5′, U12 prende il posto di U2 e si appaia con la nuova sequenza del sito di ramificazione, e due nuovi RNA non molto abbondanti, U4atac e U6atac, sostituiscono U4 e U6. Questi RNA presentano soltanto il 40% di omologia con U4 e U6, però si appaiano tra loro per formare una struttura analoga a U4/U6. È significativo il fatto che U6atac formi con U12 una serie di interazioni simile a quella formata da U6 e U2 nel centro dello spliceosoma (fig. 7). Un'interazione tra U6atac e le sequenze introniche vicino al sito 5′ di splicing porta tale sito in prossimità di una sequenza di polipurine, AAGGAG, apparentemente analoga alla sequenza ACAGAGA altamente conservata ed essenziale che si trova in U6. Così, il modo in cui questi RNA molto diversi si assemblano testimonia l'importanza della serie centrale di interazioni in ambedue i tipi di spliceosoma. Sembra chiaro che i due meccanismi di splicing siano evolutivamente correlati.
Ruolo delle proteine nello splicing
Nel processo dello splicing sono coinvolte numerose molecole proteiche che in molti casi risultano essenziali. Esse svolgono un ruolo strutturale come componenti delle snRNP, sono coinvolte negli stadi iniziali del riconoscimento dei siti di splicing e, se vengono utilizzati siti alternativi, nella selezione del sito di splicing. Le ATPasi dello spliceosoma, molto probabilmente, spiegano la necessità dell'idrolisi dell'ATP durante lo splicing, e infine l'immediata vicinanza di certe proteine al centro dello spliceosoma suggerisce che esse potrebbero avere un ruolo, diretto o indiretto, nella catalisi stessa. Una descrizione di due classi proteiche, le proteine SR e le ATPasi spliceosomali provviste di DEAD box (acronimo della sequenza di amminoacidi: acido aspartico, D, acido glutammico, E, alanina, A, acido aspartico, D), e di una proteina molto conservata, la Prp8, potrà servire a illustrare che cosa si conosce e che cosa si deve ancora scoprire del ruolo svolto dalle proteine nello splicing.
Proteine SR
La caratterizzazione delle proteine SR iniziò con la scoperta che la proteina di mammifero ASF/SF2 (Alternative splicing factor, splicing factor 2) può, a seconda della sua concentrazione negli estratti, mediare la scelta tra due siti 5′ di splicing che competono fra loro nel pre-mRNA precoce di SV40. ASF/SF2 contiene all'estremità amminoterminale due sequenze che legano l'RNA, indicate come RRM (RNA recognition motif) e all'estremità carbossiterminale un dominio che contiene residui ripetuti di serine (S) e arginine (R) da cui il nome di proteine SR. Frequentemente i residui di serina sono fosforilati ed è probabile che la fosforilazione controlli l'attività delle proteine. Successivamente è stato scoperto che cellule di mammifero contengono una grande famiglia di proteine strutturalmente correlate a ASF/SF2. Anche altri fattori di splicing contengono il motivo SR; tra di essi la proteina U1A (proteina che lega lo snRNA U1) e le subunità di 65 e 35kDa dello U2AF (Auxilary factor della riboproteina U2). In generale, le proteine SR agiscono nella fase iniziale dell'assemblaggio dello spliceosoma, mediando il riconoscimento del sito 5′ di splicing oppure, nel passaggio successivo dell'assemblaggio, mediando la comunicazione tra il sito 5′ di splicing e la sequenza del sito di ramificazione.
Le proteine SR possono promuovere l'uso di un particolare sito 5′ di splicing, legandosi a tale sito, e reclutare la snRNP U1 su quel sito 5′ di splicing. In alternativa, la funzionalità di alcuni siti non ottimali può essere promossa mediante il legame di una o più proteine SR a una sequenza vicina, ricca in purine, chiamata 'enhancer esonico'. Diversi enhancer esonici possono essere attivati da combinazioni differenti di proteine SR o da specifiche proteine SR il cui ruolo è soltanto quello di promuovere l'uso di un particolare sito di splicing. In Drosophila, per esempio, specifiche proteine SR, prodotte dai geni Tra e Tra2 (Transformer), controllano il tipo di splicing del pre-mRNA del gene Double sex (Dsx) legandosi a un enhancer e reclutando altre proteine SR mediante una interazione tra i domini SR. Questo complesso meccanismo di splicing alternativo produce come risultato finale la determinazione del sesso del moscerino, a seconda che un particolare esone venga incluso (Dsx femmina specifico) o escluso (Dsx maschio specifico).
Proteine provviste di DEAD box
Di particolare importanza per la comprensione dello splicing del pre-mRNA è la determinazione del ruolo dell'idrolisi dell'ATP nella reazione in vitro. Studi di genetica, in lievito, sulle proteine necessarie allo splicing del pre-mRNA, hanno rivelato almeno uno degli elementi che spiegano la necessità di ATP nell'assemblaggio dello spliceosoma: le proteine Prp2, Prp5, Prp16, Prp22 e Prp28. Queste proteine hanno in comune vari motivi di sequenza tra i quali un tipico sito per il legame dell'ATP: la sequenza DEAD da cui origina il nome di proteine con DEAD box. Studi biochimici condotti su queste proteine ubiquitarie hanno rivelato che esse sono ATPasi RNA-dipendenti, e alcune di esse si sono dimostrate essere delle RNA-elicasi, perché sono in grado di separare in una reazione ATP-dipendente molecole di RNA a doppia elica. Per la maggior parte delle proteine contenenti la DEAD box tale attività elicasica non è stata dimostrata, per cui la descrizione più appropriata potrebbe essere quella di 'ATPasi spliceosomali'. Studi sulle proteine contenenti la DEAD box suggeriscono che ciascun passaggio ATP-dipendente nell'assemblaggio potrebbe essere mediato da una di queste proteine. La formazione del pre-spliceosoma richiede Prp5, Prp2 è richiesta per un evento di attivazione che avviene prima del primo passaggio, Prp16 per l'attivazione del secondo passaggio, e Prp22 per il rilascio dei prodotti. Prp2, Prp16 e Prp22 fanno tutte parte della famiglia di proteine a DEAD box.
Prp8
Prp8 è uno dei membri del gruppo originario di 10 geni di lievito coinvolti nello splicing del pre-mRNA e codifica una proteina molto grande, di 2413 amminoacidi, altamente conservata, dal lievito ai Nematodi ai Mammiferi. Originariamente, Prp8 fu caratterizzata come una proteina facente parte integrale della snRNP U5. I primi mutanti di Prp8 bloccavano lo splicing a livello del primo passaggio. Legami incrociati sono stati inoltre osservati tra Prp8 e nucleotidi adiacenti al sito 5′ di splicing. È stato quindi piuttosto sorprendente trovare successivamente che un nuovo allele, prp8-101, blocca lo splicing al secondo passaggio. Altri alleli di Prp8 influenzano la selezione del sito 3′ di splicing. James G. Umen e Christine Guthrie hanno inoltre dimostrato che Prp8 può formare legami crociati con sequenze prossime al sito 3′ di splicing. Quindi questa grande proteina può mediare entrambi i passaggi dello splicing e può essere localizzata vicino al centro attivo dello spliceosoma. Una possibilità è che essa serva come stampo per assicurare la corretta conformazione della struttura degli RNA nello spliceosoma, specialmente l'interazione dell'RNA U5 con entrambi i siti di splicing (fig. 6). Se così fosse, è possibile che il meccanismo di splicing, all'inizio completamente indipendente da proteine, come nel caso degli introni del gruppo II, abbia subito un'evoluzione, divenendo successivamente dipendente da proteine, come imposto al sistema dalla grande diversità di introni che devono essere eliminati.
Bibliografia
Amrein 1994: Amrein, Hubert - Hedley, Mary Lynne - Maniatis, Tom, The role of specific protein-RNA and protein-protein interactions in positive and negative control of pre-mRNA splicing by Transformer 2, "Cell", 76, 1994, pp. 735-746.
Boggs 1987: Boggs, Russell T. e altri, Regulation of sexual differentiation in D. melanogaster via alternative splicing of RNA from transformer gene, "Cell", 50, 1987, pp. 739-747.
Culver 1993: Culver, Gloria M. e altri, An NAD derivative produced during transfer RNA splicing: ADP-ribose 1*2* cyclic phosphate, "Science", 261, 1993, pp. 206-208.
Fabrizio 1989: Fabrizio, Patrizia - McPheeters, David S. - Abelson, John, In vitro assembly of yeast U6 snRNP: a functional assay, "Genes and development", 3, 1989, pp. 2137-2150.
Ge 1991: Ge, Hui - Zuo, Peng - Manley, James L., Primary structure of the human splicing factor ASF reveals similarities with Drosophila regulators, "Cell", 66, 1991, pp. 373-382.
Goralski 1989: Goralski, Thomas I. - Edström, Jan Erik - Baker, Bruce S., The sex determination locus transformer-2 of Drosophila encodes a polypeptide with similarity to RNA binding proteins, "Cell", 56, 1989, pp. 1011-1018.
Herschlag 1991: Herschlag, Daniel - Piccirilli, Joseph A. - Cech, Thomas R., Ribozyme-catalyzed and nonenzymatic reactions of phosphate diesters: rate effects upon substitution of sulfur for a nonbridging phosphoryl oxygen atom, "Biochemistry", 30, 1991, pp. 4844-4854.
Krainer 1991: Krainer, Adrian R. e altri, Functional expression of cloned human splicing factor SF2: homology to RNA-binding proteins, U1 70K, and Drosophila splicing regulators, "Cell", 66, 1991, pp. 383-394.
Krawczak 1992: Krawczak, Michael - Reiss, Jeffrey - Cooper, David N., The mutational spectrum of single base-pair substitutions in mRNA splice junctions of human genes: causes and consequences, "Human genetics", 90, 1992, pp. 41-54.
Madhani 1990: Madhani, Hiten D. - Bordonné, Rémy - Guthrie, Christine, Multiple roles for U6 snRNA in the splicing pathway, "Genes and development", 4, 1990, pp. 2264-2277.
McSwiggen, Cech 1989: McSwiggen, James A. - Cech, Thomas R., Stereochemistry of RNA cleavage by the Tetrahymena ribozyme and evidence that the chemical step is not rate-limiting, "Science", 244, 1989, pp. 679-683.
Mount 1996: Mount, Stephen M., AT-AC introns: an ATtACk on dogma, "Science", 271, 1996, pp. 1690-1692.
Peebles 1986: Peebles, Craig L. e altri, A self-splicing RNA excises an intron lariat, "Cell", 44, 1986, pp. 213-223.
Reyes 1996: Reyes, José e altri, The canonical GU dinucleotide at the 5′ splice site is recognized by p220 of the U5 snRNP within the spliceosome, "RNA", 2, 1996, pp. 213-225.
Suh, Waring 1992: Suh, Eun Ran - Waring, Richard B., A phosphorothioate at the 3′ splice site inhibits the second splicing step in a group I intron, "Nucleic acids research", 20, 1992, pp. 6303-6309.
Teigelkamp 1995: Teigelkamp, Stefan - Newman, Andrew I. - Beggs, James D., Extensive interactions of Prp8 protein with the 5′ and 3′ splice sites during splicing suggest a role in stabilization of exon alignment by U5 snRNA, "EMBO journal", 14, 1995, pp. 2602-2612.
Umen, Guthrie 1995: Umen, James G. - Guthrie, Christine, Prp16p, Slu7p, and Prp8p interact with the 3′ splice site in two distinct stages during the second catalytic step of pre-mRNA splicing, "RNA", 1, 1995, pp. 584-597.
Umen, Guthrie 1995: Umen, James G. - Guthrie, Christine, The second catalytic step of pre-mRNA splicing, "RNA", 1, 1995, 869-885.
Umen, Guthrie 1996: Umen, James G. - Guthrie, Christine, Mutagenesis of the yeast gene Prp8 reveals domains governing the specificity and fidelity of 3′ splice site selection, "Genetics", 143, 1996, pp. 723-739.
Van der Veen 1986: van der Veen, Renske e altri, Excised group II introns in yeast mitochondria are lariats and can be formed by self-splicing in vitro, "Cell", 44, 1986, pp. 225-234.
Yean, Lin 1991: Yean, Shyue-Lee - Lin, Ren-Jang, U4 small nuclear RNA dissociates from a yeast spliceosome and does not participate in the subsequent splicing reaction, "Molecular and cellular biology", 11, 1991, pp. 5571-5577.