BIOTECNOLOGIA
Biotecnologia
(App. V, i, p. 384)
Le b., intese nel significato più ampio e moderno del termine, possono essere definite come le tecnologie che utilizzano organismi viventi o loro componenti subcellulari, al fine di ottenere la produzione di sostanze utili in quantità industriali. In particolare le b. innovative sono basate sull'uso del DNA ricombinante, sulla fusione cellulare e sul trasferimento dei geni; sono essenzialmente il risultato dell'applicazione delle conoscenze acquisite dalla biologia cellulare e molecolare, dalla genetica molecolare, dalla biochimica, dall'elettronica e dall'ingegneria, e sono ben rappresentate dall'ingegneria genetica. Esse hanno già fortemente modificato, e si prevede che modificheranno ancora di più nel prossimo futuro, il modo di produrre e di fare ricerca in parecchi importanti settori produttivi.
La caratteristica principale delle b. avanzate è la possibilità di analizzare, modificare in modo predeterminato e controllare l'agente biologico utilizzato. Altri fattori, di volta in volta importanti, sono le tecniche di conduzione e ottimizzazione dei processi, la strumentazione analitica, la disponibilità di bioreattori adeguati.
Le b. non sono di per se stesse un settore produttivo, ma al contrario sono dei mezzi che possono essere utilizzati in molti di tali settori (agricoltura, industrie farmaceutica, alimentare e chimica, protezione dell'ambiente, informatica ecc.). Esse possono essere applicate sia come mezzo di produzione che come strumento di ricerca: in altre parole possono originare nuovi prodotti o produrre in modo competitivo prodotti già noti, oppure rendere possibile la produzione industriale di sostanze note ma non disponibili, come, per es., le proteine umane o altre sostanze complesse.
Negli ultimi anni le b. hanno ricevuto un notevole impulso, sia dalle nuove conoscenze in campo biologico, sia dallo sviluppo e dall'ottimizzazione di tecniche che, in parte, rappresentano la naturale evoluzione di tecnologie applicate alla biologia molecolare e, in parte, costituiscono un capitolo del tutto nuovo nella storia di questo settore. Processi o prodotti derivati dall'impiego delle b. sono ormai utilizzati con grande successo in aree che vanno dalla nutrizione alla conservazione della variabilità biologica, dalla depurazione di ambienti contaminati ai modelli animali che simulano malattie umane, fino alla terapia genica di malattie quali il cancro o l'AIDS. Contemporaneamente, alcune discipline che prima rivestivano un ruolo marginale sono divenute strategiche per la ricerca biotecnologica: la bioinformatica, per es., è ormai uno strumento necessario per il progresso di queste ricerche e la mole di dati da immagazzinare, elaborare, confrontare, sta diventando così imponente da spingere organizzazioni transnazionali come l'Unione Europea a cercare di armonizzare le procedure e i prodotti informatici risultanti da questo settore.
Qui di seguito verranno sviluppati alcuni dei punti chiave delle b.: il primo è relativo alla reazione di amplificazione a catena del DNA, una tecnica che ha segnato gli anni Novanta, ma che sicuramente continuerà a influenzare la ricerca e le applicazioni della biologia anche nel prossimo secolo; gli altri tre riguardano, rispettivamente, la mappatura e il sequenziamento di genomi di eucarioti e di batteri; la terapia genica; e, infine, le b. per lo studio e il miglioramento delle risorse genetiche degli animali e delle piante.
Reazione di polimerizzazione a catena
L'area tecnologica che ha avuto il maggior impulso e che ha esercitato un'influenza determinante, tanto sulla ricerca di base quanto su quella applicata, rimane senza dubbio la tecnologia degli acidi nucleici, in quanto ha beneficiato dello sviluppo della tecnica chiamata reazione di polimerizzazione a catena (PCR, Polymerase Chain Reaction, v. anche biologia molecolare, App. V e in questa Appendice). La specificità della reazione, la possibilità di ottenere copie teoricamente infinite di un particolare gene - e quindi dei 'difetti' a esso associati - hanno trovato immediati riscontri in ogni settore delle b.: dalla diagnostica di laboratorio alla microbiologia, all'oncologia, alle malattie ereditarie, alla medicina legale, alla paleontologia ecc.
La PCR consiste nell'amplificazione enzimatica in vitro di una definita sequenza di DNA. Questa amplificazione viene ottenuta mediante cicli ripetuti di polimerizzazione di desossiribonucleotidi in cui il prodotto di reazione diventa il reagente dell'amplificazione successiva, rendendo così possibile che il ciclo si ripeta un numero n di volte. Per poter utilizzare questo metodo, è necessario conoscere la sequenza del DNA della regione interessata all'amplificazione, o almeno di due brevi tratti di DNA ai suoi estremi 3´ e 5´, allo scopo di sintetizzare due brevi sequenze di DNA complementari che servono da innesco o iniziatore (primer) della reazione di amplificazione.
Prima di descrivere in dettaglio la PCR, è importante ricordare che i legami idrogeno della molecola del DNA possono essere distrutti dal calore o da valori elevati di pH. Tuttavia, in seguito a raffreddamento o all'abbassamento del pH, i due filamenti tendono a riassociarsi o 'ibridare' l'uno con l'altro, tornando ad assumere la conformazione a doppia elica. Fisiologicamente, la separazione dei due filamenti della doppia elica del DNA avviene durante la sua replicazione. I desossiribonucleotidi liberi si appaiano in modo complementare nel momento in cui le due catene di DNA vengono separate, fungendo da stampo per la sintesi di filamenti complementari. La reazione di sintesi del DNA è catalizzata da un enzima, una DNA-polimerasi, scoperto nel 1955 da A. Kornberg. Le DNA-polimerasi possono allungare una corta sequenza di nucleotidi, il primer, legando in successione i desossinucleotidi trifosfati all'estremità 3´ del primer in crescita. Il primer si trova ovviamente già ibridato alla sequenza complementare del filamento che fa da stampo. Sfruttando questo principio, si ha la sintesi unidirezionale di DNA, una metodologia ampiamente usata per risolvere la sequenza del DNA dei geni finora identificati.
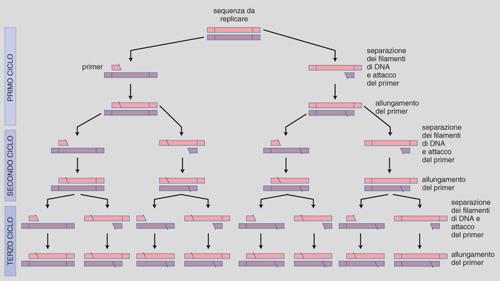
Alcuni anni fa, precisamente nel 1985, K. Mullis - allora biochimico presso un'azienda biotecnologica californiana, la Cetus Corporation - ebbe l'idea di usare un paio di primer, complementari a due sequenze nucleotidiche su ciascun filamento di DNA e delimitanti una regione genomica di interesse. Una reazione di questo genere porta alla sintesi, in direzioni opposte, di due molecole di DNA a doppia elica che, denaturate, possono poi diventare lo stampo per altri filamenti in una successione esponenziale. Questo è il semplice concetto che sta alla base della reazione di polimerizzazione a catena o PCR (fig. 1).
Un ciclo di amplificazione consiste schematicamente di tre fasi ed è preceduto da una fase in cui il DNA del campione in esame viene estratto e digerito con diversi enzimi di restrizione, secondo metodiche già descritte (v. biotecnologia, App. V). La prima fase prevede la denaturazione ad alta temperatura della doppia elica di DNA, allo scopo di separare i due filamenti. La seconda fase consiste nel legame dei primer, in precedenza aggiunti in eccesso alla miscela di reazione, alle sequenze di DNA a essi complementari. Il terzo stadio è rappresentato dalla sintesi del DNA a opera dell'enzima DNA-polimerasi. In virtù della loro alta concentrazione, i primer si legano rapidamente alle sequenze loro complementari anticipando, quando la temperatura viene abbassata, la loro riassociazione. A seguito di un successivo riscaldamento della miscela di reazione, anche le catene di DNA neosintetizzate si denaturano aprendosi: viene così reso disponibile l'attacco dei primer per la sintesi di altre catene complementari. Il ciclo quindi riprende nuovamente. In una reazione di PCR in cui la resa di amplificazione risulti ottimizzata si può raggiungere un'amplificazione di un fattore 10⁶, un milione di volte.
Le prime reazioni di PCR sono state sviluppate utilizzando la DNA-polimerasi estratta da Escherichia coli. Tuttavia, durante la fase di denaturazione ad alta temperatura la polimerasi veniva inattivata ed era perciò necessario aggiungere nuovo enzima all'inizio del successivo ciclo di amplificazione: il che rendeva il metodo difficoltoso e soggetto a errori. Attualmente viene invece impiegata una DNA-polimerasi termostabile, che resiste cioè fino a temperature di 95 °C, purificata dal batterio Thermus aquaticus, un microrganismo che vive normalmente in sorgenti calde a temperature tra 70 e 75 °C (PCR technology 1989). Con l'introduzione di questo enzima è stato possibile evitare di aggiungere continuamente enzima alla miscela di reazione e, cosa più importante, automatizzare completamente le varie fasi della PCR. Essa infatti viene ora allestita mediante appositi strumenti programmabili, in grado di variare opportunamente le temperature di reazione all'interno della microprovetta contenente tutti i reagenti necessari al mantenimento della reazione.
Il prodotto finale della reazione di polimerizzazione a catena è una certa quantità di DNA specificamente amplificato, che deve però essere evidenziato e distinto dal DNA non amplificato. A tale scopo si usano varie tecniche: la più comune risulta essere una separazione dei frammenti mediante elettroforesi in gel di agarosio, a cui fa seguito una colorazione specifica per il DNA che evidenzia la banda del prodotto di PCR, in genere molto ben visibile. Questo metodo di visualizzazione può essere integrato o sostituito dall'uso di ibridazioni secondarie con sonde specifiche marcate con la radioattività o con fluorofori.
Uno dei campi di applicazione più importanti, sia dal punto di vista clinico-epidemiologico che di ricerca, è la microbiologia: la tecnica può essere infatti usata per l'identificazione, in un campione di materiale infetto, di un qualsiasi patogeno di cui sia conosciuta anche solo una limitata porzione della sequenza genomica. Sono virtualmente analizzabili mediante PCR tutte le malattie genetiche ereditarie di cui sia stato identificato il locus genetico implicato, quali, per es., le varie forme di talassemia, di emofilia, la distrofia muscolare di Duchenne e altre. La possibilità di rilevare il DNA anche con quantità limitate di materiale cellulare ha conferito un ruolo di particolare rilievo a questa tecnologia nella diagnostica prenatale: in questo caso sono sottoposte a PCR cellule fetali ottenute da amniocentesi o campioni di villi coriali. La risoluzione della sequenza di regioni di geni amplificati con PCR ha poi consentito di individuare malattie associate a variazioni di singoli nucleotidi nei geni che codificano per gli antigeni di istocompatibilità.
Un settore che si è di recente avvalso della tecnica della PCR è quello della medicina legale, specialmente nei casi in cui i reperti contengano solo pochissime cellule: è possibile la tipizzazione del patrimonio genetico da capelli, spermatozoi e cellule del sedimento urinario (Erlich, Gelfand, Sninsky 1991). La PCR ha permesso di conseguire rilevanti risultati anche nel campo dell'oncologia. L'identificazione di diversi elementi genetici, come oncogeni (v. App. V e in questa Appendice) e geni oncosoppressori, fattori di crescita e loro recettori, ne ha chiarito il ruolo in relazione alla genesi e all'evoluzione delle malattie neoplastiche, rendendo possibile un approccio molecolare all'oncologia. Le principali applicazioni in questo ambito, che rendono unica la PCR come strumento diagnostico, includono sia la possibilità di identificare minimi cambiamenti strutturali, mutazioni puntiformi o riassetti, sia la capacità di individuare la quota minima di malattia residua, cioè le cellule tumorali resistenti alla chemioterapia (Eisenstein 1990).
Analisi di genomi
Lo spettacolare sviluppo delle tecniche di biologia molecolare ha aperto ampie prospettive nella direzione di uno dei grandi sogni della biologia moderna, vale a dire l'analisi dei genomi degli esseri viventi (v. anche progetto genoma, App. V e in questa Appendice).
Verso la metà degli anni Ottanta, la comunità scientifica aveva iniziato a pianificare una serie di progetti riguardanti il sequenziamento sistematico di genomi, con lo scopo di allestire uno strumento in grado di portare la ricerca biologica in ambiti ancora inesplorati. Tra i programmi più ambiziosi figurava la mappatura e il sequenziamento del genoma di Homo sapiens, e tale progetto ha in effetti fornito la prima opportunità per una dettagliata discussione sulle implicazioni strategiche di un'iniziativa di dimensioni così grandi, ponendo altresì una serie di problemi di sviluppo tecnologico e di collaborazione internazionale per un'impresa paragonabile al programma spaziale culminato, sul finire degli anni Sessanta, con la missione dell'uomo sulla Luna. Fu subito chiaro che i progetti di mappatura e sequenziamento dei genomi, considerando i costi, la complessità, la necessità di multidisciplinarietà e le ricadute di amplissimo respiro, non potevano essere affrontati da una sola istituzione, per quanto di grande dimensione e di alto livello di organizzazione; era invece necessaria una collaborazione mondiale che prevedesse lo sviluppo di reti di laboratori interconnessi e coordinati da centri, in grado di raccogliere tutte le informazioni messe a disposizione dai singoli laboratori. Qualunque genoma si intenda sequenziare, per la messa a punto del programma di sequenziamento sono infatti necessari due strumenti: uno che renda possibile l'orientamento spaziale dei geni identificati; l'altro che consenta di assegnare a ogni tratto sequenziato la giusta collocazione all'interno del genoma in esame, cioè la mappa genetica e la mappa fisica.
La fine degli anni Novanta segna l'inizio dell'era 'genomica'. Per la prima volta nella storia è possibile conoscere l'intero contenuto genetico di un numero sempre crescente di organismi viventi, dall'uomo agli eucarioti, ai batteri, e aprire la strada a nuove e imprevedibili scoperte. Ciò vale anche per microrganismi come Hemophilus influentiae, Micoplasma pneumoniae e Helicobacter pylori, le cui sequenze genomiche sono uno strumento potente per studiare la biologia di questi batteri e per capire le basi genetiche della patogenicità e della virulenza (Strauss, Falkow 1997).
Sequenziamento del genoma di lievito. - Il primo cromosoma completamente sequenziato di un organismo eucariote è stato il cromosoma iii di Saccharomyces cervisiae, un ceppo di lievito che contiene 16 cromosomi per complessivi 15 milioni di paia di basi nucleotidiche. Questo risultato è stato pubblicato nel maggio del 1992 sulla rivista Nature, la stessa dove era comparso, nel 1953, il famoso articolo di J. Watson e F. Crick sulla struttura a doppia elica del DNA.
I 16 cromosomi che compongono il patrimonio genetico del lievito, nonostante le ridotte dimensioni, possiedono notevoli somiglianze con quelli degli eucarioti complessi, sia per la struttura che per i meccanismi di replicazione e segregazione. La facilità con cui possono essere manipolati utilizzando tecniche di genetica sia classica sia molecolare ha permesso di identificarne tutte le regioni funzionali, vale a dire telomeri, centromeri e origine di replicazione. L'impresa ha riguardato 35 laboratori (di cui 4 italiani) finanziati dall'Unione Europea, con un contratto di ricerca che vincolava ogni laboratorio a sequenziare almeno 10 kilobasi del cromosoma in questione. Un gruppo così numeroso andava coordinato in modo da uniformare le finalità scientifiche dei laboratori coinvolti, molti dei quali lavoravano su differenti geni del lievito sulla base di differenti interessi di ricerca. Questo modo di procedere - che, pur non prevedendo l'allestimento di grandi centri dedicati al sequenziamento di genomi, è risultato vincente per quel che riguarda la strategia - si basa principalmente sull'attività di coordinamento di due momenti essenziali del processo di sequenziamento: il coordinamento delle librerie genomiche, garantito da due cosiddetti DNA coordinator, che distribuiscono ai laboratori i pezzi di cromosoma da sequenziare e che possiedono la mappa fisica relativa a tali frammenti; la raccolta e l'immagazzinamento delle sequenze, garantiti da un centro di bioinformatica, cioè da un Sequence coordinator.
È in questo ambito che risulta di vitale importanza lo sviluppo della bioinformatica, che nella mappatura e nel sequenziamento dei genomi trova una delle applicazioni più adatte. Nel 1991 fu coniato il termine in silice per definire un terzo modo di studiare i meccanismi biologici, che completa quelli in vivo e in vitro e che è a questi complementare, soprattutto nei campi della genetica e della biologia molecolare. Mai come oggi la tecnologia dell'informazione basata sui chip al silicio è entrata a far parte in modo così importante della ricerca biologica, specie delle biotecnologie. Questo nuovo approccio permette l'acquisizione e il trattamento dei dati in differenti banche di sequenze specializzate, e può inoltre costituire l'interfaccia di collegamento con banche dati che immagazzinano differenti tipi di informazioni (fisiologia, metabolismo, biochimica ecc.). L'intero sequenziamento del genoma di lievito è stato completato nel 1996 ed è stato pubblicato sulla rivista Nature.
Mappatura del genoma di topo. - Nell'ottobre 1993 venne pubblicata sulla rivista Science la mappa genetica del topo, la prima relativa a un mammifero nella storia della biologia moderna. Il topo, dato il breve periodo di gestazione e le grandi possibilità di incrocio, è stato l'animale da laboratorio d'eccellenza anche per gli studi di genetica classica. Recentemente, sono state sviluppate tecnologie che consentono di inserire in cellule embrionali di topo geni umani, e questi possono in seguito essere espressi nel corso dello sviluppo e del differenziamento dell'animale; sono stati in tal modo creati animali transgenici, in cui possono essere studiate le funzioni di tali geni mediante mutazioni che possono inattivarli o aumentarne il grado di attività. Gli effetti di tali modificazioni sono particolarmente utili per studiare numerose patologie genetiche umane, l'autoimmunità e altri disturbi del sistema immunitario, alcune patologie neurologiche, le malattie congenite, alcuni tipi di tumore, malattie metaboliche come il diabete, l'arteriosclerosi e varie anomalie della riproduzione.
Come per la mappa genetica umana, la mappa genetica del topo serve per due scopi distinti. Innanzitutto, essa fornisce uno strumento per l'analisi genetica, e quindi - date le possibilità di manipolazione - permette la mappatura di mutazioni che causano caratteristiche fenotipiche particolarmente interessanti, la localizzazione cromosomica di geni già clonati e sequenziati e la creazione di animali genotipicamente ben definiti. In secondo luogo, l'allestimento delle mappe genetiche - che è uno stadio imprescindibile per poter poi sviluppare le mappe fisiche - fornisce le indicazioni necessarie per ordinare gli YAC (Yeast Artificial Chromosome, v. biologia molecolare, App. V e in questa Appendice) contenenti i vari segmenti contigui del genoma del topo. È stata quindi preparata una mappa genetica dei 21 cromosomi di topo basata su 2616 loci, portando il potere di definizione della mappa a circa 0,6 cM (il centiMorgan dà una stima della distanza tra un gene e un altro ed è basato sulla frequenza di ricombinazione cromosomica che si instaura al momento della meiosi). Dei 2616 loci, 917 mostrano omologia con geni umani.
Un settore che potrà trarre vantaggio da questi studi è sicuramente la ricerca sul cancro, in quanto da tempo è noto che una degenerazione genetica, a carico di determinate regioni cromosomiche, è responsabile dell'insorgere e della progressione della malattia tumorale. Queste regioni possono essere riconosciute in quanto i marcatori genetici nei tessuti normali risultano eterozigoti, mentre nei tessuti cancerosi si osserva l'instaurarsi dello stato omozigote. Questo rende il topo un modello ideale per studiare geneticamente il fenomeno dell'oncogenesi.
Mappatura del genoma umano. - Per dare un'idea dello sforzo scientifico necessario, basti pensare che il genoma umano è circa 200 volte più grande del genoma del lievito di birra, che rappresenta l'organismo eucariote più semplice. Il genoma umano è costituito da una sequenza di almeno 3 miliardi di coppie di basi. Se, a titolo esemplificativo, tale sequenza venisse stampata su una pagina come quella dell'elenco telefonico avremmo più di 1200 volumi di mille pagine ciascuno. Un primo passo per cercare di leggere questa collezione di libri consiste nell'allestire una mappa genetica, cioè assegnare ad alcuni geni una posizione, l'uno rispetto all'altro, sul cromosoma dove sono localizzati. Ciò, riprendendo l'esempio precedente, equivale ad allestire un indice parziale dei libri di cui si è detto poco sopra. La mappa genetica rappresenta quindi uno strumento per indirizzare il 'lettore' in una direzione o in un'altra, cosa indispensabile per il riconoscimento delle sequenze specifiche (Nucleic acid analysis, 1996).
Ovviamente, come per le carte geografiche, tali mappe possono essere a bassa o ad alta risoluzione e questo dipende dalla quantità di informazioni che si ha a disposizione. È chiaro che si intendono allestire mappe sempre più dettagliate, dapprima con una visione a bassa risoluzione, per passare poi gradualmente a valori di discriminazione sempre più elevati, fino a scorgere i dettagli reali del paesaggio, vale a dire a identificare i nucleotidi che fanno parte della sequenza di quel particolare gene. Le mappe genetiche costituiscono il sistema storicamente più vecchio di cartografia dei genomi, in quanto il loro allestimento può essere effettuato anche con tecniche di genetica classica. Gli elementi primari di una mappa genetica sono infatti difetti genetici, che possono essere studiati sul fenotipo osservando la frequenza di ricombinazione del gene in cui la mutazione si manifesta o di geni che non sono responsabili della malattia, ma che segregano assieme al gene della malattia (Dulbecco 1995). A questi marcatori può aggiungersi una serie di altri elementi che incrementano il potere di risoluzione della mappa. Questi elementi possono essere i cosiddetti RFLP (Restriction Fragment Length Polymorphism, v. biologia molecolare, App. V e in questa Appendice) o, secondo metodi più recenti, delle brevi sequenze caratterizzate da ripetizioni di citosina e adenina, o anche delle sequenze ripetitive di tre o quattro nucleotidi che sono sparse lungo tutto il genoma. Pur non essendo assegnabili ad alcun gene, queste sequenze costituiscono degli ottimi punti di riferimento per la mappatura.
Il limite a cui tende una mappa genetica è la cosiddetta mappa fisica, che consiste nell'ordinare nella giusta sequenza la 'libreria genomica' dell'organismo di interesse. Sulla base della mappa genetica, che costituisce l'ossatura, si allestisce una mappa fisica. Uno strumento sviluppato di recente per facilitare questo lavoro è certamente l'uso di piccoli tratti di DNA a sequenza nota, associati o meno a geni, che possono essere identificati mediante PCR. Il vantaggio è che questi tratti possono essere identificati sui vari pezzi di DNA genomico che compongono la libreria; il che consente quindi di ordinare i pezzi contigui con il corretto orientamento. Lo sviluppo delle b. e di sequenziatori sempre più veloci ed efficienti ha permesso di aumentare la velocità di mappatura dei geni: nel catalogo delle malattie ereditarie umane compilato da V.A. McKusick, uno dei padri della genetica umana, dagli iniziali 1478 difetti genetici elencati nel 1966 si è giunti a 4937 nell'edizione 1990 (McKusick 1966, 1990⁹). Nel 1989, dopo almeno venti anni di ricerche di genetica sia classica sia molecolare, venne identificato e sequenziato il gene per la fibrosi cistica: passo dopo passo, dapprima 'camminando' sul cromosoma e poi passando al setaccio, mediante sonde a DNA, la zona che si riteneva contenere il gene, si raggiunse finalmente l'obiettivo. Se all'epoca fosse stata disponibile la sequenza del genoma umano, il processo di identificazione sarebbe stato di almeno dieci volte più veloce. La mappatura e la sequenza del genoma umano possono in effetti essere considerati 'il vero cuore della biologia e della medicina' (Ross 1992, 1996²).
Per quanto riguarda il genoma umano, nel settembre 1994 è stata pubblicata, sulla rivista Science, una mappa genetica ad alta risoluzione della specie umana. L'importanza di questo risultato è enorme, in quanto, per la prima volta nella sua storia, la specie umana ha la capacità di capire il proprio assetto genetico e come questo contribuisca alla morbilità individuale e della specie stessa. I tentativi di associare le malattie umane al patrimonio genetico sono iniziati negli anni Trenta, con i primi studi di linkage genetico tra il cromosoma X e il daltonismo e l'emofilia. Un serio ostacolo a questi studi di linkage è rappresentato dalla poco numerosa progenie degli uomini e dalla grande eterogeneità dei loro accoppiamenti.
Negli anni Settanta, con lo sviluppo e l'utilizzo degli elaboratori elettronici, l'analisi del linkage per la specie umana divenne più rapido ed efficiente, anche se la scarsità di marcatori genetici ne limitava grandemente l'impiego. Fu negli anni Ottanta, grazie ai calcolatori sempre più potenti e all'avvento delle moderne b., che furono evidenziati molti RFLP nel DNA umano. Vennero poi evidenziati altri tipi di marcatori, come i VNTR (Variable Number of Tandem Repeats, v. biologia molecolare, App. V e in questa Appendice), chiamati anche minisatelliti, e sequenze ripetute di nucleotidi, chiamati microsatelliti. In pratica, questi vari tipi di marcatori - geni, RFLP, VNTR - possono essere considerati alla stregua dei segnalibri, che danno un'idea di come sono posizionati gli uni rispetto agli altri e in che direzione. Ovviamente, la distanza tra un marcatore e l'altro può essere più o meno grande e questo dà un'idea del grado di risoluzione della mappa. Più sono numerosi i loci maggiore è la definizione della mappa.
Nel 1994 l'uso sempre più frequente delle tecniche di PCR ha ulteriormente aumentato il numero di sequenze che etichettano una certa zona del DNA umano, facendolo quasi triplicare nel giro di pochi mesi, tanto che la mappa genetica umana è risultata composta da 5840 loci con un potere di risoluzione di circa 0,7 cM, un valore paragonabile a quello ottenuto nella mappa genetica del topo. Tale risultato è, tra l'altro, in linea con uno degli obiettivi che il Progetto genoma umano si era prefissato, vale a dire una mappa genetica a 2 cM, e una mappa fisica basata sull'utilizzo di STS (Sequence Tagged sites) situati dapprima a una distanza media di circa 300 kilobasi l'uno dall'altro, e successivamente di 100 kilobasi, valore questo che sembra accettabile per iniziare il sequenziamento vero e proprio. In questo ambito l'obiettivo primario è quello di mettere a punto tecnologie robotizzate di sequenziamento, che consentano di ottenere velocità annue di sequenziamento non inferiori a 100 milioni di basi, così da prevedere un sequenziamento completo all'inizio del terzo millennio. Per questa grande impresa, come si è visto, è cruciale il supporto bioinformatico; attualmente vi sono tre banche dati di primaria importanza per il Progetto genoma umano: la banca di sequenze di DNA (GenBank), la banca per le mappe cromosomiche (Genoma data base) e la banca di sequenza e struttura delle proteine (Protein information resource).
Terapia genica
La tecnologia del DNA ricombinante ha fornito gli strumenti per definire il ruolo di uno specifico prodotto genico nella patogenesi di alcune malattie umane. La possibilità di caratterizzare le patologie in termini molecolari ha quindi portato alla definizione di approcci terapeutici, basati sulla correzione del difetto genico responsabile della malattia. Questo è il concetto che sta alla base delle tecniche di terapia genica (v. App. V). Una certa patologia può essere trattata trasferendo materiale genetico all'interno di specifiche cellule del paziente, anziché utilizzare un trattamento farmacologico convenzionale che in molti casi non è disponibile. Questo trattamento può avvenire in due modi: ex vivo, cioè prelevando dal paziente le cellule da trattare e reinserendole a procedimento avvenuto, oppure in vivo, vale a dire intervenendo sulle cellule di un determinato distretto corporeo direttamente nell'area in cui si trovano. In ambedue i casi è comunque necessario veicolare il DNA esogeno all'interno della cellula (fig. 2).
Attualmente esistono differenti metodi per trasferire geni in cellule bersaglio e sono in corso studi preclinici e clinici di alcune patologie umane, che vanno dalle malattie del sistema ematopoietico al cancro e all'AIDS. Questi sistemi possono essere raggruppati in due grandi categorie: i sistemi virali e quelli non virali. Nei sistemi virali le sequenze del DNA sono trasferite utilizzando come vettori alcuni virus. I vettori retrovirali e i vettori associati ad adenovirus, nei quali il DNA esogeno è integrato nel DNA cromosomico delle cellule bersaglio, vengono impiegati soprattutto nella terapia genica ex vivo. La maggior parte dei metodi non virali di trasferimento genico sfrutta i meccanismi che le cellule di mammifero usano per incorporare le macromolecole. Attualmente l'interesse è centrato su metodi che utilizzano i processi di endocitosi mediati da recettori, in quanto permettono l'entrata del DNA nelle cellule bersaglio in vivo; in questo modo il DNA esogeno viene coniugato a un polipeptide riconosciuto da uno specifico recettore di membrana, che lega il complesso DNA-polipeptide portandolo all'interno della cellula.
Nel 1990 alcuni ricercatori dei National Institutes of Health negli Stati Uniti allestirono il primo intervento di terapia genica su un essere umano: nel caso specifico si trattava di una bimba di quattro anni affetta da deficienza di adenosindeaminasi (ADA), una patologia ereditaria causata da una mutazione del gene preposto alla codificazione di detto enzima, il cui risultato finale è un'immunodeficienza congenita che mina le difese immunitarie dell'individuo rendendolo suscettibile a severe e mortali forme di infezione. In questo caso l'inserimento del gene corretto venne allestito ex vivo, prelevando le cellule del sistema ematopoietico da midollo osseo e 'correggendole' in vitro, con successivo reimpianto nel paziente. Da allora, altri pazienti in età pediatrica affetti da questa deficienza hanno beneficiato del trattamento che ha restituito loro la capacità di vivere una vita normale.
In effetti, molti degli sforzi e delle conoscenze finora acquisite nell'ambito dell'applicazione clinica della terapia genica si sono focalizzati sulle cellule del sistema ematopoietico, che meglio si prestano a essere geneticamente curate al di fuori del corpo del paziente per poi esservi reintrodotte (Anderson 1992). La difficoltà maggiore è quella relativa al trasferimento genico in cellule staminali midollari - le uniche che possono garantire la correzione permanente del difetto genetico -, che sono presenti in scarso numero nel midollo osseo e non sono facilmente riconoscibili con gli attuali metodi analitici. Vi è inoltre una problematica connessa all'efficienza di trasfezione mediante vettori retrovirali che, come si è appena detto, necessitano di cellule in proliferazione, mentre le cellule staminali midollari sono in gran parte quiescienti. Per quanto riguarda il trattamento genico di epatociti, vi sono evidenze sperimentali relative a una malattia ereditaria, l'ipercolesterolemia familiare, causata da un difetto del gene che codifica per il recettore delle lipoproteine a bassa densità (LDL, Low Density Lipoprotein). In questo caso sono state utilizzate tecniche sia ex vivo che in vivo e sono stati sperimentati vettori retrovirali e adenovirali per il trasferimento dei geni negli epatociti. Relativamente al polmone, sono da segnalare le sperimentazioni in atto per la cura genetica della fibrosi cistica, una delle malattie ereditarie a più alta incidenza nella popolazione caucasica, causata da difetti del gene che codifica per la proteina CFTR (Cystic Fibrosis Transmembrane Conductance Regulator), la quale controlla il flusso di ioni cloro attraverso la membrana cellulare. La malattia si manifesta con un'ipersecrezione di muco a livello polmonare e le cellule bersaglio della terapia genica risultano quindi essere le cellule dell'epitelio polmonare. In questo caso, data la difficoltà di prelievo e reimpianto di queste cellule, la metodologia di elezione risulta essere quella in vivo. Studi su modelli animali hanno dimostrato l'utilità di vettori adenovirali e di liposomi per trasferire il gene corretto nelle cellule polmonari. Tuttavia, sono in corso altri studi, in quanto l'espressione della proteina è transitoria, con una durata che va da una settimana a quaranta giorni circa (Rosenfeld et al. 1992).
Biotecnologie in agricoltura
Negli ultimi cento anni il miglioramento vegetale è avvenuto secondo procedimenti scientifici. Con l'avvento delle tecniche del DNA ricombinante si possono, sempre senza incrocio, trasferire geni singoli da una pianta all'altra. La ricerca più conosciuta è quella realizzata da un gruppo di biologi americani, che è riuscito a trasferire un gene da un fagiolo al girasole, cioè da una leguminosa a una composita, sintetizzando un complesso cui nessun meccanismo naturale avrebbe potuto condurre e ottenendo come risultato la produzione di olio ad alto contenuto proteico (Cernia, Degen 1995). I traguardi, in parte già raggiunti e in parte ancora allo studio, riguardano soprattutto la protezione delle piante e l'aumento della loro produttività. Gli esperimenti di ingegneria genetica sono cioè rivolti a conferire ai prodotti agricoli proprietà desiderabili, quali miglioramento in quantità e qualità nutrizionali del prodotto, resistenza agli stress ambientali, ai parassiti e agli altri patogeni. Oltre a favorire l'aumento della tolleranza alla siccità e alla salinità dell'acqua, si cerca di aumentare la resistenza alle basse temperature. Altri esperimenti riguardano lo studio di particolari batteri, modificati dall'ingegneria genetica, che, distribuiti sul terreno o sui semi, incrementeranno la produttività delle piante, potenziandone l'assunzione dei metaboliti presenti nel terreno. Particolare successo hanno avuto gli esperimenti per l'inserimento nelle piante di geni di origine batterica, che conferiscono la resistenza ai parassiti o ad altri agenti patogeni, quali funghi e virus. A tutt'oggi sono state prodotte numerose piante transgeniche, cioè piante in cui sono stati inseriti geni provenienti da altre specie: mais, frumento, riso, pomodori, patate, lattuga, cotone, soia, piselli, carote, verze, cocomeri, fragole, girasoli, barbabietole, papaie, kiwi, melanzane, pere, mele, uva, asparagi e altre.
La tecnica usata è quella classica, già descritta, e consta di vari passaggi: 1) l'isolamento del gene che si vuole trasferire, separandolo dal restante DNA mediante un enzima di restrizione; 2) l'inserimento del gene isolato in un vettore molecolare (per es., un plasmide batterico); 3) la replicazione del batterio in modo d'avere l'amplificazione del gene da trasferire; 4) il trasferimento del plasmide, che veicola il gene 'passeggero', nella cellula germinale di un'altra specie vegetale, ottenendo così una nuova pianta dalle caratteristiche volute. Un vantaggio, per gli interventi di ingegneria genetica (v. in questa Appendice), è dato dal fatto che le cellule vegetali sono totipotenti (come cellule indifferenziate sono cioè in grado di generare un intero organismo), e quindi dalle cellule trasformate possono svilupparsi piante complete, in grado di riprodursi normalmente. Per l'ingegneria genetica, la maggior parte dei metodi si basa oggi su cellule di espianti, ottenute cioè da piccoli frammenti della pianta; tali frammenti possono essere ingegnerizzati e, successivamente, dare origine a una pianta intera con le nuove caratteristiche codificate dal gene esogeno inserito.
Le tecniche del DNA ricombinante hanno consentito di produrre piante transgeniche che resistono all'attacco degli insetti: più precisamente, sono state ottenute mediante l'inserimento di un gene del batterio Bacillus thuringiensis ampiamente diffuso nel terreno, che codifica per una proteina insetticida (denominata Bt). Quando le piante, così modificate, vengono aggredite dagli insetti nocivi, i danni causati alle foglie sono minimi, mentre le larve muoiono rapidamente. La tossina prodotta, mentre è altamente tossica per diversi insetti, è innocua per l'uomo, gli animali e altri insetti non nocivi. In effetti, quando gli insetti sensibili ingeriscono frammenti della pianta contenente la proteina di Bacillus thuringiensis la scindono a opera di una proteasi intestinale specifica che solo loro producono, per cui la molecola diventa tossica. Il gene per la tossina Bt è già stato introdotto in numerose specie di vegetali, compresi mais, pomodori e cotone, che hanno dimostrato nelle prove di campo risultati veramente impressionanti circa la resistenza all'aggressione degli insetti (Shah, Rommens, Beachy 1995).
Uno dei primi prodotti con queste caratteristiche, autorizzato per l'uso in diversi paesi del mondo, è rappresentato da un nuovo tipo di mais transgenico che appunto, grazie alla capacità di produrre nelle sue cellule la proteina Bt, è altamente resistente agli attacchi di un insetto devastatore chiamato 'piralide', il principale parassita del mais che danneggia ogni anno, a livello mondiale, milioni di ettari di messi con perdite nella produzione che possono raggiungere anche il 40%.
Piante transgeniche resistenti alle malattie infettive (virali, batteriche, fungine). - I risultati più promettenti si sono per ora ottenuti trasferendo la resistenza alle malattie virali. L'approccio prevede la produzione di piante che esprimono il gene virale che codifica per la proteina principale del guscio del virus (capside): verosimilmente, la presenza di questa proteina nella cellula vegetale interferisce con la penetrazione e la replicazione di virus infettanti nella cellula stessa. Sono state, per es., già prodotte piante transgeniche di pomodoro, di patata, di melone, di riso e di tabacco, che resistono alle infezioni da parte di specifici virus, quali il virus del mosaico alfa-alfa, il virus del mosaico del cocomero, il virus del mosaico del tabacco, il virus X e il virus Y della patata.
Piante transgeniche resistenti agli erbicidi. - Gli erbicidi trovano ampia applicazione nella distruzione delle erbe infestanti che, crescendo tra le piante agricole, possono ridurre l'entità del raccolto di oltre il 10%. D'altra parte, gli erbicidi non sono molto selettivi, in quanto agiscono alterando processi fisiologici caratteristici delle piante, quali la fotosintesi e la biosintesi di amminoacidi, e quindi, sebbene più attivi verso le erbe infestanti, danneggiano in parte anche le piante agricole. Sono state pertanto prodotte piante transgeniche resistenti a erbicidi, soprattutto a quelli a ridotto impatto ambientale (per es., glifosfato, glufosinato e bromoxinolo) che sono biodegradabili e quindi più ampiamente utilizzati. Tra queste sono da segnalare: cotone, mais, soia e barbabietola, ingegnerizzate mediante inserimenti di alcuni geni di origine batterica che codificano, per es., per enzimi capaci di inattivare gli erbicidi, mediante acetilazione o idrolisi nitrilica delle molecole tossiche.
Piante transgeniche per aumentare la conservabilità dei prodotti: l'esempio dei pomodori. - Nel pomodoro la maturazione, e quindi il colore rosso, è controllata dalla produzione nel frutto del gas etilene, che è a sua volta sotto controllo genico. La rapida maturazione di questo ortaggio può causare grossi problemi, se il prodotto deve essere trasportato in località molto lontane o se deve essere conservato senza possibilità di refrigerazione, come è il caso dei paesi del Terzo Mondo. Per evitare questo processo, sono state create delle piante di pomodoro transgeniche, nelle quali il nuovo gene espresso produce un RNA messaggero antisenso che blocca il RNA messaggero senso, deputato alla sintesi dell'enzima responsabile della biosintesi dell'etilene. In questo modo i pomodori possono restare acerbi anche per molti mesi sulla pianta, acquisendo però il contenuto e i sapori tipici del frutto maturo (accumulo degli zuccheri e degli acidi caratteristici). Quando poi si desidera farli maturare, è sufficiente pompare gas etilene nelle celle in cui sono conservati i pomodori verdi, che si trasformano in frutti rossi, morbidi e saporitissimi. Questa tecnica può essere anche impiegata con altri prodotti deperibili, quali banane e meloni.
Biotecnologie in zootecnia
I progressi dell'ingegneria genetica, intesi a modificare il genotipo animale, non sono stati limitati dalle norme etiche, giustamente imposte alle sperimentazioni, che prevedono invece la manipolazione di geni umani. È stato così possibile ottenere la clonazione (v. in questa Appendice), cioè la produzione di individui tutti identici tra loro in quanto generati dalla stessa cellula progenitrice e, addirittura, introdurre e fare funzionare 'nuovi geni' nelle uova fertili di alcuni animali, producendo così soggetti transgenici (Poli 1997). La finalità di tutti questi interventi per la produzione di animali clonati o di animali transgenici è quella di ampliare il numero di soggetti con migliori caratteristiche produttive e di resistenza alle malattie. La clonazione avviene, naturalmente o sperimentalmente, nel mondo batterico e anche vegetale, dove la propagazione asessuata è spesso più diffusa di quella sessuata.
Al contrario, gli animali superiori non si riproducono asessualmente e, pertanto, per 'clonare un animale' è necessario asportare chirurgicamente i nuclei di cellule totipotenti di un embrione nelle primissime fasi di sviluppo e inserirle in uova denucleate. Esperimenti di questo tipo sono stati condotti inizialmente su embrioni di rane e, successivamente, è stato addirittura possibile ottenere un clone di rane identiche tra loro utilizzando nuclei di cellule di un individuo già sviluppato e inserendoli in altrettante cellule uovo cui era stato asportato il nucleo. Tali esperimenti hanno dimostrato che i nuclei trapiantati da cellule di rane già sviluppate sono ancora totipotenti e che, quindi, ognuno di loro può dare origine, dopo il trapianto, a una rana adulta.
Dopo questo primo esperimento, l'attenzione è stata rivolta alla possibilità di clonare anche i Mammiferi: alla base di questo tipo di intervento, che però non prevede tecniche di ingegneria genetica, vi è la tecnologia del cosiddetto embryotransfer, o trasferimento embrionale, nell'utero di una femmina ricevente. L'approccio più valido è stato quello di utilizzare blastomeri totipotenti di un embrione allo stadio di morula, che possono essere separati e introdotti in altrettante uova non fertili: si sono ottenuti così numerosi embrioni identici che, trasferiti nell'utero di animali riceventi, hanno dato origine a soggetti identici. I primi risultati di questi esperimenti sono stati soddisfacenti: è stato infatti possibile produrre capretti perfettamente identici, cioè geneticamente tutti uguali e, successivamente, anche bovini identici, con la finalità di amplificare i soggetti con le caratteristiche genetiche desiderate, ossia maggior produttività e qualità, oppure maggiore resistenza alle malattie infettive, infestive o neoplastiche.
È pertanto opportuno sottolineare che la produzione di Mammiferi perfettamente identici tra loro, mediante la tecnica della separazione di cellule totipotenti da un aggregato di cellule embrionali, non dovrebbe essere definita clonazione: è vero infatti che tutti gli animali ottenuti sono identici tra di loro, ma essi sono diversi sia dal soggetto che ha fornito la cellula uovo sia dal soggetto che ha fornito lo spermatozoo per ottenere la cellula fecondata, da cui sono poi state ottenute le cellule utilizzate per generare gli animali fotocopia. Pertanto, un intervento di questo tipo non consente di conoscere a priori le caratteristiche, sia fenotipiche sia genotipiche, del cosiddetto clone di soggetti che si ottiene a partire da una cellula fecondata, che ha ricevuto una serie di cromosomi della madre (cellula uovo) e una serie di cromosomi del padre (spermatozoo).
Come detto, la clonazione di animali, intesa nel senso più stretto del termine, con la finalità cioè di ottenere una popolazione di individui geneticamente identici alle singole cellule (e quindi al soggetto) da cui sono derivati, e di cui si vogliono avere copie identiche, prevede il prelievo di nuclei di cellule del soggetto che si vuole clonare (cioè di cui si vogliono ottenere dei perfetti multipli), nuclei che vengono inseriti in altrettante cellule uovo denucleate le quali, opportunamente incubate o inserite nell'utero di altrettante femmine riceventi, potranno dare origine a individui perfettamente identici.
Dopo i già citati successi ottenuti esclusivamente nelle rane e gli innumerevoli insuccessi di tale applicazione agli animali (soprattutto animali da laboratorio: topi e ratti), I. Wilmut e i suoi collaboratori, nel febbraio 1997, presso il Centro di ricerca del Roslin Institute di Edimburgo hanno ottenuto una pecora 'clone', derivata a partire da una singola cellula somatica (tessuto mammario), prelevata da una pecora adulta, il cui nucleo è stato trasferito in una cellula uovo denucleata, impiantata poi nell'utero di una pecora ricevente. Dopo i previsti 5 mesi di gestazione tipici di questa specie, e più precisamente dopo 148 giorni, è nata Dolly, che ha dimostrato di avere lo stesso genotipo della pecora donatrice della cellula somatica, diverso da quello della madre nel cui utero si è sviluppata.
È opportuno precisare che per ottenere questo successo sono state prodotte ben 277 cellule uovo con inserito il nuovo nucleo e che di tutte queste solo 29 sono sopravvissute in vitro per qualche giorno e quindi sono state impiantate nell'utero di 13 pecore riceventi (nell'utero di un animale si possono impiantare più ovociti): solo una di queste 13 pecore è rimasta gravida e ha dato alla luce Dolly (Wilmut, Schnieke, McWhire et al. 1997).
Animali transgenici. - Per quanto riguarda invece la tecnologia che consente di produrre animali transgenici, l'intervento prevede che uno o più geni estranei vengano inseriti nel nucleo di ovociti fecondati: è così possibile produrre animali con caratteristiche 'nuove', che mai avrebbero potuto acquisire con tecniche naturali.
L'interesse per gli animali transgenici è indirizzato a produrre linee genetiche controllate di animali da reddito, che producano alimenti migliori e in maggiore quantità (carne, latte, uova). Per es., sono stati prodotti suini transgenici che crescono più rapidamente, utilizzano meglio l'alimento e producono una carne con meno grasso. Le ricerche più avanzate in questo settore riguardano, inoltre, il trasferimento di geni che facciano esprimere ad alta efficienza negli animali l'ormone della crescita, gli enzimi metabolici, alcune proteine del latte, e inoltre quelle molecole/recettori delle cellule del sistema immunitario deputati alle difese dell'organismo; in quest'ultimo caso, la finalità è, quindi, quella di produrre linee di animali maggiormente resistenti alle malattie batteriche, virali, parassitarie o neoplastiche (fig. 3).
Vi è, per es., la possibilità di produrre bovini transgenici nei quali è sovraespresso il gene della K-caseina, in modo da avere una maggiore produzione di formaggio dal latte, in quanto questa trasformazione è direttamente proporzionale al contenuto di K-caseina nel latte. Un altro possibile esempio è rappresentato dal trasferimento, sempre nel bovino, del gene dell'enzima lattasi in modo da farlo esprimere a livello della ghiandola mammaria: ne consegue la produzione di un latte privo di lattosio (in quanto metabolizzato dal citato enzima). Un latte con queste caratteristiche sarebbe ideale per tutte quelle persone che non possono consumare il latte tradizionale in quanto sono intolleranti al lattosio e non riescono a digerire il latte e i cibi contenenti il latte.
È infine da ricordare che, diversamente da altre specie animali da reddito, per le loro particolari caratteristiche del sistema di riproduzione e di sviluppo i pesci si prestano molto bene alle tecniche per la produzione di animali transgenici, anche perché dopo l'inserimento del gene nell'uovo fecondato non è necessario eseguire l'impianto in una femmina ricevente, in quanto sia la fecondazione sia lo sviluppo dell'uovo di pesce avvengono all'esterno dell'organismo. Sono già stati prodotti carpe, trote e salmoni transgenici che si sviluppano meglio e più rapidamente degli animali di controllo (non transgenici). Le ricerche sono ora orientate all'introduzione ed espressione nei pesci di acqua dolce o salata, sia di climi temperati che tropicali, dei geni che conferiscono maggiore resistenza alle malattie, oppure maggiore tolleranza agli stress climatici e ad altri fattori ambientali indesiderati.
bibliografia
V.A. McKusick, Mendelian inheritance in man, Baltimore (Md.), 1966, 1990⁹.
J.B.Gurdon, R.A. Laskey, O.R. Reeves, The development capacity of nuclei transplanted from keratinized skincells of adult frogs, in Journal of embriology and experimental morphology, 1975, 34, pp. 93-112.
PCR technology. Principles and applications for DNA amplification, ed. H.A. Erlich, New York 1989.
H.A. Erlich, D. Gelfand, J.J. Sninsky, Recent advances in polymerase chain reaction, in Science, 1991, 252, pp. 1643-51.
W.F. Anderson, Human gene therapy, in Science, 1992, 256, pp. 808-13.
M.A. Rosenfeld et al., In vivo transfer of the human cystic fibrosis transmembrane regulator gene, in Cell, 1992, 68, pp. 143-55.
D.W. Ross, Introduction to molecular medicine, New York, 1992, 1996².
E. Cernia, L. Degen, Le biotecnologie nel settore agroalimentare, Roma 1995.
R. Dulbecco, I geni e il nostro futuro, Milano 1995.
D.M. Shah, C.M.T. Rommens, R.N. Beachy, Resistance to diseases and insects in transgenic plants. Progress and applications to agriculture, in Trends in biotechnology, 1995, 13, pp. 362-68.
Nucleic acid analysis. Principles and bioapplications, ed. Ch.A. Dangler, New York 1996.
G. Poli, Biotecnologie. Principi e applicazioni dell'ingegneria genetica, Milano 1997.
E.J. Strauss, S. Falkow, Microbial pathogenesis. Genomics and beyond, in Science, 1997, 279, pp. 707-11.
I. Wilmut, A.E. Schnieke, J. McWhire et al., Viable offspring derived from fetal and adult mammalian cells, in Nature, 1997, 385, pp. 810-13.
D.G. Wong et al., Large scale identification, mapping and genotyping of polymorphism in the human genome, in Science, 1998, 280, pp. 1077-82.
R. Vishwanath, P.O. Brown, The transcriptional program of human fibroblasts, in Science, 1999, 283, pp. 83-7.