DNA
DNA
L'acido deossiribonucleico (DNA) costituisce, con l'acido ribonucleico (RNA), la classe di polimeri informazionali definita 'acidi nucleici', componenti fondamentali delle strutture viventi. Tra tutte le molecole biologiche soltanto gli acidi nucleici possiedono la potenzialità di autoduplicazione che permette la replicazione e la trascrizione dell'informazione chimica in essi contenuta. Altri polimeri costituenti fondamentali delle strutture biologiche, quali le proteine e i polisaccaridi, sono privi di tale capacità e, pur dotati di comparabile complessità polimerica strutturale e informazionale, di ricchissima informazione chimica e di grande flessibilità funzionale, non sono in grado di rivestire un ruolo genetico. Questo ruolo è legato alla particolare organizzazione strutturale degli acidi nucleici e alle proprietà chimiche, fisiche e topologiche sia dei suoi costituenti che delle macromolecole considerate nel loro insieme.
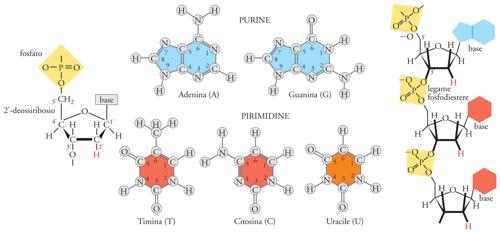
La struttura di base degli acidi nucleici è costituita dalla successione di monomeri, legati tra loro a formare polimeri che nei sistemi biologici attuali possono essere di dimensioni molto diverse: da poche unità fino a catene formate da centinaia di milioni di componenti. Il monomero fondamentale del DNA è costituito di tre elementi: uno zucchero, un gruppo fosfato, una base azotata. Le quattro basi presenti nel DNA sono due purine (Guanina e Adenina) e due pirimidine (Citosina e Timina), normalmente indicate con le loro iniziali G, A, C, T. L'informazione genetica è resa possibile dalla pressoché infinita possibilità combinatoria del susseguirsi delle quattro basi azotate. È inoltre importante considerare che il polimero nucleico è caratterizzato dal susseguirsi alternato e in sostanziale equilibrio di gruppi a carattere acido (i fosfati) e a carattere basico (le basi azotate).
Il DNA è una macromolecola instabile, ma la capacità delle basi nucleiche di formare legami a idrogeno conferisce al DNA la possibilità di stabilizzarsi in una struttura a doppia elica. L'interazione è ulteriormente stabilizzata e spazialmente organizzata dalla idrofobicità delle basi che in soluzione acquosa tendono a disporsi all'interno del doppio filamento, e dalla idrofilicità dei gruppi fosfato che si dispongono all'esterno. La forma più stabile di interazione è in forma di elica, come descritto da James Watson e Francis Crick nel loro celebre articolo pubblicato su "Nature" nel 1953. La doppia elica non è una struttura 'unica', ma può esistere in varie conformazioni, che variano per i loro parametri chimico-fisici. La doppia elica non è l'unica possibilità di organizzazione tridimensionale del DNA: sono state postulate e poi osservate anche strutture cruciformi, anisomorfe, ad ansa, a elica singola o a elica tripla e quadrupla. n
Le proprietà generali
L'eteropolimero è 'direzionale' e chimicamente 'instabile', sia nella componente costante che in quella variabile, ed è dotato di forte 'autocomplementarietà' speculare. La 'direzionalità' del DNA deriva dall'asimmetria della struttura dello zucchero (quattro atomi di carbonio all'interno della struttura ciclica, il quinto all'esterno) e dalla posizione del gruppo fosfato, legato all'atomo di carbonio 5′. La polimerizzazione avviene per formazione del legame fosfodiestere tra il fosfato legato in 5′ di un nucleotide e il carbonio 3′ di un altro nucleotide. Questa direzionalità strutturale e di sintesi 5′→3′ del DNA è rispettata nella direzionalità dell'informazione genetica in esso contenuta e nella direzionalità della sua lettura da parte degli enzimi deputati alla sua 'replicazione' (DNA-polimerasi) ed 'espressione trascrizionale' (RNA-polimerasi).
L'instabilità della catena polimerica è dovuta soprattutto alla possibilità di rottura del legame tra basi azotate e zucchero (il legame β-glicosidico) e di quello fosfoesterico tra zucchero e fosfato. Il ruolo genetico dei polimeri nucleici, legato alla possibilità di una loro sopravvivenza per tempi sufficientemente lunghi, è basato sull'azione stabilizzante esercitata da meccanismi di varia natura, i principali tra i quali sono l'autoprotezione, dovuta alla formazione di strutture secondarie sia nel DNA che nell'RNA, e l'interazione con proteine specifiche. L'instabilità chimicamente intrinseca del DNA non costituisce però soltanto un limite alla sua sopravvivenza in quanto molecola complessa; l'esistenza di meccanismi di riparazione sia a carico della parte variabile (le basi), sia a livello dello scheletro zucchero-fosfato è la chiave di fenomeni biologici fondamentali quali la mutazione e il loro controllo, la ricombinazione genetica, la trasposizione e, più in generale, l'evoluzione stessa.
Un ulteriore fattore intrinseco di instabilità è dovuto alla tautomeria delle basi. Nella Tav. I le basi sono rappresentate nelle loro forme più frequenti. Accanto a queste ne esistono altre strutturalmente alternative, presenti solo per una frazione di tempo molto breve. Nonostante la sussistenza statisticamente limitata, una forma tautomerica può essere fonte di mutazione, soprattutto se presente al momento della replicazione del polimero.
L'instabilità chimica fin qui descritta è tuttavia contrastata dalla capacità che il DNA possiede di stabilizzarsi in doppia elica, struttura resa possibile dalla formazione di legami idrogeno tra le basi. Un legame a idrogeno si forma tra un atomo di idrogeno covalentemente legato a un gruppo 'donatore' (come, per es., ‒OH oppure =N‒H) e una coppia di elettroni liberi di un gruppo 'accettore' (come O=C‒ oppure N≡). Questo legame, che possiede allo stesso tempo sia proprietà di interazione covalente che di interazione non covalente, è considerato un legame debole ed è altamente direzionale tendendo a costituire angoli di 180°. Tali sue caratteristiche e l'abbondanza nelle basi nucleiche di gruppi che possono formarlo fanno sì che due filamenti di DNA posti in prossimità tendano a interagire in modo stabile e dipendente dalla sequenza nucleotidica che li compone.
La struttura
L'Adenina forma due legami a idrogeno con la Timina, la Guanina ne forma tre con la Citosina. Questa specificità di interazione 'locale' determina la specificità di interazione 'globale' delle sequenze polimeriche. L'interazione avviene in modo stericamente compatibile tra una sequenza (per es., 5′ ATGCAATTGG 3′) e la sua complementare (3′ TACGTTAACC 5′) e darà luogo alla formazione di un doppio filamento stabilizzato, in questo caso, da ventiquattro legami a idrogeno, autocomplementare e, rispetto alla sequenza, speculare. L'interazione viene quindi stabilizzata e spazialmente organizzata dalla idrofobicità delle basi e dalla idrofilicità dei gruppi fosfato che si dispongono all'esterno. La forma più stabile di interazione è ‒ come si è detto ‒ in forma di elica, secondo quanto descritto da Watson e Crick.
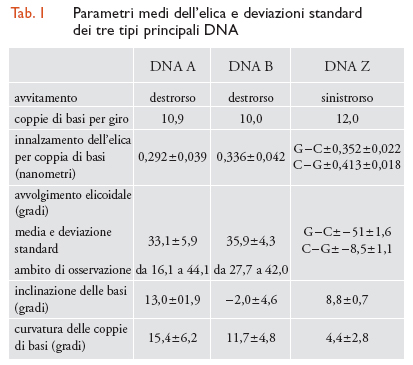
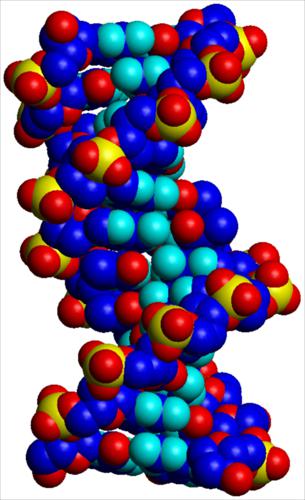
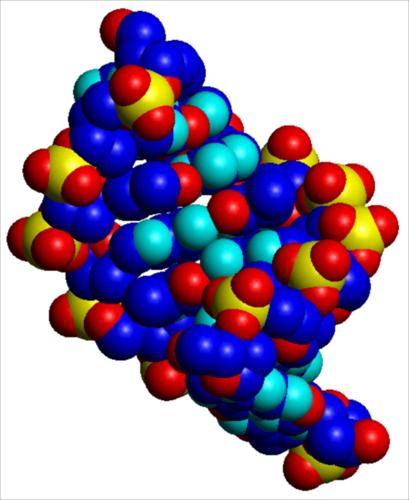
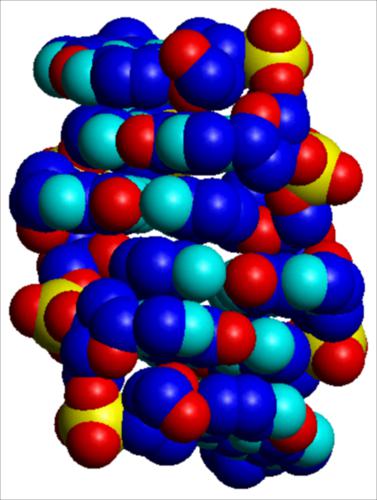
I parametri strutturali che caratterizzano la doppia elica sono riportati nella tab. 1. La fig. 2 A, B, C riproduce con modelli molecolari compatti le strutture di doppie eliche di DNA in conformazione B, e A. L'esistenza di conformazioni diverse è dovuta al fatto che il DNA ha un'organizzazione tridimensionale caratterizzata da estrema variabilità strutturale, sia locale che a lungo tratto, da flessibilità potenziale e differenziale in funzione della sequenza nucleotidica, da 'curvature' effettive caratterizzate da diversi gradi di rigidità, da forte dinamica, da capacità di cambiare conformazione in funzione sia dell'ambiente chimico-fisico nel quale è immerso, sia dell'interazione con altre macromolecole, segnatamente proteiche.
La struttura del DNA, così come definita originariamente nel modello della doppia elica Watson-Crick, è soltanto un modello astratto. Nella realtà il DNA oscilla punto per punto all'interno di un insieme di conformazioni alternative. Due di queste conformazioni sono riportate nella fig. 2. Altre conformazioni osservate e completamente diverse dalla doppia elica sono: strutture cruciformi, anisomorfe, ad ansa, a elica singola o a elica tripla e quadrupla. La doppia elica Watson-Crick è dunque semplicemente la struttura più stabile e più frequente all'interno di un vasto numero di possibilità alternative.
Un ulteriore livello di complessità strutturale e di ricchezza informazionale del DNA è quello 'topologico'. Nelle sue forme biologiche (cromosomi, episomi) il DNA non presenta estremità libere, soprattutto allo scopo di diminuire la possibilità della propria degradazione. Le estremità vengono evitate creando molecole circolari (come in moltissimi genomi virali), oppure evolvendo strutture terminali dette telomeri molecolarmente richiuse su sé stesse (come nei cromosomi), oppure legando sull'estremità della molecola di DNA singole proteine in modo covalente (come nel genoma di adenovirus). In ognuno di questi casi viene creato un 'sistema chiuso' all'interno del quale si instaurano proprietà topologiche. Una struttura topologicamente chiusa è in grado di accumulare energia torsionale. Questo fa sì che nel DNA il modo in cui un'elica si avvolge intorno all'altra può variare, aumentando o diminuendo la torsione locale. L'informazione topologica consiste essenzialmente nel modulare l'esposizione verso l'esterno (regolandone quindi l'accessibilità funzionale) dell'informazione genetica contenuta nella sequenza delle basi che, come mostrato nella fig. 2, sono normalmente confinate nella parte interna e più difficilmente accessibile della struttura.
I codici
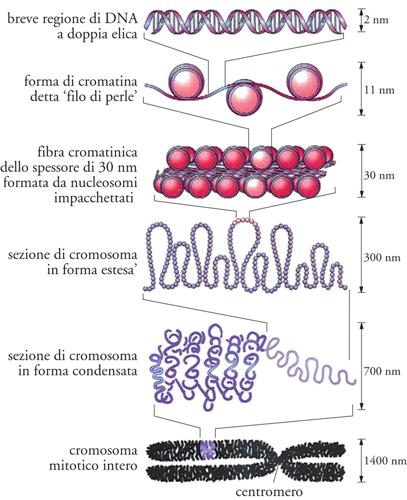
La determinazione della struttura della doppia elica ha immediatamente posto la domanda sulle modalità di codificazione dell'informazione biologica. La codificazione è basata sulla capacità di contenere (quasi) ogni immaginabile combinazione delle quattro basi A, T, G, C. È possibile quindi elaborare messaggi con un meccanismo digitale. L'informazione del DNA è dunque informazione primaria, determinata dalla sequenza nucleotidica. Il dizionario di lettere di DNA che codifica gli amminoacidi è definito 'codice genetico'. A questo codice se ne affiancano altri: il 'codice strutturale', che determina in quale conformazione il DNA sarà più stabile, ossia B, A, Z, cruciforme, ecc.; il 'codice topologico', che ne determina, tra l'altro, le proprietà di accessibilità locale; il codice epigenetico, dato dall'insieme di modificazioni chimiche delle basi della sequenza e dalle modificazioni chimiche delle proteine regolatrici che interagiscono con il DNA. Questo codice, la cui rilevanza nella regolazione dell'espressione dell'informazione contenuta nel DNA è in corso di studio, è in grado di modificare profondamente il comportamento del genoma, riguarda per definizione tutte le modificazioni genetiche che non comportano mutazione ed è sufficientemente stabile da trasmettersi attraverso le generazioni. Il 'codice regolativo' è costituito da sequenze nucleotidiche specifiche e complesse che regolano l'espressione e il silenziamento dei geni. Insieme al codice genetico sensu stricto, questo codice costituisce il 'codice digitale' del sistema informazionale del DNA. Infine la cromatina costituisce il 'codice cromatinico'. Dal momento che la complessità nucleotidica dei genomi è molto alta, l'ordinamento dal punto di vista fisico e funzionale di questo materiale ha portato all'evoluzione di un sistema di organizzazione detto 'cromatina' (fig. 3). Nel loro insieme, il codice strutturale, quello topologico e quello cromatinico costituiscono un insieme di informazioni definite 'analogiche'.
bibliografia
Calladine, Drew 1999: Calladine, Christopher R. - Drew, Horace R., Understanding DNA: the molecule and how it works, San Diego, Academic Press, 1999.
Hood, Galas 2003: Hood, Leroy - Galas, David, The digital code of DNA, "Nature", 421, 2003, pp. 444-449.
Watson, Crick 1953: Watson, James D. - Crick, Francis H.C., Molecular structure of nucleic acids. A structure of deoxyribose nucleic acid, "Nature", 171, 1953, pp. 737-738.
Wayt Gibbs 2003: Wayt Gibbs, W., The unseen genome: beyond DNA, "Scientific American", 289, 2003, pp. 78-85.