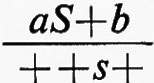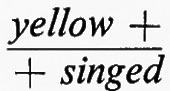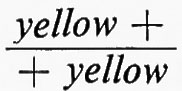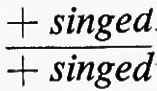Genetica
Genetica
di Harold Leslie K. Whitehouse, Claudio Barigozzi, Leslie C. Dunn, Giuseppe Montalenti
GENETICA
Genetica
di Harold Leslie K. Whitehouse
Sommario: 1. Introduzione. 2. Ereditarietà mendeliana: a) prove dell'ereditarietà mendeliana; b) la teoria cromosomica dell'ereditarietà mendeliana; c) associazione e scambio (Jinkage e crossing-over); d) crossing-over e chiasmi; e) l'ereditarietà dei caratteri quantitativi. 3. Ereditarietà non-mendeliana. 4. Problemi dell'ereditarietà mendeliana: a) alleli multipli; b) mutazioni; c) la natura del gene; d) modificazioni strutturali dei cromosomi; e) modalità d'azione del gene. 5. Conoscenze biochimiche e problemi dell'ereditarietà: a) le basi chimiche dell'ereditarietà; b) il processo di duplicazione del materiale genetico; c) sequenza dei nucleotidi e sequenza degli amminoacidi; d) il codice genetico. 6. Il controllo dell'attività genica e la ricombinazione genetica: a) il controllo dell'attività genica nei Batteri; b) il meccanismo della ricombinazione genetica; c) struttura e organizzazione del cromosoma; d) il controllo dell'attività genica negli organismi superiori. 7. Le basi dell'ereditarietà non-mendeliana. 8. Conclusioni. □ Bibliografia.
1. Introduzione
L'inizio del XX secolo costituisce, per una serie di coincidenze, una svolta decisiva nella storia del ramo della biologia che studia i fenomeni dell'ereditarietà e della variabilità, per il quale W. Bateson nel 1906 propose il nome genetica. Nei secoli precedenti si era infatti ritenuto, più o meno implicitamente, che i caratteri di un organismo fossero trasmessi direttamente agli individui della generazione successiva. Questi concetti trovarono riscontro nel pensiero di Ch. Darwin, il quale riteneva che dei ‛granuli' passassero da ciascun organo del corpo nelle cellule germinali, per essere così trasmessi alla generazione successiva. L'ereditarietà era pertanto diretta e questo comportava il concetto, generalmente associato al nome di J.-B. Lamarck, che i cambiamenti indotti in un individuo ad opera di fattori ambientali esterni, potessero diventare ereditari ed essere quindi trasmessi alla progenie. Anche se mancavano le basi sperimentali per convalidare questa teoria diretta dell'ereditarietà, non vi erano nemmeno delle prove conclusive per rifiutarla. Nell'anno 1900, H. De Vries, C. Correns e E. von Tschermak riuscirono, ciascuno indipendentemente dall'altro, ad ottenere queste prove e, nel consultare la bibliografia, si imbatterono nel lavoro originale di G. Mendel. Questo lavoro era stato pubblicato 34 anni prima, ma non aveva esercitato alcuna influenza sul pensiero scientifico dell'epoca.
La riscoperta del lavoro di Mendel costituisce una vera pietra miliare nella storia della genetica, poiché i suoi studi e quelli dei suoi riscopritori dimostravano che il meccanismo dell'ereditarietà non era quel semplice processo postulato da Darwin e dai suoi predecessori. In effetti, nel 1884, A. Weismann aveva suggerito con notevole acume che l'ereditarietà fosse determinata dagli appena scoperti ‛cromosomi' (che ricevettero questo nome qualche anno dopo) e non dagli stessi caratteri ereditari. Benché Weismann non avesse dati sperimentali per sostenere la sua ipotesi, questa rappresentò una rivoluzione nel pensiero riguardante i meccanismi ereditari: le sue idee contribuirono infatti a cambiare la concezione generale, con il risultato che la teoria di Mendel fu immediatamente accettata nel 1900, mentre era passata del tutto inosservata nel 1866.
La differenza fra il punto di vista mendeliano e quello darwiniano sull'ereditarietà è così profonda che forse non deve sorprendere il fatto che sia passato un terzo di secolo prima che le nuove idee venissero accettate. La teoria di Mendel sosteneva che l'ereditarietà era indiretta, cioè che non erano i caratteri stessi ad essere trasmessi, ma qualcos'altro, costituito da particelle discrete a cui successivamente W. Johannsen diede il nome di ‛geni'. Questi geni differivano sostanzialmente dai ‛granuli' di Darwin (che erano parte dello stesso carattere), in quanto i geni erano dei determinanti che a seconda delle circostanze potevano o meno condurre, durante la vita dell'individuo, alla comparsa del carattere in questione. Così una pianta di pisello (Pisum sativum) che alla sua origine avesse ricevuto il gene per cotiledoni gialli da un genitore e quello per cotiledoni verdi dall'altro (cioè un eterozigote per l'alternativa gialla nei confronti di quella verde), mostrava il carattere cotiledoni gialli. Mendel disse che il giallo era ‛dominante' e che il verde era ‛recessivo'. La presenza del gene per i cotiledoni verdi, tuttavia, si poteva osservare nuovamente nelle generazioni successive. Di converso, una pianta di pisello che avesse ricevuto il gene per cotiledoni verdi da entrambi i genitori (e cioè omozigote per questo carattere) sviluppava il pigmento verde. Evidentemente il gene per il pigmento verde non funzionava nella pianta eterozigote, o, se funzionava, il suo effetto era oscurato dall'allele (cioè dal gene corrispondente) per il pigmento giallo. In realtà Mendel non osservò direttamente i geni, ma ne postulò l'esistenza in base alla constatazione che le frequenze della progenie con uno dei due caratteri di una coppia alternativa (quale appunto il colore giallo dei cotiledoni nei confronti del colore verde) ricorrono secondo rapporti semplici, come 3:1 o 1:1.
La teoria indiretta mendeliana dell'ereditarietà implica una complessità ben maggiore nell'organizzazione degli organismi viventi che non la teoria darwiniana diretta: infatti, anzitutto, i geni mendeliani devono racchiudere in una qualche magica maniera un particolare carattere che poi specificheranno in qualche modo e, secondariamente, il gene per un particolare carattere può apparentemente rimanere inattivo in un determinato individuo, come per esempio il gene per cotiledoni verdi nel pisello eterozigote. Questo implica che o il gene stesso o il suo prodotto durante lo sviluppo dell'individuo interferiscano con l'allele per il pigmento giallo o con il prodotto di questo gene. Mendel dimostrò che il gene per cotiledoni verdi non veniva affatto modificato durante il suo passaggio in una pianta eterozigote con cotiledoni gialli, dato che i piselli verdi, ottenuti nelle successive generazioni, mostravano di essere puri (omozigoti) per quel carattere. L'interazione nella pianta eterozigote sembrerebbe pertanto avvenire più tra i prodotti del gene che tra gli alleli stessi.
2. Ereditarietà mendeliana
a) Prove dell'ereditarietà mendeliana.
Entro due o tre anni dalla riscoperta del lavoro di Mendel, furono descritti numerosi esempi di ereditarietà mendeliana sia negli animali sia nelle piante, prendendo in esame una grande varietà di caratteri (v. Bateson, 1909). Alcune prove dell'ereditarietà mendeliana sono state ottenute non con esperimenti di incroci controllati tra particolari individui, ma con osservazioni sulla frequenza relativa di individui con differenti caratteri in una popolazione. Questo è possibile in base al teorema fondamentale della genetica delle popolazioni, formulato nel 1908, indipendentemente, da O. A. Hardy e da W. Weinberg. Questi autori dimostrarono che, in assenza di mutazione (un cambiamento improvviso da un allele all'altro) o di selezione, le frequenze dei due omozigoti (AA e aa) e del loro eterozigote (Aa) rimanevano costanti di generazione in generazione (A e a rappresentano una coppia di alleli). Questa conclusione si basa su due ulteriori assunzioni: che la dimensione della popolazione sia abbastanza grande da rendere trascurabili le fortuite fluttuazioni delle frequenze degli alleli e che gli accoppiamenti avvengano in modo del tutto casuale rispetto ai caratteri considerati.
Ed ecco un esempio di applicazione dell'equilibrio di Hardy-Weinberg per le frequenze geniche in una popolazione: il 68,5% degli individui di una popolazione umana è in grado di poter avvertire il sapore amaro di una sostanza chimica (la feniltiocarbammide, FTC) mentre il rimanente 31,5% non mostra questa capacità. Quando entrambi i genitori sono incapaci di percepire il sapore della FTC, anche i loro figli non riescono ad avvertirlo: questo significa che la condizione ‛non gustatore' (aa) è recessiva, per cui gli accoppiamenti tra omozigoti recessivi non danno luogo a segregazioni con ricomparsa dell'altra alternativa. L'ipotesi di Hardy-Weinberg inoltre prevede che il 68,5% della popolazione fornito dell'allele dominante (A) consista del 19,2% di omozigoti (AA) e del 49,3% di eterozigoti (Aa). Sulla base di queste frequenze è quindi possibile determinare la probabilità che nascano figli ‛non gustatori' quando uno o entrambi i genitori siano invece ‛gustatori' ammettendo che la capacità di gustare la FTC sia una caratteristica dominante. Poiché il numero osservato di questi bambini si dimostra in buon accordo con la previsione teorica, questo conferma che le differenze nelle capacità gustative sono espressione di un'ereditarietà di tipo mendeliano.
b) La teoria cromosomica dell'ereditarietà mendeliana
Mendel non localizzò in una qualche struttura della cellula le particelle - i determinanti delle caratteristiche ereditarie - di cui aveva supposto l'esistenza. Come già accennato, Weismann suggeri che i cromosomi - strutture filamentose presenti nei nuclei delle cellule - potessero essere la base dell'ereditarietà. Non appena il lavoro di Mendel fu riscoperto nel 1900, diversi autori indicarono nei cromosomi i possibili vettori dei fattori mendeliani. Prove circonstanziate per questa opinione erano fornite da due osservazioni: 1) gli stessi cromosomi sembrano ereditari, dato che non si formano de novo, ma si originano sempre da preesistenti cromosomi; 2) il numero di cromosomi per cellula è raddoppiato alla fecondazione (numero diploide) ed è in seguito dimezzato (numero aploide) mediante una speciale divisione nucleare chiamata ‛meiosi' durante la formazione delle cellule riproduttive. Questo insieme di fatti si dimostrò sovrapponibile al comportamento dei fattori mendeliani, che sembravano esistere in coppie nei tessuti somatici delle piante e degli animali, ma che segregavano dai loro partners nella formazione delle cellule riproduttive.
La teoria cromosomica dell'ereditarietà mendeliana fu confermata principalmente dalle ricerche di T. H. Morgan e della sua scuola sul moscerino della frutta Drosophila melanogaster. Questo insetto si riproduce rapidamente e ciò consente quindi di studiare numerosi caratteri ereditari. Il primo carattere che risultò associato direttamente ai cromosomi fu il sesso dell'individuo. E. B. Wilson dimostrò che in alcuni insetti il maschio possedeva un cromosoma senza omologo, che chiamò ‛cromosoma X', mentre la femmina mostrava due cromosomi corrispondenti. In altri insetti, come Drosophila, il maschio non solo mostrava un singolo cromosoma X, ma anche un cromosoma più piccolo che non aveva corrispondente nella femmina: Wilson lo denominò cromosoma Y. Così le femmine possedevano due cromosomi X e, dopo la meiosi, le loro cellule uovo contenevano un solo cromosoma X. I maschi potevano avere o un solo cromosoma X o un cromosoma X più un cromosoma Y: gli spermatozoi potevano perciò essere di due tipi, metà con un cromosoma X e quindi destinati a produrre progenie femminile e l'altra metà senza cromosoma X (o in certe specie con un cromosoma Y) destinati ad originare progenie maschile. In seguito si trovò che anche i Mammiferi posseggono un meccanismo simile per la determinazione del sesso, ma che negli Uccelli e nei Lepidotteri la femmina ha nelle sue cellule cromosomi X e Y e i maschi due cromosomi X.
La teoria cromosomica mendeliana dell'ereditarietà si affermò quando Morgan scoprì che un carattere di Drosophila, ereditato secondo gli schemi mendeliani, mostrava di essere associato al sesso. Le Drosofile normalmente hanno occhi rossi, ma presto si trovò una condizione occhi bianchi, che era ereditata come carattere recessivo. Quando moscerini ad occhi rossi e ad occhi bianchi derivanti da linee pure vengono incrociati, la progenie di sesso maschile mostra il colore dell'occhio uguale a quello della madre, il che corrisponde al comportamento del cromosoma X, dato che i maschi ricevono il loro cromosoma X dalla madre. Una conferma che, in Drosophila, il gene per il colore rosso o bianco dell'occhio era localizzato sul cromosoma X fu ottenuta da un collaboratore di Morgan, C. B. Bridges: questi dimostrò che ad una anomalia nella trasmissione ereditaria del colore dell'occhio corrispondeva la formazione di femmine anormali che avevano un cromosoma Y oltre ai loro due cromosomi X. La presenza del cromosoma Y causava alcune volte la non disgiunzione dei due cromosomi X e questo determinava l'eredità anormale del colore dell'occhio.
c) Associazione e scambio (linkage e crossing-over)
Sebbene la teoria cromosomica dell'ereditarietà mendeliana venisse formulata intorno al 1915 da Morgan e collaboratori (v. Morgan e altri, 1915) sulla base dello studio dell'eredità del colore rosso e bianco dell'occhio in Drosophila nei confronti del comportamento dei cromosomi X e Y, essa continuò ad incontrare una certa opposizione per i seguenti motivi: era facile supporre che il numero dei caratteri controllati dai fattori ereditari negli individui di una specie potesse largamente superare il numero dei cromosomi, e questo era senz'altro vero per Drosophila che ha solo 4 cromosomi come assetto aploide; se la teoria cromosomica fosse valida, ci si dovrebbe attendere che molti caratteri mendeliani mostrino di essere ereditari in maniera permanentemente associata, data la localizzazione dei loro geni nello stesso cromosoma; tuttavia mai fu dimostrata un' associazione così permanente tra i caratteri. Lo stesso Mendel aveva studiato la trasmissione ereditaria contemporanea di un certo numero di coppie di caratteri alternativi in Pisum sativum (pisello da orto) ed aveva notato che in ogni caso le coppie di caratteri alternativi erano ereditate indipendentemente l'una dall'altra. Subito dopo la riscoperta del lavoro di Mendel, vennero trovati nel pisello altri esempi di eredità indipendente per coppie alternative di caratteri, cosicché il loro numero venne a superare quello del corredo aploide, che è 7. Questa scoperta sembrava in contraddizione con la teoria cromosomica dell'ereditarietà.
Eccezioni all'eredità indipendente per coppie di caratteri furono invece riscontrate prima da W. Bateson e collaboratori nel 1905 in Lathyrus odoratus (pisello da giardino) e poi da Morgan e collaboratori nel 1911 utilizzando Drosophila. L'aspetto sorprendente di queste osservazioni era che l'associazione si manteneva nell'eredità solo parzialmente, cioè a dire le combinazioni dei caratteri presenti nei genitori tendevano a permanere unite nella progenie e si separavano solo in una piccola percentuale della discendenza. Così, nell'esempio di Bateson, fiori di color porpora e polline di forma allungata erano i caratteri presenti in un genitore, mentre l'altro aveva fiori rossi e polline rotondo. Le frequenze nella progenie mostrarono che nell'88% dei gameti i geni in questione rimanevano associati, mentre nel rimanente 12% si separavano e si ricombinavano in nuova maniera. Nell'esempio di Morgan, occhi di colore bianco e ali ridotte di un genitore, con occhi rossi e ali normali dell'altro, si separavano nel 37% della progenie (frequenza di ricombinazione del 37%); mentre per occhi bianchi e colore del corpo giallo (nei confronti di occhi rossi e colore del corpo grigio) la percentuale corrispondente era solamente dell'1,3%.
I caratteri studiati separatamente da Morgan mostra- vano un'associazione con il sesso (come abbiamo già vi- sto per occhi bianchi nei confronti di occhi rossi). Diversamente da Bateson, egli non esitò quindi a concludere che i geni, per i caratteri che mostravano un'associazione (linkage) parziale, erano tutti localizzati sullo stesso cromosoma. E fu pertanto portato a concludere che un processo di scambio, o crossing-over come lo chiamò, intervenisse alla meiosi tra le coppie di cromosomi omologhi. Ne dedusse che i geni per il colore dell'occhio rosso nei confronti del bianco e per il colore grigio del corpo nei confronti del colore giallo erano piuttosto vicini sul cromosoma X, in maniera da separarsi solamente nell'1,3% dei gameti, mentre i geni per il colore dell'occhio e per le dimensioni delle ali erano molto più distanti così da permettere al crossing-over di intervenire molto più frequentemente.
Molti biologi si mostrarono riluttanti ad accettare la teoria cromosomica dell'ereditarietà, in quanto il processo di scambio di segmenti corrispondenti tra cromosomi omologhi era in contrasto con il concetto della persistenza ed integrità dei cromosomi intesi come supporto dei geni. In realtà, tuttavia, lo studio dell'associazione parziale fornì nuovi validi argomenti a favore della teoria cromosomica. In primo luogo fu risolto il dilemma del numero dei caratteri alternativi ereditati indipendentemente, eccedenti il numero dei cromosomi, perché, come risultato dello scambio, i geni molto distanti lungo un cromosoma verrebbero ereditati indipendentemente, o quasi. Secondariamente, uno dei collaboratori di Morgan, A. H. Sturtevant, dimostrò che le frequenze di ricombinazione erano additive e quindi che i geni potevano essere rappresentati in una configurazione lineare corrispondente all'aspetto filamentoso del cromosoma. In terzo luogo, Morgan e la sua scuola trovarono che in Drosophila i geni responsabili dei caratteri alternativi potevano essere compresi in quattro gruppi: all'interno di questi gruppi si verificava un'associazione parziale nell'ereditarietà, mentre per geni appartenenti a gruppi diversi l'ereditarietà era del tutto indipendente. I quattro gruppi di associazione corrispondevano esattamente ai quattro cromosomi non soltanto nel numero ma anche nelle dimensioni, dato che uno dei gruppi di associazione era molto piccolo, come appunto uno dei cromosomi.
Sebbene gli studi di Morgan portassero necessariamente alla conclusione che coppie di cromosomi omologhi alla meiosi si scambiavano dei segmenti, fu solamente 20 anni dopo la scoperta del crossing-over che si ottenne una prova diretta di questo scambio. H. B. Creighton e B. McClintock con esperimenti su Zea mais (granoturco) e Stern su Drosophila riuscirono ad ottenere degli individui in cui un cromosoma era contrassegnato da due marcatori citologici e da due geni diversi. In altre parole, le linee dei genitori differivano per due caratteri controllati da geni localizzati su quel cromosoma ed erano anche diverse per due riordinamenti strutturali del cromosoma visibili al microscopio. Si poté controllare che, allorquando la progenie mostrava crossing-over tra i caratteri alternativi dei genitori, contemporaneamente si manifestavano nuove combinazioni delle caratteristiche strutturali del cromosoma.
d) Crossing-over e chiasmi
Allo stadio diplotene della meiosi (v. genetica: Citogenetica) i due cromosomi omologhi sono tenuti insieme da una serie di punti di contatto intercalati ad anse. A questo stadio, ciascun cromosoma consta di due filamenti fratelli chiamati ‛cromatidi', che decorrono parallelamente. Ai punti di contatto, o chiasmi, tuttavia, i cromatidi sembrano scambiarsi i partners (v. fig. 1). In altre parole, ad ogni chiasma due filamenti si toccano e due no. Attualmente si ritiene in generale che i chiasmi osservati al diplotene siano causati dal crossing-over, cioè dallo scambio di segmenti tra cromosomi omologhi, e si pensa che questo scambio avvenga durante il precedente stadio della meiosi, il pachitene. La prova che i chiasmi osservati citologicamente corrispondono ai ricombinanti osservati geneticamente si basa su numerosi dati sperimentali di diverso tipo.
In primo luogo è stato possibile dimostrare che lo scambio di segmenti tra cromosomi omologhi avviene non tra gli interi cromosomi, ma tra i singoli cromatidi di ciascun genitore, come ci si attendeva dall'aspetto citologico del chiasma. La dimostrazione che il crossing-over ha luogo tra cromatidi fu fornita da Bridges e E. G. Anderson con una serie di esperimenti pubblicati tra il 1916 e il 1925. Questi due autori studiarono l'ereditarietà di caratteri legati al sesso, usando diversi tipi di femmine anormali di Drosophila, in cui entrambi i cromosomi X rimanevano nella stessa cellula uovo. Una dimostrazione ancora più diretta che il crossing-over avviene tra cromatidi fu fornita da C. C. Lindegren nel 1933 utilizzando il fungo Neurospora crassa. Il vantaggio di usare questo organismo per questo scopo è dato dal fatto che i prodotti della meiosi sono spore, che si ritrovano insieme in una cellula gigante chiamata asco. Lindegren usò ceppi che differivano per caratteri associati (diversi cioè per geni associati sullo stesso cromosoma) e dimostrò, facendo germinare le spore da singoli aschi, che quando era intervenuto un crossing-over tra i geni dei genitori solamente due dei quattro prodotti della meiosi erano ricombinanti.
L'idea che un chiasma corrispondesse ad un crossing-over trovò convalida nel confronto della loro rispettiva frequenza e distribuzione in particolari cromosomi. Nel 1916, un collaboratore di Morgan, H. J. Muller, lavorando su Drosophila, scoprì che i ricombinanti si distanziavano in un certo modo lungo il cromosoma, e cioè che tra di loro vi era una interferenza positiva. Più tardi, nel 1931, J. B. S. Haldane sulla base di dati ottenuti da T. Maeda su Vicia faba (fava commestibile) dimostrò che i chiasmi erano a loro volta corrispondentemente distanziati lungo il cromosoma. C. D. Darlington, nel 1934, trovò una soddisfacente concordanza tra le frequenze dei chiasmi osserva- ti alla meiosi nelle cellule madri del polline di Zea e le frequenze di ricombinanti basate sulla lunghezza delle mappe di associazione. Queste mappe sono costruite sulla base delle frequenze con cui determinati caratteri associati compaiono nella progenie in nuove combinazioni, e sono pertanto una misura della frequenza con cui gli scambi si verificano tra cromosomi omologhi nei vari segmenti delimitati dai geni studiati. Come atteso, la frequenza dei chiasmi era alquanto più grande della frequenza di ricombinazione, in quanto i chiasmi che si verificano vicino alle estremità dei cromosomi non possono essere computati, a meno che non si conoscano delle caratteristiche controllate da geni molto vicini alle estremità.
Prove dirette che i chiasmi corrispondono ai ricombinanti sono state ottenute studiando i cromosomi meiotici in diverse situazioni speciali. Queste includono: a) coppie di cromosomi strutturalmente ineguali; b) tre o più cromosomi dello stesso tipo; c) la presenza in uno dei due omologhi di un segmento invertito. L'ultima ha fornito la prova più convincente, perché quando i due genitori differiscono per una inversione su di un braccio cromosomico, una ricombinazione all'interno dell'inversione dà luogo ad un cromosoma con due centromeri e a un frammento acentrico. Ne risulta un ponte e un frammento all'anafase I che costituiscono la prova di un crossing-over avvenuto entro il tratto invertito. J. Brown e D. Zohary nel 1955 paragonarono le frequenze dei chiasmi contenuti in un inversione di Lilium formosanum con le frequenze di ricombinanti rilevabili come ponti e frammenti in anafase, e trovarono una soddisfacente corrispondenza.
Vi sono pertanto argomenti validi per ritenere che ciascun chiasma visibile allo stadio di diplotene durante la meiosi sia la conseguenza di uno scambio di segmenti tra due dei quattro cromatidi costituenti le tetradi. Il meccanismo effettivo per questi scambi verrà discusso più avanti.
e) L'ereditarietà dei caratteri quantitativi
Durante il primo decennio del XX secolo si vennero a formare opinioni contrastanti sull'ereditarietà dei caratteri quantitativi, quali altezza e peso. Oggi è evidente che gran parte di queste controversie si originò dalla confusione teorica sulla natura dell'ereditarietà mendeliana. Il metodo per studiare l'ereditarietà introdotto da Mendel era quello di trovare linee pure che differissero per caratteri qualitativi ben evidenti, e quindi di incrociare tali linee seguendo i caratteri prescelti nelle successive generazioni. Questo metodo tuttavia non è applicabile ai caratteri quantitativi, tanto che all'inizio fu impossibile stabilire se le variazioni quantitative fossero ereditate nella stessa maniera o diversamente da quelle qualitative. La difficoltà si origina, come dimostrato dagli studi di Johannsen (v., 1909) sull'eredità del peso dei semi di Phaseolus vulgaris (fagiolo comune), dal fatto che anche all'interno di una linea pura vi è una discreta variabilità nel peso dei semi: con il risultato che, quando vengono incrociati fagioli leggeri con fagioli pesanti, la progenie si dispone lungo una scala continua di variazioni di peso. Johannsen risolse la prevalente confusione concettuale stabilendo per la prima volta una chiara distinzione tra carattere ereditario (in questo caso il peso del fagiolo) e suoi determinanti, che ritenne fossero un certo numero di fattori mendeliani (e per i quali coniò il termine gene). Introdusse anche il termine ‛genotipo' per indicare l'insieme dei fattori ereditari di un organismo, e ‛fenotipo' per indicare l'aspetto di un organismo nei riguardi dei caratteri considerati. La sua tesi era che il fenotipo fosse una conseguenza dell'interazione tra il genotipo e l'ambiente durante lo sviluppo. Così un fagiolo di peso intermedio poteva essere il risultato di fattori sfavorevoli durante il suo sviluppo (quale per es. la sua posizione nel baccello) che si erano opposti agli effetti di un genotipo favorevole, oppure poteva conseguire a condizioni di crescita ottimali per una razza a basso peso medio. In altre parole, per usare la sua terminologia, il fenotipo intermedio poteva essere la conseguenza di un genotipo mediocre e di favorevoli condizioni ambientali, o viceversa.
Gli esperimenti di Johannsen dimostrarono che in una popolazione mista la selezione in ogni generazione per fagioli pesanti poteva far sì che un numero sempre maggiore di essi appartenesse al genotipo in grado di dare origine ai fagioli più pesanti, fino a che tutti i fagioli risultassero appartenenti a tale ceppo: a questo punto la selezione non avrebbe avuto ulteriore effetto. I suoi studi dimostrarono chiaramente che la selezione non poteva avere efficacia nell'aumentare il peso medio dei fagioli quando la variabilità fosse dovuta solamente a fattori ambientali. Questa sua scoperta ha delle implicazioni ovvie per gli allevatori di piante e di animali: la variabilità ereditaria è essenziale per ottenere dei miglioramenti nelle piante e negli animali di interesse economico.
I risultati di Johannsen erano in perfetto accordo con l'ipotesi che il peso dei fagioli fosse controllato da numerosi geni per effetto additivo, ammesso però che le variazioni dovute ai geni potessero essere modificate dagli effetti ambientali. Tra le prove più evidenti a favore dell'ipotesi dei geni multipli quali determinanti delle variazioni continue dei caratteri quantitativi vi furono gli studi di E. M. East nel 1910 e di R. A. Emerson e East nel 1913 sull'eredità dei caratteri quantitativi in Zea. Quando una pianta di granturco di una linea pura a spighe corte veniva incrociata con una pianta di una linea pura a spighe lunghe, la progenie (prima generazione filiale o F1) era relativamente omogenea e con spighe di lunghezza intermedia. Questo risultato è in accordo con l'ipotesi dei geni multipli, in quanto tutte le piante dell'F1 avranno lo stesso genotipo, essendo eterozigoti per tutti i geni per cui differivano i genitori. Con l'incrocio tra di loro, o con l'autofecondazione delle piante F1, si ottenne una seconda generazione o F2. Queste piante possedevano spighe con una lunghezza media che era approssimativamente quella della F1, ma con una variabilità tra gli individui molto maggiore. Questi dati rafforzano notevolmente l'ipotesi dei geni multipli, perché molte delle piante F2, contrariamente a quelle F1, differivano alquanto nel loro genotipo. E così si spiegherebbe anche il perché della grande variabilità nella lunghezza delle spighe tra le piante F2. Emerson e East ottennero risultati simili con molti altri caratteri quantitativi nel granturco, quali il peso delle cariossidi, l'altezza delle piante e il numero dei nodi lungo lo stelo.
L'idea che i caratteri quantitativi non seguissero l'ereditarietà mendeliana si era sviluppata dopo il lavoro di F. Galton e di K. Pearson sulla correlazione tra parenti per caratteri quali la statura. I dati più accurati furono raccolti nel 1903 da Pearson e N. Lee, e dimostrarono come il coefficiente di correlazione per la statura tra padre e figlio fosse 0,51 e tra fratelli 0,54 (un coefficiente = 0 significa assenza di correlazione, mentre = 1 significa correlazione completa). Pearson dimostrò tuttavia che i coefficienti di correlazione previsti, in base all'ipotesi che la statura fosse determinata da numerosi fattori mendeliani, avrebbero dovuto essere 0,33 e 0,42 rispettivamente e concluse pertanto che non si trattava di un caso di eredità di fattori multipli. Pearson aveva però supposto che tutti i fattori mendeliani avessero effetti uguali ed additivi, che i due alleli di una coppia fossero ugualmente frequenti e che, nel caso di eterozigosi, uno dei due alleli sarebbe stato completamente dominante. Bisognò arrivare al 1918 per comprendere il divario tra i dati di Pearson e le previsioni teoriche, quando il problema venne riesaminato da R. A. Fisher. Questi dimostrò che i bassi coefficienti di correlazione previsti da Pearson sulla base dell'ipotesi multifattoriale erano una conseguenza delle sue assunzioni sulla dominanza e sull'eguaglianza delle frequenze dei vari alleli, e dell'eguaglianza degli effetti per i vari fattori. Fisher non partì da tali assunzioni e poté dimostrare che il coefficiente di correlazione più alto tra fratelli che non tra padre e figlio poteva essere in accordo con la teoria dei geni multipli nel caso vi fosse una certa dominanza tra gli alleli. Concluse che i dati erano in pieno accordo con questa teoria, essendo quasi tutte le differenze di statura tra fratelli attribuibili alla segregazione di fattori mendeliani per cui i genitori erano eterozigoti. Fisher introdusse inoltre concetto di additività della varianza, quest'ultima definita come la misura della quantità per cui una serie di conteggi o misure differiscono dalla media. Ed ancora dimostrò che quando vi siano molteplici cause di variabilità entro una popolazione, i loro contributi alla varianza sono additivi. Le sue conclusioni sul coefficiente di correlazione per la statura tra fratelli = 0,54 (calcolato da Pearson e Lee) furono che il 54% della varianza dei fratelli era dovuto al fatto che essi avevano ricevuto geni identici determinanti la loro altezza. Fisher stimò inoltre che non più del 5% della varianza tra fratelli fosse dovuto a fattori ambientali e che il rimanente 41% dovesse essere attribuito all'aver ricevuto dai loro genitori alleli diversi.
La scomposizione della varianza tra le varie cause della variabilità è servita di base a molti studi successivi sulla ereditarietà dei caratteri quantitativi ed ha permesso a K. Mather di riconoscere l'associazione parziale tra alcuni dei fattori mendeliani multipli. Tale associazione è del tutto comparabile con quella già descritta per i geni determinanti i caratteri qualitativi. L'idea che i caratteri provvisti di una variabilità continua sono ereditati essenzialmente nella stessa maniera dei caratteri studiati da Mendel è adesso provata da numerosissimi dati. La differenza essenziale è che i caratteri quantitativi sono controllati da numerosi geni invece che da un sol paio e che gli effetti di questi geni sono additivi.
3. Ereditarietà non-mendeliana
Sebbene esistano numerose prove che la maggior parte delle variabilità continue siano ereditate secondo schemi mendeliani, non è detto che tutti i caratteri degli organismi viventi siano ereditati nella stessa maniera. Nel 1909 C. Correns e E. Baur indipendentemente scoprirono l'ereditarietà non-mendeliana.
Correns (v., 1909) studiò l'eredità del carattere foglia pallida o variegata in diverse piante da fiore. La maggior parte di queste differenze dalle normali foglie verdi mostrò un'eredità di tipo mendeliano, ma in Mirabilis jalapa le foglie variegate con bianco giallastro (albomaculate) si comportavano in maniera diversa. Sulle piante albomaculate alcune volte vi erano dei germogli interamente verdi o interamente bianchi. Correns trovò che i fiori sui germogli verdi davano solamente progenie verde, i fiori sui germogli bianchi solamente progenie bianca (che moriva abbastanza precocemente) e che i fiori sui germogli variegati davano progenie verde, variegata e bianca senza un rapporto fisso: tanto più grandi le aree verdi, tanto più numerosa la progenie verde. L'origine del polline, che provenisse cioè da fiori di germogli verdi, o bianchi, o variegati, non aveva nessuna influenza sul tipo di progenie. Questi risultati differivano in maniera molto netta dagli schemi dell'ereditarietà mendeliana, specialmente per la presenza di un'eredità di tipo materno e per la mancanza di precisi rapporti di segregazione.
Baur (v., 1909) studiò l'eredità del carattere foglie a margine bianco in Pelargonium zonale e trovò che quando tali piante erano autofecondate o incrociate tra loro, la progenie si presentava interamente bianca e perciò moriva. Questo risultato era comprensibile in quanto in Pelargonium a margini bianchi lo strato sottoepidermico, da cui si originano le cellule germinali, è privo di clorofilla. Quando le piante a margini bianchi erano incrociate con piante normali verdi, si potevano osservare nella progenie piante verdi, a margine bianco e bianche, ma le proporzioni dei tre tipi di piante variavano sensibilmente da un incrocio all'altro. Diversamente dai risultati ottenuti da Correns con Mirabilis albomaculata, Baur trovò che gli incroci reciproci davano risultati sostanzialmente simili. In altre parole, negli incroci tra piante verdi e piante a margini bianchi, l'incrocio poteva essere effettuato scambiando il sesso dei genitori, ma la progenie consisteva sempre di piante verdi, a margine bianco e bianche in proporzioni irregolari.
La caratteristica più rilevante dell'ereditarietà non-mendeliana, messa in evidenza da questi esempi, è l'assenza di uniformità per la progenie F1 e la segregazione irregolare dei caratteri considerati. Baur spiegò i suoi risultati postulando che i plastidi fossero delle strutture autonome autoduplicantesi, e che nelle aree bianche di Pelargonium a margini bianchi essi si trovassero in forma incolore per mancanza di clorofilla. L'ereditarietà non-mendeliana sarebbe quindi la conseguenza della trasmissione di plastidi difettivi attraverso il citoplasma. Questa ipotesi fu avvalorata da numerose considerazioni ed osservazioni: 1) il fatto che si ottengano tre tipi di piante e più precisamente verdi, variegate e bianche, può essere spiegato e messo in relazione con individui aventi uno dei tipi di plastidi o una commistione dei due tipi; 2) il rapporto notevolmente variabile tra i tre tipi di piante si spiegherebbe se il meccanismo con cui le cellule figlie ricevono i loro plastidi fosse alquanto impreciso; 3) le zone bianche mostrano di possedere plastidi piccoli e senza colore; 4) l'eredità materna per il carattere albomaculata in Mirabilis si spiegherebbe se i plastidi raggiungessero l'uovo fecondato solo attraverso il gamete femminile, mentre la similarità degli incroci reciproci in Pelargonium suggerirebbe che in quel genere i plastidi arrivano nella cellula uovo sia con i tubi pollinici, sia attraverso il tessuto materno.
L'ereditarietà mendeliana per il carattere foglie variegate, riscontrata in molte piante anche da Correns, si spiegherebbe se alcune delle caratteristiche dei plastidi fossero controllate da geni localizzati sui cromosomi entro i nuclei cellulari. Correns non condivise l'ipotesi di Baur dei plastidi anomali per spiegare l'ereditarietà non mendeliana, sulla base delle seguenti considerazioni: 1) nelle piante variegate, al confine tra le aree verdi e quelle bianche, vi era una gradazione del colore dei plastidi, piuttosto che una mescolanza dei due tipi di plastidi. Questo valeva nel caso che la variegazione mostrasse un'eredità mendeliana o non-mendeliana, e suggeriva che in entrambi i casi una qualche sostanza diffondesse dalle aree bianche e interferisse con il normale sviluppo dei plastidi; 2) non vi erano abbastanza divisioni cellulari durante lo sviluppo perché i due ipotetici tipi di plastidi potessero segregare in diverse cellule, tranne in rari casi, e con tutto ciò le aree verdi e le aree bianche si formavano piuttosto frequentemente. Queste obiezioni evocarono la possibilità che altre particelle nel citoplasma, oltre ai plastidi, potessero essere responsabili dell'eredità non-mendeliana della variegazione. Recenti progressi nella comprensione dell'ereditarietà non-mendeliana verranno discussi più avanti.
4. Problemi dell'ereditarietà mendeliana
a) Alleli multipli.
L. Cuénot scoprì nel 1904 che il numero di alternative per caratteristiche che mostravano un'eredità di tipo mendeliano non era limitato solamente a due. Nel lavoro di Mendel erano state studiate, nei piselli, coppie di caratteri alternativi, quali liscio e grinzoso. Cuénot si occupò dell'eredità nel topo (Mus masculus) di tre colori del mantello - giallo, grigio e nero - e trovò che, per ogni coppia, i colori si comportavano come se fossero allelici, con il giallo dominante sul grigio e il nero, e con il grigio dominante sul nero. Emerson, nel 1911, descrisse uno dei primi esempi di alleli multipli trovati nelle piante - una serie di colori del tutolo (l'asse su cui si formano le cariossidi della spiga) e del pericarpo (il tessuto ovarico che circonda le cariossidi) nel granturco (Zea mais). Fu dimostrato che in questi tessuti le varie gradazioni e combinazioni di antocianine rosse erano tutte ereditate come caratteristiche alternative, con la presenza del pigmento e della gradazione più marcata dominanti nei confronti della assenza di pigmento o della gradazione più chiara.
Il lavoro di Morgan e dei suoi collaboratori sull'eredità dei colori dell'occhio in Drosophila melanogaster illustrò gli aspetti caratteristici degli alleli multipli. Il carattere occhi bianchi, ereditato come recessivo nei confronti del carattere normale occhio rosso, è già stato discusso. Fu trovato che un colore rosa giallognolo, denominato eosin, era un allele di white e si comportava da dominante nei confronti di white e da recessivo nei confronti di occhio rosso. D'altra parte il colore rosa (pink) dell'occhio si dimostrò non allelico con eosin e con white e veniva ereditato come carattere indipendente da quelli. Da un incrocio di moscerini di linee pure pink e white, la progenie F1 mostrava occhi rossi, indicando che ciascun genitore era portatore dell'allele normale dominante nei confronti del mutante recessivo mostrato dall'altro genitore. Nella F2 si ottennero pertanto moscerini doppio-mutanti e normali (con occhi rossi), oltre a moscerini con occhi pink e white. Al contrario i mutanti allelici eosin e white non davano, quando incrociati, alcun moscerino a occhi normali o doppio-mutante. Era evidente che i moscerini occhio eosin e occhio white erano portatori di diversi alleli dello stesso gene, mentre i moscerini a occhio pink possedevano un allele di un diverso gene, anch'esso in grado di alterare il colore dell'occhio. Questa conclusione fu confermata da studi sull'associazione e ricombinazione: il carattere eosin mostrava le stesse frequenze di ricombinazione del carattere white con diversi altri geni legati al sesso, mentre l'occhio pink mostrava di essere associato a caratteri determinati da geni di un altro gruppo di associazione. Questo significava che il gene per l'occhio pink era localizzato in un cromosoma diverso da quello del gene per l'occhio white e eosin.
Un esempio molto noto di alleli multipli è quello dei gruppi sanguigni umani O, A, B e AB, riconosciuti per la prima volta da F. Bernstein nel 1925 come appartenenti ad una serie allelica. I quattro gruppi sanguigni corrispondono alla presenza o all'assenza di due antigeni, A e B, negli eritrociti. Gli individui di gruppo A possono essere omozigoti per l'allele A o eterozigoti AO; similmente avviene per il gruppo B, mentre il gruppo O è omozigote OO ed è privo di ambedue gli antigeni. Weinberg nel 1909 aveva esteso il teorema di Hardy-Weinberg al caso di tre alleli in una popolazione, e Bernstein mostrò che le frequenze relative dei quattro gruppi sanguigni erano in accordo con le previsioni basate sulla ipotesi di Weinberg per gli alleli multipli.
b) Mutazioni
Uno dei fondamentali problemi posti dall'ereditarietà mendeliana fu quello dell'origine delle differenze alla base dei caratteri allelici. Il termine usato per descrivere la base di tali differenze è ‛mutazione'. Questo termine fu introdotto nella letteratura biologica da H. De Vries nel 1901, ma per descrivere un diverso processo e più precisamente l'origine delle nuove specie. Egli pensava che le specie potessero originarsi improvvisamente e senza gradualità dalle specie preesistenti ed ottenne quella che considerava la conferma sperimentale della sua ipotesi in Oenothera lamarckiana. Solamente alcuni anni dopo, tuttavia, si riuscì a comprendere appieno il comportamento di questa pianta. Oggi si sa che Oenothera lamarckiana contiene una certa quantità di variabilità genetica inespressa: cioè un certo numero di geni recessivi normalmente non manifestano il loro effetto perché sono mantenuti permanentemente allo stato eterozigote da un meccanismo alquanto elaborato. Le mutazioni di De Vries erano la manifestazione di questi geni piuttosto che la loro origine, ma il termine è usato adesso in quest'ultimo senso, cioè ad indicare l'improvviso cambiamento di un gene che dà origine ad un nuovo allele. Gli aspetti caratteristici della mutazione, in questo senso, sono che il cambiamento è improvviso, agisce su un solo carattere alla volta ed è permanente, cioè la condizione mutante è ereditaria.
Queste caratteristiche delle mutazioni furono descritte da Morgan e collaboratori nel loro fondamentale lavoro su Drosophila melanogaster. Il mutante ad occhi bianchi, di cui si è già parlato, si originò dal normale ad occhi rossi nell'anno 1910 ed il terzo allele, colore dell'occhio eosin, derivò dall'occhio bianco nell'anno successivo. In seguito, si poté stabilire che l'occhio eosin poteva originare direttamente da quello rosso e che occasionalmente poteva retromutare a rosso o a bianco. Evidentemente gli alleli multipli rappresentavano forme multiple di un gene e il processo della mutazione li poteva trasformare uno nell'altro in ogni direzione. Tuttavia la frequenza con cui si verificavano le mutazioni era estremamente bassa, con il risultato che il carattere normalmente veniva trasmesso inalterato. La bassa frequenza e la natura imprevedibile delle mutazioni rese impossibile lo studio sperimentale di questo fenomeno sino a quando H. J. Muller, uno dei collaboratori di Morgan, non sviluppò delle tecniche molto ingegnose per poterne fare un'analisi sperimentale.
Muller ritenne, per una serie di considerazioni teoriche, che le mutazioni che avrebbero determinato la morte dell'individuo dovessero essere più frequenti di quelle che ne avrebbero alterato solo il fenotipo senza essere mortali, e così mise a punto un metodo per individuare le mutazioni letali. Il suo lavoro con Drosophila aveva dimostrato che la maggior parte delle mutazioni sono recessive, e pertanto anche le mutazioni letali, nella stragrande maggioranza, non si sarebbero manifestate al momento della loro insorgenza, ma solamente quando si fossero trovate in omozigosi in una generazione successiva. Il problema di individuare le mutazioni letali era straordinariamente difficile, dato che ci si poteva aspettare che gli omozigoti morissero poco dopo la fecondazione dell'uovo. Il metodo che Muller escogitò e mise a punto viene generalmente chiamato tecnica del ClB, dove C sta per repressore della ricombinazione, i per mutazione letale e B per occhio a sbarra. Il principio su cui si basa la tecnica era che, siccome i maschi di Drosophila hanno solamente un cromosoma X mentre le femmine ne hanno due, una singola dose di una mutazione recessiva letale in quel cromosoma avrebbe ucciso i maschi ma non le femmine. I maschi ricevono il loro cromosoma X dalle madri, ma vi è una probabilità del 50% di ricevere l'uno o l'altro dei due cromosomi X materni. Muller fece sì che un determinato gene letale recessivo (l'l nel ClB) fosse presente in uno dei due cromosomi X, in maniera che se un altro gene letale recessivo fosse stato indotto nell'altro cromosoma X queste femmine non avrebbero prodotto progenie maschile. Era anche necessario avere un repressore di crossing-over (C) per impedire che si formassero, in seguito a ricombinazione, cromosomi X privi di entrambi i letali, mentre il gene dominante per occhi a sbarra (B) legato al sesso venne usato per riconoscere le femmine portatrici di C e l.
Nel 1927 Muller, utilizzando questa tecnica del ClB, scoprì che i raggi X aumentavano grandemente la frequenza di mutazioni, se confrontata con quelle che si potevano verificare spontaneamente. Irradiò delle drosofile di sesso maschile, le incrociò con femmine ClB eterozigoti e di nuovo accoppiò le femmine F1 con l'occhio a sbarra, e quindi ClB, con maschi normali. Se questo incrocio non dava progenie maschile, evidentemente una mutazione letale recessiva era stata indotta sul cromosoma X trasmesso dal maschio originario. Con la dose massima di raggi X usata (un po' al di sopra dei 4.000 r) Muller trovò che di 772 femmine esaminate 89, cioè l'11,5% non avevano progenie maschile: questo costituiva un aumento di 140 volte rispetto alla frequenza di mutazioni spontanee. La frequenza di mutazioni sembrava essere direttamente proporzionale alla dose totale dei raggi X, senza che vi fosse una dose soglia e senza un effetto dell'intensità della dose. Furono così indotte mutazioni capaci di alterare molti diversi caratteri oltre a quelle con effetto letale, sebbene queste ultime, come Muller aveva previsto, fossero circa 10 volte più frequenti di quelle non letali. Tuttavia non si raccolsero indicazioni sulla possibilità che i raggi X fossero in grado di causare la mutazione preferenziale di geni specifici, e, sebbene diversi geni mutassero con frequenze significativamente diverse, questo era comparabile con quanto avveniva per le mutazioni spontanee.
Contemporaneamente alla scoperta di Muller che i raggi X erano in grado di indurre mutazioni in Drosophila, L. J. Stadler scoprì che i raggi X e i raggi y causavano mutazioni in Hordeum vulgare (orzo). Cariossidi in germinazione furono irradiate e lasciate crescere fino a maturità: in seguito ad autofecondazione alcune spighe produssero tra la progenie alcuni individui anormali, ad esempio mutanti per il colore delle foglie.
Poco tempo dopo queste scoperte iniziali, si accertò che tutte le radiazioni ionizzanti e la luce ultravioletta potevano causare mutazioni dei più diversi tipi. Si prospettò così la possibilità che i raggi cosmici e la radioattività ambientale svolgessero un ruolo importante nell'indurre mutazioni, ma alcuni calcoli hanno dimostrato che la frequenza di mutazioni spontanee in Drosophila era di 500-1.000 volte più alta di quella che poteva essere attribuita alla radioattività naturale. Inoltre la frequenza di mutazioni era più bassa in organismi a lungo ciclo vitale, quale l'uomo, che non in Drosophila a corto ciclo vitale: questo perché, evidentemente, la selezione naturale ha favorito una maggiore stabilità dei geni negli organismi a lungo ciclo vitale. Questa conclusione implicava che la frequenza di mutazioni poteva essere controllata chimicamente.
La prima prova diretta che sostanze chimiche fossero in grado di indurre mutazioni fu ottenuta nel 1941, anche se fu resa pubblica alcuni anni dopo. C. Auerbach e J. M. Robson (v., 1947), utilizzando la tecnica di Muller del ClB, scoprirono che l'azotoiprite e l'aldeide formica, rispettivamente, potevano indurre mutazioni. Successive ricerche rivelarono che queste ed altre sostanze, quali l'uretano, il cloruro di manganese e l'acqua ossigenata (perossido di idrogeno) davano origine ad un ampio spettro di mutazioni e che, come nel caso delle radiazioni ionizzanti, non vi era nessuna prova per una qualche specificità d'azione dei mutageni nei confronti di geni particolari.
c) La natura del gene
L'esistenza degli alleli multipli e la possibilità di mutazioni furono prese in considerazione nel definire teoricamente il gene. Il gene poteva essere definito sulla base dei suoi specifici effetti su di un carattere o su un gruppo di caratteri, dei suoi cambiamenti in una forma allelica per effetto della mutazione e della ricombinazione con geni confinanti per effetto del crossing-over. La maggior parte dei genetisti ritenne che questi tre concetti dell'unità ereditaria fossero equivalenti tra loro, e cioè che esistesse una struttura molecolare discreta alla quale potesse essere applicata la definizione di gene. Questa molecola, o aggregato molecolare, poteva influenzare lo sviluppo di un organismo in una maniera specifica, poteva andare incontro a cambiamenti chimici improvvisi in maniera da modificare quello sviluppo, e poteva infine essere separata dai suoi vicini in un cromosoma ad opera di un processo di scambio. Una maniera conveniente per rappresentare questa ipotesi del gene era quella di assimilare il cromosoma ad una collana di perline dove ciascuna perlina rappresentasse un diverso gene. J. Belling, nel 1928, suggerì che la somiglianza di un cromosoma durante la profase precoce, prima della meiosi, con un filo di perline non fosse una coincidenza, e che ciascuna perlina o cromomero corrispondesse ad un gene (v. fig. 2).
Fu sottolineato, tuttavia, da R. B. Goldschmidt nel 1938 e da D. Raffel e Muller nel 1940, che non vi era alcuna evidenza che i segmenti cromosomici definiti in base all'azione del gene, alla mutazione ed alla ricombinazione corrispondessero tra di loro, ed anzi numerose indicazioni suggerivano il contrario. L'idea dei geni come particelle discrete e indipendenti fu messa in dubbio per la prima volta in seguito a un'importante scoperta fatta da Sturtevant. Come già accennato, in Drosophila la forma ristretta dell'occhio chiamato occhio a sbarra (bar) risultò essere dovuta alla trasmissione di un gene dominante legato al sesso. Questo carattere si comportava in maniera insolita, tuttavia, poiché mostrava una frequente mutazione ad occhio normale rotondo e anche ad una forma più estrema chiamata ultrabar (adesso denominata più spesso doppiobar). Sturtevant e Morgan trovarono che questi cambiamenti erano associati con il crossing-over in una regione del cromosoma corrispondente al gene bar e che quindi non erano in effetti delle vere mutazioni. Nel 1936 Muller (e collaboratori) e Bridges, indipendentemente, scoprirono che la forma a sbarra dell'occhio bar era associata a un breve segmento duplicato di cromosoma e che ultrabar era dovuto allo stesso segmento triplicato. Il cambiamento da bar a normale e a ultrabar, in seguito a crossing-over, poteva adesso essere spiegato come il risultato di un appaiamento sfalsato della duplicazione bar. Un crossing-over che fosse avvenuto fra i due cromosomi X proprio in questa regione avrebbe generato un cromosoma X con solamente una singola copia del segmento e quindi progenie con occhi normali, e un altro cromosoma X con tre copie e quindi progenie ultrabar.
Sturtevant nel 1925 trovò che gli eterozigoti per ultrabar e normale avevano occhi più ridotti che non i bar omozigoti. Alla luce della successiva scoperta che il carattere bar era dovuto a una duplicazione in tandem, la scoperta di Sturtevant significava che tre copie di un segmento cromosomico in un cromosoma e una in quello omologo davano origine a un fenotipo diverso da quello dovuto a due copie in ciascun cromosoma. Non vi era dubbio che questi due genotipi, l'eterozigote per la triplicazione e la singola copia, e l'omozigote per la duplicazione, contenessero lo stesso materiale genetico, poiché non solo l'ultrabar e il normale si originavano dal bar omozigote in seguito a crossing-over, ma poteva verificarsi anche la situazione reciproca: bar poteva formarsi come risultato di un crossing-over nell'eterozigote di ultrabar e normale. Era così evidente che il comportamento di un gene mostrava un effetto di posizione, cioè era influenzato dalle regioni adiacenti del cromosoma. Evidentemente i geni non si comportavano sempre alla stregua di particelle discrete come si era supposto.
La scoperta che l'occhio bar dipende da un segmento ripetuto di cromosoma era stata possibile perché le larve di Drosophila possiedono dei cromosomi giganti nei nuclei delle cellule delle ghiandole salivari. Questi cromosomi giganti dei Ditteri, descritti sin dal XIX secolo ma poi dimenticati, furono nuovamente scoperti da T. S. Painter nel 1934. I cromomeri dei cromosomi meiotici sono rappresentati nei cromosomi delle ghiandole salivari come dei dischi trasversali o bande di dimensioni diverse. Nel cromosoma X di Drosophila possono essere identificate oltre 1.000 bande in una sequenza caratteristica. Nei moscerini con occhio bar, sei di queste bande furono trovate ripetute nella stessa sequenza immediatamente dopo la sequenza originaria. Nei cromosomi delle ghiandole salivari, i cromosomi omologhi sono strettamente appaiati per tutta la loro lunghezza, in maniera che ciascuna banda trasversale è associata con la sua omologa. Nei moscerini eterozigoti per il carattere dell'occhio bar e dell'occhio normale, il segmento ripetuto apparirà in maniera evidente, poiché non si potrà appaiare con la regione omologa dando così origine ad un'ansa sporgente.
Una serie di lavori su Drosophila dimostrarono inoltre una mancanza di corrispondenza tra le unità ereditarie definite in base alla funzione, mutazione e ricombinazione. Uno degli studi più chiari fu una ricerca fatta da E. B. Lewis nel 1941 su due caratteri mutanti dell'occhio chiamati star. Questi mutanti determinavano un occhio più piccolo, con una superficie più ruvida del normale, e sembravano essere allelici. Gli eterozigoti per i due alleli, tuttavia, davano progenie ad occhio normale con una frequenza di uno ogni qualche migliaio di individui mutanti. Lewis trovò che questa progenie normale era sempre associata con il verificarsi del crossing-over nel cromosoma alla posizione del gene star. Questo fu dimostrato dal comportamento di altri geni strettamente associati ad entrambi i lati della regione considerata. Risultò così che i due alleli star non occupavano posizioni identiche nel cromosoma e che il crossing-over poteva intervenire occasionalmente tra di loro, poiché tutta la progenie ad occhio normale aveva un particolare genotipo ricombinante per i geni esterni. Così, se a e b indicano i mutanti esterni, S ed s i mutanti star (uno dominante e l'altro recessivo) e un segno + sta per i geni normali, l'eterozigote parentale aveva il genotipo
e la rara progenie ad occhio normale aveva il genotipo ++b. Se ne poteva dedurre che il genotipo iniziale fosse in realtà
da cui mediante un crossing-over tra S e s poteva originarsi progenie ad occhio normale +++b.
Come già accennato, una delle caratteristiche di una coppia di mutanti allelici è che l'eterozigote, cioè l'individuo con un mutante su di un cromosoma e l'altro sull'omologo, mostra un fenotipo mutante. McClintock, nel 1944, trovò un'eccezione a questa regola. Due mutanti allelici per il gene yellow-green-2 in Zea (granturco) davano plantule rispettivamente giallo-verdi e giallo pallide quando omozigoti, mentre l'eterozigote per i due mutanti mostrava foglie di colore verde, normali. Sembrava così che mutanti allelici potessero in certi casi essere complementari fra loro, ciascuno supplendo alla deficienza dell'altro. Ciò implicava che i due mutanti differivano dal normale nei punti che non corrispondevano tra loro.
La scoperta di Sturtevant dell'effetto di posizione, di Lewis della ricombinazione intragenica e di McClintock della complementarità intragenica suggerivano nel loro insieme che l'analogia tra i geni nei cromosomi e le perline della collana era senz'altro un'eccessiva semplificazione (v. gene).
d) Modificazioni strutturali dei cromosomi
Muller scoprì che i raggi X non solo causavano una più alta frequenza di mutazioni, ma anche cambiamenti strutturali nei cromosomi. Questo fu dimostrato da cambiamenti nelle relazioni di associazione tra i geni in Drosophila e fu confermato dall'analisi dei cromosomi giganti delle ghiandole salivari. Dalla sequenza delle bande trasversali in questi cromosomi e dal tipo di appaiamento tra gli omologhi negli eterozigoti strutturali si poteva infatti stabilire con precisione la natura del cambiamento strutturale. Questi riordinamenti includevano scambi di segmenti tra cromosomi non omologhi (traslocazione reciproca) e inversioni di un segmento entro un cromosoma (inversione). Già era stato osservato che cambiamenti di questo tipo avvenivano spontaneamente con bassa frequenza, cosicché, come per le mutazioni, l'effetto dei raggi X era quello di aumentarne la frequenza.
Studi condotti da Muller, T. S. Painter e T. Dobzhansky, utilizzando cambiamenti strutturali indotti da raggi X in Drosophila, confermarono uno dei postulati di base della teoria cromosomica e precisamente che la sequenza dei geni in una mappa di associazione, stabilita sulla base delle frequenze di ricombinazione, corrispondeva alla sequenza fisica dei componenti del corrispondente cromosoma. Una scoperta sorprendente, tuttavia, fu che le dimensioni fisiche dei segmenti cromosomici trasferiti in una nuova posizione non erano necessariamente proporzionali alle loro dimensioni nella mappa di associazione. Si dovette pertanto concludere che il crossing-over era più frequente in certe regioni del cromosoma rispetto ad altre, dato che le regioni con alte frequenze di crossing-over sarebbero apparse più ampie nella mappa di associazione rispetto a quelle con basse frequenze.
Dopo la scoperta che l'azotoiprite causava mutazioni, si giunse anche a dimostrare (v. Auerbach e Robson, 1947) che questo gas induceva anche riordinamenti strutturali. Molte altre sostanze chimiche mostrarono di possedere questa proprietà, ed inoltre nel 1947 J. M. Thoday e J. Read scoprirono che la frequenza dei riordinamenti strutturali indotti da raggi X era grandemente ridotta in assenza di ossigeno. Era così evidente che nell'induzione di questi cambiamenti da parte delle radiazioni ionizzanti erano implicate, in qualche maniera, anche delle reazioni chimiche. Kihlman nel 1961 classificò gli agenti fisici e chimici in grado di indurre riordinamenti strutturali, nella seguente maniera: a) ad azione indipendente dalla concentrazione di ossigeno, per es. particelle α, luce ultravioletta, azotoiprite; b) ad azione molto ridotta in assenza di ossigeno, ma insensibili agli inibitori della respirazione, per es. raggi X; c) ad azione molto ridotta in assenza di ossigeno o in presenza di inibitori della respirazione, per es. idrazide maleica.
Nonostante la diversità nell'azione iniziale, tutti questi agenti, con poche eccezioni, producevano uno spettro di cambiamenti strutturali molto simile. Sin da quando si scoprì che i raggi X potevano indurre cambiamenti strutturali, vi sono state diversità di opinioni sulla natura degli agenti mutageni a livello cromosomico. Una peculiarità dei riordinamenti strutturali, infatti, comunque indotti, è che essi quasi invariabilmente implicano rotture in due punti, e che il ricongiungimento avviene con nuovo assetto tra i frammenti cromosomici, senza ricostituzione delle strutture originarie.
Due sono state le ipotesi principali proposte per spiegare questi fenomeni: 1) rottura e ricostituzione: secondo questa ipotesi gli eventi iniziali sono delle rotture in due punti diversi della struttura cromosomica, seguite da ricongiungimento, al quale non possono partecipare le terminazioni dell'ordinamento normale; 2) scambio non omologo: una serie di eventi viene iniziata in due punti diversi, con il risultato di uno scambio di segmenti cromosomici. Si pensa che questo scambio possa essere paragonabile al normale crossing-over, con la sola differenza che esso interverrebbe tra punti non corrispondenti di cromosomi omologhi.
Quest'ultima ipotesi ha il vantaggio di spiegare come mai le terminazioni ‛normali' dei frammenti cromosomici non partecipino al ricongiungimento. L'ipotesi dello scambio è tra l'altro avvalorata dai risultati ottenuti da S. H. Revell nel 1959 sulle frequenze relative dei diversi tipi di variazioni strutturali e dalla relazione tra dose di raggi X e frequenza di rotture cromatidiche.
e) Modalità d'azione del gene
Un altro problema fondamentale dell'ereditarietà mendeliana è quello di come i geni determinino i particolari caratteri cui ciascuno di essi si riferisce. Subito dopo la riscoperta del lavoro di Mendel nel 1900, Garrod (v., 1909) e Bateson (v., 1909) suggerirono che vi potesse essere una qualche correlazione tra geni ed enzimi. Gli enzimi sono costituiti da proteine e la loro azione consiste nel catalizzare specifiche reazioni biochimiche (v. enzimi). Garrod aveva studiato, tra l'altro, una particolare malattia umana chiamata alcaptonuria che è determinata dall'incapacità di metabolizzare completamente la fenilalanina e la tirosina. Egli suggerì che molto probabilmente questa incapacità dovesse essere spiegata con la mancanza dell'enzima necessario: una previsione che fu del tutto convalidata qualche anno più tardi. Bateson e E. R. Saunders avevano intanto dimostrato, nel 1902, sulla base dei dati raccolti da Garrod, che l'alcaptonuria veniva trasmessa secondo gli schemi dell'ereditarietà mendeliana e che era determinata da un singolo gene recessivo.
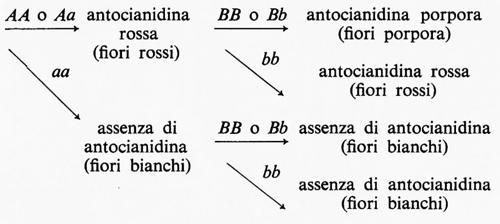
Bateson e collaboratori inoltre, lavorando sull'ereditarietà del colore dei fiori, raccolsero alcune prove a favore dell'ipotesi di una relazione tra geni ed enzimi. Essi condussero esperimenti su molte specie di piante con fiori di diverso colore ed in particolare su Lathyrus odoratus (pisello da giardino) e Salvia horminum. Tutti i casi esaminati mostrarono di uniformarsi agli schemi dell'ereditarietà mendeliana e in generale le diverse variazioni di colore in una specie erano trasmesse indipendentemente, come se fossero determinate da geni localizzati in diversi cromosomi o in regioni molto distanti dello stesso cromosoma. In molti casi, tuttavia, si verificavano delle interazioni tra i diversi geni o, molto più probabilmente, tra i loro prodotti. Per esempio, un gene non mostrava il suo effetto quando si trovava in presenza di un particolare allele di un altro gene. Quest'ultimo venne definito da Bateson nel 1907 ‛epistatico', mentre il primo fu detto ‛ipostatico'. Una plausibile spiegazione per tale interazione tra due geni diversi, o tra i loro prodotti, è quella di supporre che i geni siano implicati nel controllo di tappe sequenziali necessarie alla sintesi del pigmento nella corolla dei fiori. Se il gene epistatico controlla un processo precedente a quello controllato dal gene ipostatico, ne deriva che in presenza di un particolare allele del gene epistatico non si può verificare un determinato processo iniziale. Questo significherebbe che il substrato per l'azione del gene ad azione successiva o ipostatico non verrà prodotto, e quindi che gli alleli di questo gene rimarranno senza effetto. Così, si supponga che il gene ad azione iniziale A conduca alla formazione di una antocianidina rossa e che il gene ad azione successiva B agisca su questa antocianidina in maniera da modificarla a color porpora. In un omozigote aa, quando a sia un allele recessivo del gene iniziale che porterà all'incapacità di produrre antocianidina, i fiori saranno bianchi. Data questa circostanza, il gene B sarà incapace di produrre il pigmento porpora e questo accadrà sia nel caso che questo allele sia presente, sia che l'individuo sia omozigote bb, essendo b un allele difettivo: in entrambi i casi non vi sarà differenza e i fiori saranno bianchi.
Bateson e collaboratori nel 1905 trovarono che questa situazione si verificava in Salvia horminum. Vi sono tuttavia altre spiegazioni possibili per l'interazione genica e, prima di concludere definitivamente che i geni A e B agiscono in quell'ordine in punti diversi dello stesso processo metabolico, sarebbe necessario indentificare le singole reazioni biochimiche che si succedono. Fu molto importante, tuttavia, mettere in evidenza come questi risultati si trovassero in buon accordo con l'ipotesi di una relazione tra geni ed enzimi. Molti altri studi sull'ereditarietà della pigmentazione, sia nelle piante sia negli animali, si dimostrarono a favore di questa ipotesi anche se non riuscirono a fornire una chiara prova della sua validità.
Un importante progresso fu compiuto quando G. W. Beadle e E. L. Tatum (v., 1941) decisero di accertare se i geni controllassero delle reazioni biochimiche conosciute. Tutti gli studi precedenti avevano tentato di trovare le basi biochimiche per delle differenze genetiche ben note. Beadle e Tatum lavorarono con il fungo Neurospora crassa e trovarono che questo organismo poteva crescere su un terreno sintetico, che definirono terreno minimo, contenente solamente sali inorganici, un carboidrato e biotina; i due ricercatori conclusero che il fungo poteva sintetizzare ogni altra sostanza necessaria alla sua crescita, come amminoacidi e altre vitamine. Conidi del fungo vennero trattati con raggi X e vennero quindi usati per fecondare gli organi riproduttivi femminili di un ceppo diverso. Le spore ottenute come progenie furono quindi isolate e fatte crescere in un terreno arricchito contenente malto ed anche estratto di lievito quali sostanze capaci di soddisfare le richieste essenziali. Ciascuna linea isolata fu quindi trasferita nel terreno minimo. Di 2.000 ceppi-progenie saggiati, 3 si dimostrarono incapaci di crescere nel terreno minimo. Una serie di prove sistematiche, effettuate aggiungendo una alla volta varie sostanze, dimostrò che ciascuno di questi mutanti aveva perduto la capacità di sintetizzare una specifica sostanza; rispettivamente piridossina, tiammina ed acido p-amminobenzoico. Inoltre, ognuna di queste deficienze era ereditata come una caratteristica dovuta alla differenza in un singolo gene rispetto al normale.
Un vantaggio ulteriore di Neurospora per lo studio dell'azione del gene fu sottolineato da Beadle e collaboratori nel 1944. Il micelio del fungo può formare facilmente un eterocarionte se due diversi ceppi della stessa specie vengono fatti crescere insieme. In un eterocarionte nuclei di diversi genotipi si trovano insieme nella stessa cellula. Quando dei mutanti con richiesta di acido p-amminobenzoico e acido nicotinico venivano inoculati a stretto contatto nello stesso terreno minimo si formava un eterocarionte che cresceva normalmente in quanto ciascuno dei due mutanti suppliva alla deficienza dell'altro.
Questa tecnica venne applicata anche a mutanti di origine indipendente, che sembravano avere la stessa deficienza, per esempio, una richiesta di triptofano. Si trovò così che alcuni mutanti per triptofano erano allelici e cioè non erano in grado di completarsi reciprocamente, mentre altri risultavano non allelici. Una conferma della validità del saggio di allelismo mediante l'eterocarionte si ottenne da studi sulla progenie: i mutanti allelici erano causati da mutazioni indipendenti dello stesso gene, quelli non allelici da diversi geni che venivano ereditati generalmente in maniera indipendente. Studi più dettagliati sui mutanti non allelici mostrarono che le mutazioni riguardavano reazioni biochimiche diverse. Per esempio, fra i mutanti per il triptofano, uno sembrava bloccato nella sintesi di acido antranilico, in grado quindi di utilizzare questa sostanza o l'indolo al posto del triptofano, mentre un secondo mutante poteva utilizzare l'indolo ma non l'acido antranilico. La dimostrazione che quest'ultimo mutante accumulava acido antranilico nei suoi tessuti, confermò che non si svolgeva la reazione da acido antranilico ad indolo.
In una maniera molto simile A. M. Srb e N. H. Horowitz dimostrarono che per la sintesi dell'arginina era necessaria tutta una serie di reazioni e che singoli geni controllavano ciascuna reazione.
Alla luce di questi studi Beadle (v., 1945) formulò l'ipotesi ‛un gene-un enzima'. Egli suggerì che ogni reazione biochimica venisse controllata da uno specifico gene e che la funzione primaria dei geni - e quasi certamente la loro sola funzione - fosse quella di determinare la configurazione delle molecole proteiche. Nel giro di pochi anni si accumularono gli esempi, tratti da una grande varietà di organismi, di mutanti privi di uno specifico enzima, o forniti di una sua forma modificata. La natura della relazione tra geni ed enzimi è oggi conosciuta abbastanza dettagliatamente e verrà discussa più avanti.
La scoperta di Beadle e Tatum sui mutanti biochimici in Neurospora condusse ad altre importanti scoperte. C. H. Gray e Tatum nel 1944, mediante irradiazioni con raggi X, ottennero dei mutanti simili nel batterio Escherichia coli e questo permise a J. Lederberg e E. L. Tatum (v., 1946) di dimostrare la ricombinazione genetica in Escherichia coli. Partendo da queste basi si sviluppò l'intero capitolo della genetica dei batteri e dei batteriofagi e sorse così la scienza della biologia molecolare. Risultò che i batteri, sebbene siano privi di cromosomi normali, possiedono geni che controllano singole reazioni biochimiche nella stessa maniera di quanto avviene negli organismi con cromosomi, quali i funghi. Inoltre fu possibile dimostrare che i mutanti batterici, similmente a quelli dei funghi, erano spesso privi di un enzima specifico.
5. Conoscenze biochimiche e problemi dell'ereditarietà
a) Le basi chimiche dell'ereditarietà
Nei primi decenni del XX secolo si accettò per dimostrato il concetto che le basi chimiche dell'ereditarietà fossero le proteine. Si sapeva che i cromosomi erano composti da proteine ed acido desossiribonucleico (DNA), quest'ultimo identificato per la prima volta da Miescher verso la fine del XIX secolo (v. acidi nucleici; v. biologia). Tuttavia non si ritenne che il DNA fosse importante perché dalla sua idrolisi si ricavarono solamente due purine - adenina e guanina - e due pirimidine - citosina e timina -, lo zucchero desossiribosio e acido fosforico. Non sembrava probabile che il DNA fosse la base chimica dell'ereditarietà perché si riteneva che la sua struttura, dati questi costituenti, fosse alquanto uniforme. D'altro canto si sapeva che le proteine presentavano una grande diversità di strutture e che erano costituenti di enzimi specifici. Era quindi ragionevole supporre, sulla base delle prove di una relazione tra geni ed enzimi, che anche i geni fossero costituiti da proteine. Non si sapeva tuttavia come venissero sintetizzate le proteine negli organismi viventi. Infatti, se in qualità di enzimi esse erano in grado di catalizzare tutte le reazioni necessarie alla sintesi di altre sostanze nella cellula, era difficile immaginare come potessero catalizzare la propria sintesi.
La prima prova che contraddiceva questa ipotesi sulla natura proteica del gene fu ottenuta da E. Knapp e altri (v., 1939), che condussero una serie di ricerche sulla lunghezza d'onda della luce ultravioletta (UV) in grado di causare mutazioni nell'epatica Sphaerocarpos donnellii: trattarono gli spermatozoi con UV e trovarono che la mutagenicità massima si aveva con lunghezza d'onda di 265 nm, il che corrispondeva alla lunghezza d'onda assorbita dal DNA. Knapp e collaboratori furono molto cauti nelle loro conclusioni, ma i loro esperimenti indicavano chiaramente che il DNA poteva essere considerato la base molecolare dei processi ereditari.
La prova conclusiva concernente il DNA fu ottenuta da O. T. Avery, C. M. MacLeod e M. McCarty (v., 1944) con lo studio del fenomeno della trasformazione genetica nel batterio Diplococcus pneumoniae (Pneumococcus). La trasformazione era stata scoperta da F. Griffith nel 1928. Questi aveva dimostrato che un ceppo virulento con colonie lisce, se veniva riscaldato fino all'uccisione delle cellule, e quindi mescolato con un ceppo non virulento a colonie ruvide, poteva rigenerare un ceppo virulento liscio quando questa miscela di cellule venisse inoculata in topi: il carattere virulenza era in seguito ereditato in maniera indefinita. Avery e altri dimostrarono che la sostanza responsabile di questa trasformazione genetica era il DNA, dopo essere riusciti ad isolare il ‛principio trasformante' in una forma altamente purificata.
Un terzo esperimento che dimostrava come il materiale ereditario fosse composto da DNA e non da proteine, venne eseguito da A. D. Hershey e M. Chase nel 1952 utilizzando un batteriofago del tipo n. 2 (T2) di Escherichia coli. Essi marcarono le proteine del virus T2 con zolfo radioattivo (35S) quindi infettarono le cellule ospiti con le particelle virali. In seguito ad agitazione in un frullatore ad alta velocità, almeno l'80% dello zolfo marcato venne rimosso dalle pareti cellulari insieme all'involucro proteico delle particelle virali, che rimasero all'esterno del batterio dopo averlo infettato. Inoltre, ben poco o nulla dello zolfo marcato si trovò nelle particelle virali della progenie formatasi all'interno della cellula ospite. Al contrario, quando l'esperimento fu ripetuto usando del 32P per marcare il DNA virale, si trovò che la maggior parte del fosforo penetrava nell'interno delle cellule ospiti e non veniva quindi rimosso dall'agitazione, mentre il 30% o più lo si poteva ritrovare nelle particelle virali costituenti la progenie.
Queste osservazioni confermarono le conclusioni a cui si era pervenuti con gli studi chimico-fisici e con l'osservazione dell'aspetto delle particelle virali al microscopio elettronico. Le particelle mostravano una testa esagonale, composta da un involucro proteico racchiudente una massa di DNA e una coda, anch'essa di composizione proteica, con un sottilissimo canale centrale. Il contenuto di DNA, della testa sembrava venir iniettato all'interno della cellula batterica al momento dell'infezione passando attraverso la coda, mentre l'involucro proteico rimaneva all'esterno del batterio. Kleinschmidt e collaboratori nel 1962 confermarono che l'interno della testa del fago consisteva di DNA, in quanto riuscirono a dimostrare che, in seguito a shock osmotico, il DNA fuorusciva dalla testa del fago e poteva essere raccolto su una pellicola di proteine galleggianti sull'acqua. Al microscopio elettronico si poteva osservare che dalla testa del fago emergeva un lunghissimo filamento di DNA ripiegato in una serie di anse. Con opportune misurazioni fu dimostrato che questo filamento era lungo circa 55μm, sebbene la testa del virus non superasse 100×65 nm. Evidentemente il DNA si trova ripiegato molte volte e strettamente impaccato entro l'involucro della testa del fago.
R. Vendrely e C. Vendrely nel 1948 scoprirono che il contenuto di DNA dei nuclei di diversi tessuti di Bos taurus (bovini domestici) era di 6,5 pg, mentre quello dei nuclei degli spermatozoi era di 3,4 pg. Questa quantità costante nei nuclei dei tessuti diploidi e il suo dimezzamento in quelli degli spermatozoi aploidi era quanto ci si attendeva nel caso che il DNA fosse il materiale genetico. L'anno successivo, A. E. Mirsky e H. Ris confermarono queste osservazioni in un gran numero di specie e trovarono che diverse specie di animali differivano grandemente nel loro contenuto di DNA nucleare. E. Chargaff e collaboratori, nello stesso anno, scoprirono che diversi tessuti di Bos taurus avevano la stessa composizione chimica di DNA, ma che il DNA di due altri organismi e precisamente Saccharomyces cerevisiae (lievito) e Mycobacterium tuberculosis avium, aveva una diversa composizione. Evidentemente, ciascuna specie aveva un caratteristico contenuto e composizione di DNA nucleare, che era indipendente dal tessuto di origine dei nuclei (a parte il contenuto dimezzato degli spermatozoi). Le differenze nella composizione riscontrate da Chargaff e collaboratori riguardavano le proporzioni relative delle quattro basi azotate (adenina, guanina, citosina e timina) recuperate dall'idrolisi del DNA. Chargaff (v., 1950) inoltre attirò l'attenzione dei ricercatori su una peculiarità della composizione in basi del DNA: l'adenina (A) e la timina (T) si trovavano in quantità equimolecolari, e similmente avveniva per la guanina (O) e la citosina (C). Le differenze tra le specie riguardavano le proporzioni relative delle coppie di basi A-T e G-C.
J. D. Watson e F. H. C. Crick (v., A structure..., 1953) proposero una struttura molecolare per il DNA che tenesse conto delle osservazioni di Chargaff. Essi suggerirono che la molecola consistesse di due catene di nucleotidi avvolte ad elica una sull'altra, tenute insieme da legami a idrogeno tra l'adenina di una catena e la timina dell'altra, e similmente tra guanina e citosina. Nella struttura da loro proposta per il DNA, le basi si trovano disposte perpendicolarmente all'asse dell'elica e le differenze tra le specie consisterebbero nella diversità tra le sequenze di coppie di basi lungo la molecola. Le due catene sarebbero quindi formate da sequenze di basi complementari. La sequenza delle basi, in tal modo, non solo potrebbe costituire la specificità del gene, ma il processo di replicazione del DNA prima della divisione nucleare potrebbe consistere nella separazione delle due catene e nella sintesi non del duplicato delle due eliche, ma del complemento di ciascuna di esse. Lo straordinario e grandissimo impatto del modello di Watson e Crick sulle idee dei genetisti fu una conseguenza di queste due nuove ipotesi per spiegare la specificità dei geni e il loro modo di replicazione (v. acidi nucleici; v. biologia; v. genetica: Citogenetica).
Watson e Crick erano stati indotti a proporre la loro ipotesi sulla struttura del DNA dal lavoro di cristallografia ai raggi X di M. H. F. Wilkins, R. E. Franklin e collaboratori, i quali, indipendentemente da loro, conclusero che vi erano due catene avvolte ad elica, e che le basi si trovavano all'interno della molecola dato che il DNA di diverse specie, e quindi di diversa composizione di basi, dava lo stesso tipo di diffrazione ai raggi X.
Secondo il modello di Watson e Crick, il DNA ha la configurazione di una scala a pioli avvolta a spirale, con gli scalini formati dalle coppie di basi (A-T, T-A, G-C o C-G) tenute insieme da legami a idrogeno, mentre i montanti della scala consistono di fosfato e zucchero (desossiribosio) alternantisi, tenuti insieme da legami covalenti 3′, 5′-fosfodiestere (3′ e 5′ si riferiscono agli atomi di carbonio nella molecola asimmetrica del desossiribosio a cui sono attaccati i radicali fosforici). Lungo uno dei due montanti i legami 3′, 5′ decorrono in direzione ascendente mentre nell'altro hanno direzione opposta, discendente, così che ciascun montante con le basi attaccate alle molecole di zucchero sarà invertito rispetto all'altro (v. fig. 3). L'appaiamento specifico delle basi (A-T, G-C) si pensa sia imposto da considerazioni di spazio tra i due montanti della scala a chiocciola formata dai regolari legami fosfodiestere. Le dimensioni della molecola sono di 2 nm di spessore, mentre un giro della spirale destrorsa comprende 10 coppie di basi e corrisponde a 3,4 nm di lunghezza. La lunghezza totale della molecola è indefinita ma comunque molto grande.
Il modello fu sottoposto all'analisi sperimentale della diffrazione a raggi X e fu confermato entro pochi anni dalla data in cui era stato proposto. Sulla base di questi ultimi studi, dovettero essere apportate al modello solamente delle piccole modificazioni di dettaglio. Tuttavia, le ipotesi sulla specificità del gene e sulla maniera della sua replicazione non erano necessariamente valide e le prove su questi due punti verranno discusse qui di seguito.
b) Il processo di duplicazione del materiale genetico
Il metodo di replicazione del DNA proposto da Watson e Crick deve esser considerato a due livelli di analisi. Essi suggerirono che le due catene del DNA si separassero e che lungo ciascuna di esse se ne sintetizzasse una nuova complementare. Questo metodo di replicazione fu definito da M. Delbrück e G. S. Stent nel 1957 come di tipo semiconservativo in quanto le due molecole figlie consisterebbero ciascuna di una catena vecchia derivante dalla molecola parentale e di una nuova appena sintetizzata. Ad un livello di analisi più spinto, Watson e Crick suggerirono che le due catene si separassero ad una estremità della molecola e che una nuova catena venisse sintetizzata lungo ciascuna delle vecchie catene nella stessa direzione fisica procedendo dal punto di separazione. Questo potrebbe essere paragonato all'aprirsi di una chiusura lampo e al contemporaneo formarsi di due nuove chiusure lungo ciascuna delle due metà aperte. Tuttavia, poiché le due catene nucleotidiche di una molecola di DNA hanno opposte polarità per quanto riguarda i loro assi di legame fosfodiesterici, le due nuove catene sintetizzate nella stessa direzione fisica procedendo dall'apertura avrebbero ciascuna una sintesi chimica di direzione opposta: una da 3′ a 5′ e l'altra da 5′ a 3′, e la replicazione procederebbe in maniera continua in ciascuna di esse (v. fig. 4, a). Ci possiamo pertanto porre due domande: 1) la replicazione del DNA è veramente semiconservativa? 2) procede contemporaneamente in entrambe le direzioni chimiche, si da consentire la replicazione continua in una direzione fisica?
La prima prova in appoggio alla replicazione di tipo semiconservativo fu ottenuta da J. H. Taylor e altri (v., 1957). Era stato dimostrato da A. Howard e S. R. Pelč nel 1951 che negli apici radicali di Vicia faba a temperatura ambiente le divisioni nucleari in una cellula si succedono ogni 24 ore e che la replicazione del DNA si compie entro un periodo di circa 8 ore durante l'interfase e termina 10 ore prima della successiva metafase. Taylor e altri fecero crescere degli apici radicali di Vicia faba in una soluzione di timidina triziata per 8 ore, quindi li lavarono accuratamente e 10 ore più tardi li fissarono, colorarono e schiacciarono su vetrini: in contatto con le cellule così trattate misero infine della emulsione fotografica per alcune settimane. La timidina e incorporata specificamente nel DNA e il trizio emette particelle β che impressionano l'emulsione fotografica al loro passaggio. Taylor e collaboratori trovarono che entrambi i cromatidi di ciascun cromosoma metafasico erano stati marcati dal trizio. Se gli apici venivano fatti crescere per altre 24 ore prima della fissazione, interveniva un'altra replicazione che però si verificava in assenza di timidina triziata. Taylor e collaboratori constatarono che, dopo questa seconda duplicazione, un cromatidio di ciascun cromosoma metafasico risultava marcato mentre l'altro era privo di marcatura (v. fig. 5). Questo è proprio quanto ci si dovrebbe attendere se l'asse del cromosoma consistesse in un'unica molecola gigante di DNA e se la sua duplicazione fosse di tipo semiconservativo. Quando fu realizzata questa scoperta, molti citologi si mostrarono riluttanti ad ammettere che l'asse di un cromosoma consistesse di una sola molecola di DNA. I dati di Taylor non escludono infatti alternative più complesse come quella di un cromosoma con molte molecole di DNA parallele tra loro disposte perpendicolarmente all'asse: ciascuna di queste molecole avrebbe una replicazione semiconservativa, con la caratteristica che ciascuna delle nuove molecole passerebbe ad un cromatidio, mentre tutte le vecchie rimarrebbero associate all'altro cromatidio fratello. Anche altre modalità di replicazione per la molecola di DNA potrebbero essere compatibili con le osservazioni di Taylor, ammesso che il vecchio materiale si separi dal nuovo al momento della divisione dei cromatidi.
Esperimenti più recenti, che discuteremo più avanti, hanno ormai fornito prove piuttosto convincenti che l'asse del cromosoma consiste di una sola molecola di DNA. La scoperta di Taylor della replicazione semiconservativa del cromosoma ci fornisce oggi pertanto anche la prova della duplicazione semiconservativa del DNA, anche se la prima prova diretta di questo fu ottenuta da M. Meselson e F. W. Stahl (v., 1958). Questi ricercatori fecero crescere il batterio Escherichia coli per un certo numero di divisioni cellulari in un terreno di cultura in cui tutte le fonti di azoto consistevano nel suo isotopo pesante 15N. I batteri vennero quindi trasferiti in un terreno contenente del normale 14N e a intervalli successivi di tempo, corrispondenti a una divisione cellulare, furono prelevati dei campioni che vennero posti in sospensione in una soluzione di cloruro di cesio con un gradiente di densità ottenuto in una ultracentrifuga. Si trovò così che dopo una duplicazione nel terreno con azoto leggero (14N), il DNA aveva una densità intermedia, e che dopo due duplicazioni metà era di tipo intermedio e metà di tipo leggero (v. fig. 6). Questo è quanto ci si attende da una duplicazione di tipo semiconservativo. Dato che con il riscaldamento le due catene nucleotidiche del DNA si separano, Meselson e Stahl riscaldarono il DNA di densità intermedia ed ottennero la ricostituzione di molecole di DNA pesante e leggero: come ci si attendeva nel caso che le molecole a densità intermedia consistessero di una catena nucleotidica leggera e di una catena pesante.
Nello stesso anno in cui Meselson e Stahl dimostrarono che il DNA si replicava in maniera semiconservativa, A. Kornberg e collaboratori (v. Kornberg, 1960) isolarono un enzima di Escherichia coli, la DNA-polimerasi, capace di catalizzare la sintesi di DNA in vitro. L'enzima aveva molte delle proprietà previste dall'ipotesi di Watson e Crick: per esempio era necessaria come modello (templa- te) una catena nucleotidica e la catena sintetizzata dall'enzima risultava complementare a quella. Inaspettatamente l'enzima operava solo in una direzione chimica aggiungendo nucleosidi 5′-fosfato alla terminazione 3′-idrossile di una catena già iniziata. Più recentemente si è potuto accertare che l'enzima di Kornberg si occupa di riparare eventuali danni presenti nel DNA, e non è quindi responsabile della normale duplicazione. Questo è stato messo in evidenza da un lavoro di P. De Lucia e J. Cairns del 1969 in cui si dimostra come un mutante di Escherichia coli difettivo per questo enzima possa ciononostante replicare il proprio DNA normalmente. Il problema è pertanto ancora aperto su come avvenga la normale duplicazione del DNA: se in ambedue le direzioni chimiche, come supposto originariamente da Watson e Crick, o se soltanto in una direzione, come avviene con l'enzima per la riparazione di Kornberg.
R. Okazaki e collaboratori, sin dal 1968, hanno studiato la lunghezza delle catene nucleotidiche di DNA di nuova sintesi e le hanno trovate piuttosto corte, composte da 1.000-2.000 nucleotidi all'incirca. Essi pertanto sono in favore di una sintesi discontinua, tutta in una sola direzione chimica con l'unione successiva di corti segmenti mediante la formazione dei necessari legami fosfodiestere (v. fig. 4, b-c). Altri autori, per esempio R. Werner nel 1971, hanno suggerito che i segmenti di Okazaki siano dovuti alla necessità di punti di rotazione per lo svolgimento della doppia elica, o alternativamente siano da mettere in rapporto con l'attività genica piuttosto che con la duplicazione del DNA.
J. W. Drake e E. O. Greening scoprirono nel 1970 che la DNA-polimerasi del virus T4 di Escherichia coli riveste una grande importanza nell'induzione di mutanti da sostanze chimiche. In ceppi del virus caratterizzati da forme mutanti dell'enzima di replicazione, la mutazione ad opera di certe sostanze veniva soppressa. Drake e Greening spiegarono questo risultato ritenendo che l'enzima fosse implicato nella selezione dei nucleotidi giusti per una duplicazione accurata.
Numerosi esperimenti hanno dimostrato che il DNA dei Batteri si duplica progressivamente da un capo all'altro della molecola e, sorprendentemente, che questi due punti sono uniti si che la molecola risulta circolare. Questo è stato dimostrato per la prima volta da J. Cairns (v., 1963) utilizzando la tecnica di marcatura del DNA con timidina triziata, estraendo la molecola intatta dalle cellule e preparando delle autoradiografie. Egli trovò così che il DNA di Escherichia coli forma un cerchio la cui circonferenza è approssimativamente di 1 mm e che al punto di replicazione si può notare una forcella, con nuove catene di nucleotidi sintetizzate lungo ciascuna ramificazione, come ci si attende secondo il modello di replicazione semiconservativa. Il potere risolvente della tecnica di marcatura con il trizio (3H) non è tuttavia sufficiente a distinguere tra la replicazione continua in entrambe le direzioni chimiche ed il processo necessanamente discontinuo qualora si verificasse in una sola direzione chimica. Per permettere la rotazione della doppia elica necessaria a separare le due catene di nucleotidi prima della replicazione, è necessario, nel caso di una molecola circolare di DNA, supporre che vi sia almeno una rottura in uno dei due filamenti.
F. Jacob e S. Brenner nel 1963 introdussero il termine ‛replicone' per indicare un segmento discreto di DNA che si replichi come un'unità. Nei batteri e nei loro virus l'intera quantità del DNA costituisce un singolo replicone, mentre negli eucarioti, a differenza dei procarioti, vi sono numerosi repliconi che si susseguono lungo la struttura di ciascun cromosoma. Questo fu dimostrato per la prima volta da J. H. Taylor nel 1960 utilizzando cellule di Cricetulus griseus (hamster cinese) coltivate in vitro. La marcatura con trizio ha dimostrato che ciascun cromosoma ha una sua caratteristica distribuzione di regioni che si replicano precocemente o tardivamente. Cairns ha estratto DNA marcato con trizio da cromosomi di Mammiferi ed ha osservato che regioni a duplicazione successiva erano collegate estremità a estremità in successione lineare tra loro. La natura del replicone negli eucarioti sarà discussa ulteriormente più avanti.
c) Sequenza dei nucleotidi e sequenza degli amminoacidi
L'idea che la specificità del gene risiedesse nella sequenza dei nucleotidi lungo la catena nucleotidica del DNA, fu dapprima suggerita nel 1952 da E. Chargaff e collaboratori contemporaneamente a A. L. Dounce, e ricevette notevole appoggio nell'anno successivo dalla teoria di Watson e Crick sulla struttura e replicazione del DNA. Infatti, due catene di nucleotidi complementari, replicantisi con la formazione di nuovi filamenti a loro complementari mediante un meccanismo di modello-stampo, sono il presupposto ideale per mantenere costante una sequenza di basi.
Dounce (v., 1952) e G. Gamow (v., 1954) combinarono questa ipotesi della sequenza delle basi come fondamento della specificità del gene, con l'ipotesi ‛un gene-un enzima' di Garrod (v., 1909), Bateson (v., 1909) e Beadle (v., 1945). Essi suggerirono che la sequenza di nucleotidi del DNA potesse specificare la sequenza degli amminoacidi nelle proteine. Si sa che le molecole proteiche consistono di uno o più polipeptidi, e che un polipeptide è costituito dalla successione lineare di amminoacidi uniti tra loro mediante la formazione di legami peptidici tra il gruppo carbossilico (−COOH) di un amminoacido ed il gruppo α-amminico (−NH2) del successivo. Nelle proteine sono presenti 20 amminoacidi e quindi l'ipotesi di Dounce-Gamow implicava che la sequenza delle 4 basi nell'acido nucleico determinasse la sequenza dei 20 diversi amminoacidi nei polipeptidi. Sia Dounce che Gamow suggerirono che una sequenza di tre nucleotidi consecutivi specificasse un amminoacido, dato che con tre si ottengono 64 possibili sequenze, mentre con due si formano solo 16 possibili sequenze che sono insufficienti. Questa ipotesi del codice genetico solleva due fondamentali questioni: a) la costituzione del gene è lineare e contiene numerosi siti di possibili mutazioni? b) la mutazione altera la sequenza degli amminoacidi?
Come già descritto, alcuni anni prima era stata ottenuta la prova che potevano verificarsi eventi di ricombinazione intragenica. I dati erano stati forniti dai lavori di E. B. Lewis nel 1941 con i mutanti star in Drosophila e questo implicava che le mutazioni potessero verificarsi in diversi siti all'interno di un gene. Ulteriori dettagliate informazioni sull'organizzazione del gene derivarono dalla scoperta di G. W. Beadle e E. L. Tatum (v., 1941) sui mutanti della Neurospora con specifiche richieste nutrizionali per la crescita. Fu infatti possibile selezionare ricombinanti entro un altissimo numero di individui progenie con genotipo parentale, dato che quando vengono incrociati due mutanti allelici a richiesta nutrizionale, soltanto la progenie ricombinante può crescere su terreno minimo. Esperimenti di questo tipo furono compiuti per la prima volta da G. Pontecorvo e dai suoi collaboratori, J. A. Roper e R. H. Pritchard, utilizzando il fungo Aspergillus nidulans: i risultati dimostrarono che il gene era di costituzione lineare (v. gene). Le indicazioni sulla linearità si basarono sulla frequenza di ricombinanti allelici e sul comportamento dei geni marcatori esterni a loro associati, nella stessa maniera utilizzata da Lewis per determinare la posizione relativa dei due mutanti del gene star in Drosophila. Inoltre, da questi dati, non solo si poté stabilire che il gene era lineare, ma altresì che esso era colineare con il cromosoma.
I lavori di Roper e Pritchard dimostrarono inoltre che quando due mutazioni entro un gene, di origine indipendente, si trovano in posizioni diverse, esse inattivano l'intero gene, determinando la richiesta di uno specifico fattore nutrizionale. Evidentemente l'unità funzionale del materiale ereditario appariva più valida che non quella del materiale mutazionale. Il lavoro di Roper e Pritchard riguardava un gene per la sintesi dell'adenina in Aspergillus. Questi autori dimostrarono che un diploide con due diverse mutazioni in quel gene, una in ciascuno dei due cromosomi omologhi, presentava richiesta di adenina nel terreno, mentre un diploide con ambedue le mutazioni in un cromosoma ed un gene normale nell'altro, non richiedeva adenina. Osservazioni simili erano state fatte da Lewis con i mutanti dell'occhio star e da M. M. Green e K. C. Green con i mutanti dell'occhio lozenge in Drosophila. Questi costituivano degli esempi di effetto di posizione, poiché ciascun diploide conteneva lo stesso materiale genetico, disposto tuttavia in maniera diversa. Ed erano diversi tuttavia dall'esempio originario fornito da Sturtevant con l'occhio bar, poiché star e lozenge non erano associati a duplicazioni di un segmento cromosomico. Al nuovo fenomeno fu dato il nome di ‛effetto di posizione cis-trans', seguendo la proposta di Haldane del 1941 secondo la quale i termini cis e trans, in analogia con il loro uso in chimica, dovevano essere applicati ai genotipi isomerici con due mutazioni in un cromosoma e nessuna nell'altro (cis) e con una in ciascuno (trans).
La possibilità di selezionare ricombinanti all'interno di un gene da una numerosa popolazione di genotipi parentali, che Pontecorvo e collaboratori utilizzarono con l'Aspergillus, venne anche impiegata da Benzer con il batteriofago T4 di Escherichia coli. S. Benzer (v., 1955) ottenne dei mutanti (denominati mutanti rII) di questo virus che potevano svilupparsi nel ceppo B, ma non in quello K, dell'ospite. I mutanti potevano essere incrociati fra di loro infettando l'ospite B e si potevano quindi selezionare i ricombinanti capaci di svilupparsi nelle cellule K. Si osservò che le frequenze di ricombinazione tra i vari mutanti davano luogo a una mappa lineare. Provocando infezioni miste dei mutanti nell'ospite K, Benzer trovò che i mutanti potevano essere raggruppati in due gruppi di complementazione. Quelli che mappavano in una regione localizzata a sinistra erano incapaci di supplire reciprocamente alle loro deficienze, e similmente avveniva per quelli che mappavano alla loro destra. D'altra parte, invece, due mutanti appartenenti ai due diversi gruppi (chiamati A e B) potevano complementare reciprocamente e rendevano possibile lo sviluppo di fagi sul ceppo K. All'interno di ciascun gruppo la situazione sembrava paragonabile a quella trovata da Pontecorvo e collaboratori in Aspergillus: una sequenza lineare con numerosi siti mutazionali all'interno dell'unità funzionale, ed un effetto di posizione di tipo cis-trans. Benzer coniò il termine di ‛cistrone' per indicare l'unità funzionale, e quelli di ‛mutone' e di ‛recone' per indicare le più piccole unità contenute nel cistrone ed associate rispettivamente a mutazione e ricombinazione. Era quindi chiaro che il gene, nel mal definito senso dell'unità responsabile di tutte queste attività, era stato suddiviso. In seguito, l'unità funzionale fu ritenuta adatta ad essere considerata quale gene, che pertanto venne identificato con il cistrone.
Questi studi di Pontecorvo, Roper, Pritchard e Benzer stabilirono quindi che il gene, sia in un fungo sia in un virus batterico, quale unità funzionale di materiale ereditario conteneva nel suo interno numerosi siti mutazionali disposti linearmente, tra i quali poteva verificarsi la ricombinazione. Gli studi di Benzer sui cistroni A e B dell'rII dimostrarono che in ciascuno di questi geni ci dovevano essere oltre 100 siti mutazionali: le stime della quantità di DNA presente in ogni cistrone dettero origine all'ipotesi che questi siti potessero corrispondere a coppie di singoli nucleotidi lungo la doppia elica di DNA. I risultati erano infatti in accordo con l'ipotesi del codice genetico, che postulava che il gene consistesse di un particolare tratto di DNA con una sequenza specifica di coppie di nucleotidi e che un cambiamento (mutazione) in un qualsiasi punto della sequenza avrebbe inattivato il tutto.
Ritornando al secondo problema, posto dall'idea che una sequenza di nucleotidi specifichi una sequenza di amminoacidi nei polipeptidi, e più precisamente al problema se la mutazione di un gene possa alterare la sequenza degli amminoacidi, la prima risposta a questa domanda venne dallo studio di una malattia umana chiamata anemia a cellule falciformi. Il nome deriva dalla forma a falce degli eritrociti e la malattia è in genere letale nei primi anni di vita. Viene ereditata attraverso un fattore mendeliano recessivo e l'eterozigote presenta un'accresciuta resistenza alla malaria. V. M. Ingram e J. A. Hunt hanno dimostrato che l'emoglobina nell'anemia falciforme differisce da quella normale per la sostituzione di un singolo amminoacido (la valina al posto dell'acido glutammico) in una particolare posizione di una delle due catene polipeptidiche (la catena β) di ciascuna semi-molecola (v. sangue: Anemie emolitiche; v. cellula: Patologia della cellula). Questo risultato è in pieno accordo con l'ipotesi del codice genetico. In seguito vennero identificati individui eterozigoti per singole sostituzioni di amminoacidi in entrambe le catene α e β della loro emoglobina (v. Baglioni, 1963), e risultò che le due anomalie venivano trasmesse indipendentemente. Il che significava chiaramente che i due polipeptidi erano specificati da geni non associati. Questo è di nuovo in accordo con l'ipotesi che la sequenza dei nucleotidi di un gene specifichi la sequenza degli amminoacidi in un particolare polipeptide.
J. R. S. Fincham e J. A. Pateman scoprirono nel 1957 che due mutanti di Neurospora, apparentemente difettivi per lo stesso gene, dato che erano strettamente associati ed incapaci di sintetizzare un particolare enzima (la glutammico-deidrogenasi), erano tuttavia capaci di effettuare la sintesi se fatti crescere insieme in un eterocarionte. Una scoperta simile fu fatta nello stesso anno da N. H. Giles, C. W. H. Partridge e N. J. Nelson con mutanti che dimostravano un'alterata capacità di sintetizzare l'enzima adenilsuccinasi. Questi gruppi di ricercatori, insieme a D. G. Catcheside e A. Overton che utilizzarono dei mutanti con richiesta di arginina, scoprirono che il tipo di complementazione tra alleli poteva essere rappresentato da una mappa lineare. Questo suggeriva che la complementazione fosse dovuta ad una interazione tra strutture lineari con difetti in regioni diverse. Inoltre, molti mutanti all'interno di questi geni erano incapaci di complementazione reciproca, come se il gene non fosse divisibile in regioni discrete capaci di diverse funzioni. I risultati suggerivano un'interazione tra prodotti lineari del gene, ed i mutanti incapaci di complementare avrebbero avuto dei difetti che non potevano essere rettificati da un prodotto genico difettivo in qualche altra posizione. L'interazione a livello di proteine venne convalidata dalla scoperta che l'attività enzimatica dovuta a due mutanti allelici complementari era anomala ed inferiore al normale: l'ipotesi venne infine confermata quando si riuscì ad ottenere la complementazione in vitro tra proteine isolate da quegli stessi mutanti (v. Fincham, 1966). Sembra quindi che molti enzimi normalmente consistano di più unità di un identico polipeptide e che negli eterocarionti di mutanti allelici i polipeptidi difettivi in posizioni diverse possano a volte interagire formando un prodotto sufficientemente normale, in grado di fornire una qualche attività enzimatica. Un esempio di complementazione apparente tra alleli è stato descritto precedentemente. Una tale interazione implica che una definizione esatta del gene richiede lo studio dell'enzima specifico a esso associato. In un diploide o nell'eterocarionte mutanti in geni diversi consentiranno la produzione di enzimi normali; se però i mutanti saranno nello stesso gene l'enzima risulterà anormale.
M. E. Case e N. H. Giles, per esempio, nel 1960 fecero un paragone tra la mappa di complementazione di una serie di alleli e la mappa delle posizioni dei siti mutanti al- l'interno del gene basata sulle frequenze di ricombinazione. Ne derivò una notevole corrispondenza tra le due mappe, come era da attendersi nel caso che la posizione di un difetto nel gene corrispondesse alla posizione di un difetto nel polipeptide. Ulteriori studi più approfonditi hanno mostrato, tuttavia, che le mappe di complementazione possono anche essere circolari, o presentare delle caratteristiche complesse. È noto come i polipeptidi consistano in una sequenza lineare di amminoacidi (la loro struttura primaria) avvolta in una α-elica, ed inoltre, nel caso di enzimi, ripiegata irregolarmente per dare la cosiddetta struttura terziaria. F. H. C. Crick e A. Orgel nel 1964 supposero che la forma della mappa di complementazione in relazione alla mappa genetica dipendesse da questa struttura terziaria, e O. J. Gillie sostenne due anni dopo che le mappe di complementazione rispecchiavano la forma dell'interfacie tra i polipeptidi che costituiscono l'enzima. Dato che questi studi sulla complementazione allelica sono in rapporto alla struttura terziaria del polipeptide, non è possibile trarne una prova definitiva a favore dell'ipotesi del codice genetico, anche se i risultati sono del tutto compatibili con essa. Ne consegue che per stabilire la validità di questa ipotesi è necessario studiare la sequenza delle mutazioni di un gene in relazione alla struttura primaria, e cioè alla sequenza degli amminoacidi, del corrispondente polipeptide.
Un tale paragone tra gene e polipeptide fu compiuto per la prima volta nel 1964. A. S. Sarabhai, A. O. W. Stretton, S. Brenner e A. Bolle paragonarono la struttura della proteina che costituisce l'involucro della testa del batteriofago T4 di Escherichia coli in una serie di mutanti del gene responsabile di questa proteina. È noto che i polipeptidi sono sintetizzati sequenzialmente dal gruppo amminico a quello terminale carbossilico, e che i successivi amminoacidi sono aggiunti uno ad uno. In una certa classe di mutanti denominati amber (che verranno discussi più avanti), la sintesi del polipeptide si interrompe precocemente, prima di giungere alla sua normale terminazione. La digestione con tripsina della proteina di rivestimento della testa di un virus normale determina la formazione di 8 peptidi, mentre un simile trattamento sulle proteine dei mutanti dà come risultato da 0 a 7 peptidi. Quando i mutanti vennero ordinati in sequenza sulla base dei peptidi ottenuti, si trovò che questa sequenza coincideva con quella delle posisizioni dei siti mutanti entro il gene. Questo portò a stabilire che la posizione di un'anormalità nel DNA corrispondeva alla posizione di un'anormalità nella proteina e quindi confermò l'ipotesi del codice genetico. Questo esperimento fu possibile per l'esistenza di un ceppo di Escherichia coli in cui il carattere amber era soppresso, permettendo così la propagazione dei mutanti.
Contemporaneamente a questo studio sulle proteine della testa del T4, C. Yanofsky e altri paragonarono la struttura di uno dei polipeptidi costituenti l'enzima triptofanosintetasi in Escherichia coli (ceppo normale) con quella di alcuni mutanti per il gene che specificava questo enzima. Si osservò che ciascun mutante differiva dal normale per uno soltanto dei 267 amminoacidi costituenti questo polipeptide, e che la posizione della sostituzione dell'amminoacido nel polipeptide corrispondeva alla posizione del sito di mutazione nel gene. È quindi evidente che la sequenza di nucleotidi nel DNA del gene specifica la sequenza degli amminoacidi nel polipeptide.
d) Il codice genetico
Al fine di chiarire il meccanismo con cui la sequenza di nucleotidi nel DNA viene tradotta in una sequenza di amminoacidi nella proteina, è necessario conoscere la natura molecolare del processo di mutazione. Watson e Crick (v., Genetical implications..., 1953) avevano suggerito che la mutazione potesse essere prodotta dall'inserzione di un nucleotide sbagliato al momento della duplicazione del DNA, come risultato della possibilità che una particolare base si presentasse temporaneamente in una forma tautomerica. Una simile ipotesi trovò convalida quando si scoprì nel 1956 che un analogo di una base, il 5-bromouracile (5BU), causava mutazioni. Questa sostanza è incorporata nel DNA al posto della timina (5-metiluracile) e Benzer e E. Freese scoprirono nel 1953 che il 5BU causava mutazioni in particolari siti dei geni rII del virus T4. Questi autori riportarono anche che vari mutageni davano uno spettro diverso di mutazioni, alcuni inducendo un'elevata frequenza di mutazione in relativamente pochi siti ed altri, quali la proflavina studiata da Brenner, Benzer e Barnett, risultando invece attivi su molti siti. Questa specificità di azione, evidentemente a livello di limitate sequenze di nucleotidi, contrastava con l'assenza di specificità per l'azione mutagena a livello di interi geni (v. sopra). Studi sulla capacità dei mutageni di indurre gli stessi mutanti a tornare normali mostrarono che il 5BU era in grado di operare ciò sui mutanti indotti con 5BU, mentre era inefficace su quelli indotti dalla proflavina. Queste ed altre prove indussero Brenner e collaboratori a proporre che la profiavina causasse mutazione aggiungendo o sottraendo nucleotidi al DNA, anziché mediante la sostituzione di un nucleotide con un altro. Lo studio dei mutanti indotti con la profiavina nei geni rII del virus T4 compiuto da Crick e altri (v., 1961) confermò questa addizione o sottrazione di nucleotidi e rivelò la natura generale del codice genetico.
Crick e collaboratori ottennero un certo numero di retromutazioni di un mutante rII nel gene B, e con studi genetici dimostrarono che erano dovute a mutazioni nella stessa regione del gene B. Questi retromutanti, una volta separati dal mutante originario su cui agivano come soppressori, si comportavano anch'essi come mutanti rII. Crick e collaboratori ritennero che il mutante originario indotto da proflavina fosse dovuto all'aggiunta di un nucleotide e che ciascun soppressore consistesse nella delezione di un nucleotide dalle immediate vicinanze. Se la sequenza dei nucleotidi era letta a partire da un determinato punto iniziale, vi sarebbe stato solamente un corto segmento, a partire dal mutante ‛più' sino al mutante soppressore ‛meno', che sarebbe stato letto fuori fase. In conseguenza, solamente un segmento piuttosto piccolo del corrispondente polipeptide avrebbe contenuto degli amminoacidi sbagliati e pertanto il mutante soppresso sarebbe apparso quasi normale, come appunto venne osservato. Sulla base di questi assunti furono ottenuti dei soppressori di mutanti meno e si ritenne che fossero dei mutanti più. Quando il gene B conteneva tre mutanti più o tre mutanti meno, il fenotipo del virus risultava normale o molto simile al normale. E questo era molto diverso dal fenotipo mutante ottenuto con uno o due mutanti più, o uno o due mutanti meno del gene. Crick e altri dedussero da ciò che tre nucleotidi corrispondevano ad un amminoacido e che, con l'aggiunta o la sottrazione di tre nucleotidi, il gene era letto di nuovo in fase, eccetto che per il breve segmento compreso tra i mutanti. La conferma che l'unità di codice, o codone, per un amminoacido consisteva di tre nucleotidi, e non di un multiplo di tre, richiese una più approfondita conoscenza del meccanismo con cui dal DNA l'informazione passa alla proteina.
Nel processo di traduzione dal DNA alla proteina si ritenne che un secondo tipo di acido nucleico, l'acido ribonucleico, o RNA, agisse da intermediario. Questo perché studi di J. Brachet, T. Caspersson ed altri (v. Brachet, 1957) avevano dimostrato che l'RNA è implicato nella sintesi proteica. In particolare si prospettò la possibilità che la sequenza di nucleotidi del DNA venisse trasferita all'RNA prima della sua traduzione in una sequenza di amminoacidi. Questa possibilità era sembrata plausibile sin dal 1956 quando studi di A. Gierer, G. Schramm e H. Fraenkel-Conrat con il virus del mosaico del tabacco, che contiene RNA e proteine ed è privo di DNA, avevano dimostrato che l'RNA di per se stesso era infettivo, e che la progenie di infezioni miste con l'RNA di un ceppo e la proteina di un altro ereditava solamente i caratteri dell'RNA di origine. Evidentemente l'RNA, come il DNA, può contenere l'informazione per specificare una proteina. Da un punto di vista molecolare l'RNA è abbastanza simile al DNA: la differenza principale è la presenza in esso di zucchero D-ribosio al posto del 2-deossi-D-ribosio. Come nel DNA, anche nell'RNA vi sono quattro tipi di nucleotidi con le stesse basi azotate, fatta eccezione per l'uracile che sostituisce la timina (5-metiluracile) del DNA.
Nel 1964 J. T. August, S. Cooper, L. Shapiro e N. D. Zinder dimostrarono l'esistenza di una RNA-polimerasi RNA-dipendente che può operare la duplicazione dell'RNA di un virus a RNA di Escherichia coli, senza l'intervento del DNA. Nel 1970 D. Baltimore, H. M. Temin e S. Mizutani isolarono una DNA polimerasi RNA-dipendente da alcuni virus a RNA che inducono tumori nei Vertebrati. Questo enzima fa sì che venga sintetizzato del DNA partendo da uno stampo di RNA, e ciò dimostra che l'informazione, sotto forma di sequenza di nucleotidi, può essere trasferita dall'RNA al DNA. S. Spiegelman e collaboratori riscontrarono una stretta correlazione tra la presenza di questo enzima in un virus a RNA e la sua capacità di indurre tumori, in accordo con l'ipotesi che nel cancro una copia in DNA dell'RNA virale possa integrarsi in un cromosoma dell'ospite. L'esistenza di RNA e DNA polimerasi dipendenti dall'RNA come modello conferma che ambedue gli acidi nucleici sono in grado di contenere l'informazione per specificare un polipeptide.
Il termine ribosoma fu coniato nel 1958 da R. B. Roberts per indicare una particella di circa 20 nm di diametro che da alcuni esperimenti di marcatura con amminoacidi radioattivi risultava essere associata alla sintesi proteica. Si trovò inoltre che queste particelle erano composte di RNA e proteine.
Negli studi sulla sintesi proteica che ha luogo in Escherichia coli dopo infezione con i virus T4 e T2 si scoprì che veniva sintetizzato anche dell'RNA a vita media breve. Questo fece pensare ad un ‛RNA messaggero' in grado di trasferire l'informazione dal DNA alle proteine. B. D. Hall e Spiegelman dimostrarono nel 1961 che questo tipo di RNA formava delle molecole ibride con il DNA virale e suggerirono che potesse essere costituito da una sequenza complementare di nucleotidi. Dopo breve tempo, S. B. Weiss e collaboratori e J. Hurwitz e collaboratori isolarono una RNA polimerasi DNA-dipendente dal fegato di ratto. Questi autori congiuntamente a M. Chamberlin e P. Berg trovarono che l'RNA sintetizzato in presenza di questo enzima e di DNA aveva una sequenza di basi complementare a quella del DNA usato come modello. S. Brenner, F. Jacob e M. Meselson, mediante l'uso di precursori radioattivi per marcare l'RNA a vita breve e le proteine di nuova sintesi, confermarono, in seguito a infezione virale in Escherichia coli, che questo RNA e le proteine erano sintetizzati in associazione a vecchi ribosomi: questi potevano essere distinti dai nuovi per mezzo di un marcatore di densità. Tutto ciò dimostrava che i ribosomi erano semplicemente una struttura di supporto per la sintesi proteica, di certo usati ripetutamente da diversi messaggeri. Nel 1963 fu suggerito che l'aggregato lineare di ribosomi osservato al microscopio elettronico durante la sintesi proteica fosse costituito da una successione di ribosomi che si muovevano lungo molecole individuali di RNA messaggero.
Nel 1969 R. Burgess, A. A. Travers, J. J. Dunn e E. K. F. Bautz riuscirono, mediante cromatografia su fosfocellulosa, a separare l'RNA polimerasi DNA-dipendente di Escherichia coli in un nucleo enzimatico e in un fattore sigma. Essi trovarono inoltre che il nucleo enzimatico da solo poteva catalizzare la sintesi dell'RNA. Nel 1970 M. Su- giura, T. Okamoto e M. Takanami scoprirono che l'RNA-polimerasi privata del fattore sigma non era in grado di individuare i siti addetti all'inizio della sintesi dell'RNA, con il risultato che la trascrizione avveniva per ambedue le catene nucleotidiche della molecola di DNA, cioè entrambi i filamenti venivano usati come modello per la sintesi dell'RNA. All'aggiunta del fattore sigma l'inizio della sintesi dell'RNA veniva limitato a siti specifici e per ogni determinata regione un solo filamento del DNA era utilizzato per la trascrizione. J. W. Roberts nel 1969 isolò da Escherichia coli una proteina che denominò fattore rho (per rilascio) in quanto determinava un arresto specifico e successivo rilascio dell'RNA durante la sintesi. Evidentemente delle proteine aggiuntive potevano unirsi al nucleo enzimatico dell'RNA polimerasi conferendogli specificità per l'inizio e la fine della sintesi dell'RNA messaggero (v. Burgess, 1971).
Nel 1957 F. H. C. Crick predisse l'esistenza di una molecola adattatrice per garantire l'unione tra nucleotidi codificanti e lo specifico amminoacido. Gli amminoacidi sono così diversi nella loro struttura che sembrava improbabile che la codificazione potesse avvenire per diretto contatto con l'acido nucleico. Poco più tardi, nello stesso anno, M. B. Hoagland e collaboratori isolarono l'RNA di trasferimento (transfer RNA) dal fegato di ratto. Queste molecole possedevano le proprietà previste da Crick in quanto potevano legarsi agli amminoacidi e trasferirli ai peptidi. Sembrava che vi fosse almeno un tipo di RNA di trasferimento per ciascun amminoacido ed uno specifico enzima per legare ciascun amminoacido al suo appropriato RNA di trasferimento. Ciascuna molecola di RNA di trasferimento era fornita di una particolare sequenza di nucleotidi - l'anticodone - che poteva adattarsi ad una sequenza complementare presente nell'RNA messaggero. Questa interazione ha luogo in associazione con un ribosoma e porta all'inserimento dell'amminoacido appropriato nel polipeptide (v. fig. 7). Ciascun RNA di trasferimento, dopo aver inserito il suo amminoacido nella proteina, rimuove l'RNA di trasferimento che lo precede e fa avanzare il messaggero di un codone. Il ribosoma e il messaggero sono quindi in grado di ricevere il successivo RNA di trasferimento. Il processo attraverso il quale un RNA di trasferimento è scelto in maniera che il suo anticodone possa riconoscere il codone successivo del messaggero è la chiave per una accurata traduzione della sequenza dei nucleotidi in una sequenza di amminoacidi.
La validità dell'ipotesi di Crick sull'adattatore venne confermata nel 1962 quando F. Chapeville e collaboratori trasformarono l'amminoacido cisteina in alanina dopo che la cisteina si era attaccata al suo RNA di trasferimento. Si trovò allora che l'alanina veniva incorporata nel polipeptide al posto della cisteina. Questo dimostrò che non era l'amminoacido di per sé, ma piuttosto l'RNA di trasferimento a cui si era unito, a determinare l'esatta posizione in cui doveva essere inserito nella molecola proteica.
R. W. Holley e collaboratori nel 1965 riuscirono a determinare l'intera sequenza nucleotidica di un RNA di trasferimento per l'alanina nel lievito Saccharomyces cerevisiae. Si trovò che consisteva in una serie lineare di 77 nucleotidi e che l'amminoacido specifico era attaccato a una estremità della catena. Holley e collaboratori, sulla base della sequenza dei nucleotidi, suggerirono che la molecola fosse ripiegata a forma di trifoglio con le basi complementari appaiate tra le porzioni di catena ripiegate su se stesse nei picciuoli di ciascuna fogliolina. Si pensò che l'anticodone fosse situato all'estremità superiore della fogliolina di mezzo. La sequenza nucleotidica è stata da allora determinata per una serie di molecole di RNA di trasferimento estratte dal lievito, da Escherichia coli e da cellule di mammifero: tutte hanno dimostrato di possedere una forma a trifoglio. Nell'RNA di trasferimento si può riscontrare una grande diversità di nucleotidi, tutti derivati apparentemente dai quattro nucleotidi standard in seguito all'azione di enzimi specifici che entrano in attività dopo che è stata messa insieme la sequenza nucleotidica. Gli studi di diffrazione ai raggi X hanno indicato che l'RNA di trasferimento è una molecola compatta e quindi vi deve essere un ulteriore ripiegamento oltre a quello che determina la forma a trifoglio.
Il codice genetico fu decifrato tra gli anni 1961 e 1967: l'impulso più notevole fu dato dalla scoperta di M. W. Nirenberg e J. H. Matthaei (v., 1961), i quali riuscirono ad ottenere in vitro la sintesi di proteine usando tutti i vari componenti estratti da Escherichia coli, più un RNA sintetico. L'osservazione iniziale fu che quando nell'RNA le basi erano tutte costituite da uracile, nel polipeptide sintetizzato veniva incorporato soltanto l'amminoacido fenilalanina. Un ulteriore notevole sviluppo si ebbe con la messa a punto, ad opera di Nirenberg e P. Leder nel 1964, di un metodo per misurare l'interazione dell'RNA di trasferimento con l'RNA messaggero. Questa tecnica venne sfruttata da H. O. Khorana e altri (v., 1967) utilizzando come messaggero un RNA che conteneva sequenze ripetute di nucleotidi: fu così possibile decifrare l'intero codice (v. tab. I). Si confermò che tre nucleotidi adiacenti codificavano per un amminoacido e che nella lettura del messaggio vengono considerate triplette consecutive non sovrapponentisi.
Una prova particolarmente chiara del fatto che il codice rilevato mediante questi studi in vitro è valido anche in vivo è stata fornita dagli esperimenti di O. Streisinger e collaboratori sui mutanti dovuti a scorrimento dello schema di lettura e sui loro retromutanti nel gene per il lisozima del batteriofago T4. I mutanti da scorrimento dello schema di lettura sono quelli, come descritto da Crick e collaboratori per il gene rII, dovuti alla delezione o aggiunta di uno o due nucleotidi (ma non tre). Streisinger e collaboratori accertarono la sequenza degli amminoacidi nella regione del lisozima corrispondente a quella del gene per il lizosima, tra il punto dove era stato aggiunto un nucleotide e quello in cui era stato rimosso. All'interno di questa regione i nucleotidi venivano letti fuori fase e pertanto ne derivava una sequenza anormale di amminoacidi nel polipeptide (v. tab II).
Studi sugli effetti delle mutazioni capaci di alterare la sequenza degli amminoacidi, specialmente nella proteina del virus del mosaico del tabacco ad opera di H. O. Wittmann, e nell'emoglobina umana, hanno confermato che il codice batterico si applica tale e quale ad un largo spettro di organismi e probabilmente a tutti.
Poiché vi sono 64 codoni (sequenze diverse di tre nucleotidi) ma solamente 20 amminoacidi, vi è una certa abbondanza di codoni per molti amminoacidi. Crick, nel 1966, dette un ordine sistematico a tutto questo con la sua ipotesi del ‛tentennamento', secondo la quale l'appaiamento del primo nucleotide dell'anticodone dell'RNA di trasferimento con il terzo codone dell'RNA messaggero non è rigoroso, il che rende possibile numerose alternative.
I lavori di K. A. Marcker e F. Sanger, e di J. M. Adams e M. R. Capecchi tra il 1964 e il 1966 stabilirono inoltre che l'inizio del polipeptide avviene mediante uno speciale RNA di trasferimento a cui è attaccato l'amminoacido metionina. Dopo l'unione dell'amminoacido, il gruppo α-amminico della metionina viene formulato da uno speciale enzima. Come già ricordato il gruppo α-amminico di un amminoacido è quello che si combina con il gruppo carbossilico del precedente amminoacido per la formazione del legame peptidico. La formilazione avrebbe come risultato di impedire la formazione di questo legame. Si trovò poi che un diverso RNA di trasferimento era invece responsabile dell'inserimento della metionina in una posizione intermedia nei polipeptidi e che questa metionina non poteva essere formilata. Queste osservazioni implicavano che tutti i polipeptidi iniziassero con la formil-metionina e quando vennero descritte delle eccezioni si ritenne che la formilmetionina fosse stata rimossa enzimaticamente in un secondo tempo. Negli eucarioti non vi è formilazione del gruppo amminico della metionina iniziale (v. Lucas-Lenard e Lipmann, 1971).
Studi con i mutanti ambra condussero alla scoperta nel 1965, ad opera di M. G. Weigert e A. Garen, e di S. Brenner, A. O. W. Stretton e S. Kaplan, che la tripletta dell'RNA UAG (ambra) codifica la fine del polipeptide; Brenner e collaboratori dimostrarono inoltre che le triplette UAA (ocra) e UGA (opale) segnalano anch'esse il termine della formazione di un polipeptide. Capecchi e M. S. Bretscher fornirono inoltre la prova che non esistono molecole di RNA di trasferimento per la fine del polipeptide e che il completamento di una proteina si attua in qualche altro modo. E. Scolnick, R. Tompkins, T. Caskey e M. W. Nirenberg nel 1968 isolarono due fattori responsabili della separazione del polipeptide dal ribosoma. Uno di questi era in grado di reagire con le triplette ocra ed ambra dell'RNA messaggero, mentre l'altro interagiva con le triplette ocra e opale. Entrambi i fattori risultarono essere delle proteine acide.
Circa 15 anni dopo che l'ipotesi della sequenza - l'idea che la sequenza dei nucleotidi specificasse la sequenza degli amminoacidi - venne proposta per la prima volta, il meccanismo di questa specificazione era chiarito nelle sue linee principali. Un particolare filamento del DNA del gene è trascritto per dare un filamento complementare di RNA: l'RNA messaggero. Questo è tradotto in una sequenza di amminoacidi mediante un complicato processo in cui molecole specifiche di RNA di trasferimento costituiscono il legame essenziale tra l'RNA messaggero e il polipeptide in formazione. Il messaggio di RNA è letto sotto forma di triplette di nucleotidi consecutive non sovrapponentisi a partire da un determinato punto iniziale: ciascuna tripletta a sua volta interagisce con una tripletta complementare di una molecola di RNA di trasferimento a cui è legato un particolare amminoacido. Questa interazione avviene in certe particelle, i ribosomi, che sono anch'esse costituite da numerose molecole di RNA e proteine. Il meccanismo della sintesi proteica ha avuto evidentemente una lunga storia evolutiva. Un gran numero di molecole proteiche di particolare e specifico tipo sono necessarie per la sintesi di ciascuna di tali molecole e questo chiaramente non è avvenuto sin dall'inizio. Similmente, le complesse interazioni tra gli acidi nucleici e le proteine implicate nell'azione dei geni, cioè nella sintesi proteica, pongono svariati e complessi problemi sull'origine e l'evoluzione della vita nel nostro pianeta.
6. Il controllo dell'attività genica e la ricombinazione genetica
a) Il controllo dell'attività genica nei Batteri
M. Demerec e P. E. Hartman scoprirono nel 1956 che nel batterio Salmonella typhimurium i geni responsabili delle reazioni susseguentisi in un particolare processo biochimico erano strettamente associati; B. N. Ames e collaboratori nel 1959 fornirono la prova che questi geni agivano come unità di regolazione. Il rapporto tra l'attività di un enzima e quella di un altro nello stesso processo era costante, anche se i livelli assoluti potevano variare grandemente. Questo fenomeno venne indicato come ‛induzione coordinata' o ‛repressione coordinata', a seconda che una sostanza chimica specifica aumentasse (induttore) o diminuisse (repressore) il livello di attività dell'enzima.
F. Jacob e J. Monod (v., 1961) studiarono un insieme (cluster) di tre geni in Escherichia coli implicati nel metabolismo del lattosio ed avanzarono un'ipotesi particolarmente semplice per spiegare l'attività coordinata degli enzimi specificati da quei geni. I due autori suggerirono che il controllo dell'attività enzimatica venisse esercitato a livello della trascrizione, cioè della sintesi dell'RNA messaggero sul DNA, e che questo controllo fosse possibile attraverso una molecola (repressore) che si legava direttamente al DNA impedendo la sintesi dell'RNA messaggero (v. fig. 8). Il corollario essenziale di questa ipotesi era che l'RNA messaggero avrebbe dovuto avere una vita breve, così che l'arresto della trascrizione avrebbe impedito la sintesi dell'enzima. Jacob e Monod coniarono il termine ‛operatore' per il sito dove la molecola del repressore poteva legarsi al DNA e ‛operone' per il gruppo coordinato di geni che venivano influenzati dal repressore: suggerirono inoltre che tutti questi geni venissero trascritti in una singola molecola di RNA messaggero. Questo avrebbe spiegato l'attività coordinata, in quanto un solo operatore all'inizio della trascrizione sarebbe stato sufficiente per l'intera molecola di RNA messaggero. Gli enzimi per il lattosio in Escherichia coli sono normalmente prodotti soltanto in risposta a un induttore, che è lo stesso lattosio o altri β-galattosidi, e si ritenne che la loro azione consistesse nel legarsi alle molecole del repressore rimuovendole dall'operatore. Jacob e Monod isolarono dei mutanti che sembravano avere delle molecole di repressore difettive - localizzati al sito del gene per il repressore - ed altri mutanti che sembravano avere un operatore difettivo e mappavano al suo sito presunto. Entrambe queste classi di mutanti producevano degli enzimi per la demolizione del lattosio in assenza dell'induttore, come ci si attendeva qualora il difetto - sia nel repressore che nell'operatore - impedisse a questi di unirsi tra loro e di bloccare così la trascrizione.
Nel 1966 W. Gilbert e B. Müller-Hill isolarono e purificarono il repressore dell'operone per il lattosio, trovando che si trattava di una molecola proteica che si poteva legare direttamente al DNA dell'operatore. Questa fu una scoperta di considerevole importanza per svariate ragioni, dato che in primo luogo costituì una diretta conferma dell'ipotesi di Jacob e Monod. Secondariamente la scoperta che una molecola proteica, apparentemente non associata ad alcun acido nucleico, potesse legarsi direttamente ad un particolare segmento del DNA a doppio filamento significava che la proteina poteva riconoscere la sequenza nucleotidica in uno o in entrambi i filamenti a spirale della doppia elica. Un tale riconoscimento poteva essere importante per altre caratteristiche fondamentali del materiale genetico, quali la sua duplicazione o ricombinazione, come pure nell'azione e nella regolazione dei geni. Jacob e Monod avevano così individuato due classi di geni: i geni strutturali capaci di specificare proteine funzionanti come enzimi o componenti cellulari, e i geni regolatori la cui funzione era quella di regolare l'attività dei geni strutturali. Questi ultimi non furono considerati necessariamente diversi a livello del gene, dato che entrambi i tipi di geni specificavano la sequenza degli amminoacidi dei polipeptidi.
Nel 1967 M. Ptashne isolò un repressore che controllava l'attività del virus di Escherichia coli. Anche questa molecola di repressore risultò essere una proteina in grado di legarsi a regioni specifiche del DNA a doppio filamento del virus.
Oltre al controllo negativo dell'attività genica per mezzo della repressione, si conosce anche un controllo positivo attraverso l'attivazione genica. Nel 1965 E. Englesberg, J. Irr, J. Power e N. Lee descrissero un tale sistema per i geni implicati nel metabolismo dello zucchero arabinosio in Escherichia coli. La molecola del repressore per l'operone dell'arabinosio è separata dall'operatore per l'intervento dello zucchero, e si ritiene che il complesso del repressore con l'arabinosio promuova attivamente la sintesi degli enzimi per la scissione dell'arabinosio. Tuttavia, diversamente dagli enzimi per la demolizione del lattosio, questa sintesi non si verifica per la sola assenza del repressore. Nel 1971 G. Zubay, L. Gielow ed Englesberg, e J. Greenblatt e R. Schleif confermarono con studi in vitro che per rendere l'operone dell'arabinosio attivo è necessaria una proteina che agisca da regolatore positivo.
L'operone per il lattosio in Escherichia coli ha fornito inoltre informazioni su un secondo sito regolatore denominato ‛promotore'. Questo è il luogo di inizio per la sintesi dell'RNA messaggero e si ritiene che non coincida con l'operatore, poiché i mutanti, di cui si è già parlato, con un operatore difettivo mostrano di possedere una piena attività dell'operone. J. R. Beckwith e collaboratori dimostrarono che i mutanti al sito promotore, riconosciuti per la ridotta attività di tutti i geni lattosio, mappavano dal lato opposto dell'operatore rispetto a questi geni. Essi suggerirono che l'RNA polimerasi si legherebbe al promotore e che il passaggio dell'enzima verso i geni lattosio sarebbe bloccato qualora il repressore fosse unito all'interposto operatore.
Sebbene i siti promotore e operatore siano stati individuati per la prima volta nell'operone per il lattosio, sembra probabile che tali siti si trovino anche all'inizio di geni strutturali singoli, non organizzati in un operone. Anche se N. H. Giles nel 1967 dimostrò nel fungo Neurospora crassa l'esistenza di un operone con mutanti a richiesta di amminoacidi aromatici, i geni strutturali per processi biosintetici negli eucarioti, contrariamente a molti batteri, non risultano raggruppati in operoni, ma si presentano o indipendenti o debolmente associati. Quanto poi il controllo dell'attività genica negli eucarioti sia paragonabile a quello riscontrato per l'operone lattosio in Escherichia coli, rimane materia per congetture.
b) Il meccanismo della ricombinazione genetica
Il verificarsi di scambi di segmenti tra cromosomi omologhi, uno di origine paterna, l'altro di origine materna, è già stato discusso precedentemente. In questi ultimi anni sono stati fatti alcuni progressi verso la comprensione del meccanismo di questo processo di ricombinazione.
Una possibilità è che la duplicazione del cromosoma avvenga copiando un cromosoma parentale per un tratto della sua lunghezza e copiando il rimanente tratto dal corrispondente cromosoma dell'altro genitore. La conseguenza di questa ipotesi della scelta della copia (copy-choice) sarebbe che tutti i ricombinanti implicherebbero cromosomi di nuova sintesi e mai quelli parentali. Gli studi, riportati in precedenza, che stabilirono come il crossing-over si verificasse tra cromatidi piuttosto che tra cromosomi parentali, mostrarono anche, tuttavia, che da diversi cromatidi di un cromosoma potevano originare ricombinanti multipli: ciascun cromatidio paterno poteva effettuare scambi con qualsiasi cromatidio materno. E questa osservazione è in aperto contrasto con l'ipotesi del copy-choice.
Un'altra osservazione contrasta questa ipotesi. Come già descritto, Taylor e altri (v., 1957) dimostrarono che il processo di duplicazione del cromosoma è semiconservativo, cioè a dire consiste in due subunità longitudinali che si separano in ciascuno dei due cromatidi che ne derivano mentre accanto ad ognuno di questi si affianca una nuova subunità. In tal modo ciascun cromatidio consiste di una vecchia subunità e di una nuova. È pertanto sbagliato considerare un cromatidio come parentale e l'altro come quello di nuova sintesi, come suggerito dall'ipotesi del copy-choice.
L'alternativa al copy-choice è il processo di rottura e fusione. Teoricamente le due alternative possono essere saggiate con esperimenti di marcatura radioattiva, dato che l'ipotesi rottura e fusione, contrariamente al copy-choice, prevede che atomi di cromosomi parentali vadano a costituire i cromosomi ricombinanti. In pratica, questi esperimenti di marcatura si sono dimostrati di difficile esecuzione. Per la prima volta furono realizzati nei procarioti da M. Meselson e J. J. Weigle nel 1961, utilizzando il virus λ di Escherichia coli e negli eucarioti da Taylor nel 1965, utilizzando un ortottero. In entrambi i casi i risultati indicarono nel processo di rottura e fusione il meccanismo della ricombinazione. La comprensione dei dettagli di questo processo di rottura e fusione non è stata tuttavia ancora possibile e vi sono numerose ragioni per spiegare questa lacuna. Studi a livello molecolare, da cui si può attendere la chiave del meccanismo di ricombinazione, possono essere compiuti utilizzando i batteri e i loro virus, ma l'interpretazione dei risultati è complicata da diversi fattori. In primo luogo la ricombinazione nei procarioti non avviene fra interi cromosomi derivati da due genitori durante la meiosi come nel caso degli eucarioti. Nei batteri si conoscono diversi meccanismi per il trasferimento del materiale ereditario da una cellula all'altra. Tra questi il primo ad essere stato scoperto è la trasformazione, di cui abbiamo già trattato. Nel 1952, J. Lederberg e altri dimostrarono che nel batterio Salmonella typhimurium del materiale ereditario di un ceppo poteva essere trasferito in cellule di un altro ceppo ad opera di un virus. Per indicare questo processo fu coniato il termine di ‛trasduzione'. Nel 1958 gli studi di F. Jacob, E. L. Wollman e W. Hayes stabilirono inoltre che del DNA poteva essere trasferito da una cellula di Escherichia coli a un'altra in seguito a un processo di coniugazione. In tutti questi processi di trasferimento, solamente un frammento del DNA del ceppo donatore viene passato al ricevente, e questo complica l'analisi degli studi a livello molecolare.
Una seconda difficoltà per l'interpretazione dei dati sulla ricombinazione nei procarioti è che raramente si possono ricuperare entrambi i componenti - il donatore ed il ricevente - di eventi individuali, poiché la ricombinazione non è ristretta a una fase particolare del ciclo vitale, ma può verificarsi ripetutamente. E questo è particolarmente vero per la ricombinazione nei virus.
In terzo luogo, sembra che i procarioti abbiano più di un meccanismo di ricombinazione, entro una singola specie. Questo può essere esemplificato dal virus λ di Escherichia coli. Studi compiuti con mutanti ad alterata ricombinazione, specialmente ad opera di A. J. Clark, H. Echols, E. R. Signer e altri, hanno dimostrato l'esistenza di tre sistemi chiamati rec, red e int. Il sistema rec è determinato dai geni dell'ospite, ma i loro prodotti possono influenzare la ricombinazione nel virus λ. Il sistema red invece interessa soltanto il virus. Il sistema int è un sistema piuttosto specializzato che integra il cromosoma virale entro quello dell'ospite mediante una rottura e una riunione di entrambi in un punto determinato. Una tale integrazione mediante un processo di ricombinazione fu proposta da A. M. Campbell nel 1962 e da allora è stata confermata da numerosi esperimenti. Il cromosoma del virus assume una forma circolare, prima dell'integrazione, in seguito alla saldatura delle due estremità della molecola del DNA.
Da un punto di vista genetico, invece, gli organismi più adatti per studiare la ricombinazione sono i funghi Ascomiceti. Oltre a conservare i prodotti della meiosi entro una singola cellula (l'asco) ed a fornire la possibilità di selezionare i ricombinanti entro un vasto numero di genotipi parentali, alcuni Ascomiceti presentano anche dei mutanti per il colore delle ascospore. Queste caratteristiche sono state utilizzate per la prima volta da H. Zickler nel 1934 con il fungo Bombardia lunata. Si scoprì allora che nell'incrocio tra un ceppo normale e uno mutante, alcune volte si verificava un processo di conversione che portava alla formazione di aschi con 6 spore di un colore e 2 dell'altro, invece di 4 e 4 (v. fig. 9, A e B). La conversione fu riscoperta da Lindegren nel 1953 in Saccharomyces cerevisiae (lievito): nel 1955 M. B. Mitchell dimostrò con Neurospora crassa che essa era causata da una segregazione anomala del sito mutante e non dell'intero cromosoma. Nel 1959, L. S. Olive scoprì in Sordaria fimicola che da un incrocio tra un ceppo normale e uno mutante per il colore delle spore, si potevano ottenere anche degli aschi con 5 spore di un colore e 3 dell'altro (v. fig. 9C), mentre nel 1962 Y. Kitani e altri trovarono un quinto rapporto anomalo, che denominarono ‛4:4 aberrante', in quanto la segregazione del colore delle spore era regolare ma non corrispondeva alla segregazione di alcuni mutanti associati sullo stesso cromosoma.
C. Levinthal nel 1955 suggerì, insieme ad altre alternative, che nella ricombinazione virale si verificasse l'associazione delle catene nucleotidiche complementari derivanti dai due genitori; nel 1962-1964 R. Holliday e H. L. K. Whitehouse mostrarono come questo principio della formazione di un eteroduplex o DNA ibrido potesse anche spiegare molti aspetti della ricombinazione degli eucarioti, se si assumeva che un sistema di enzimi potesse riconoscere e rimuovere delle basi erroneamente appaiate derivanti da una tale riassociazione delle catene parentali (v. fig. 10). Si pensò inoltre che la correzione dei tratti mal appaiati nel DNA ibrido consistesse nella rimozione di un segmento di catena nucleotidica comprendente alcune centinaia di nucleotidi. Questo spiegherebbe la conversione simultanea di due o più mutanti strettamente associati allo stesso genotipo parentale, come osservato da S. Fogel, D. D. Hurst e R. K. Mortimer nell'analisi degli aschi non selezionati in Saccharomyces cerevisiae. Una rimozione piuttosto estesa darebbe conto anche del fenomeno dell'allungamento della mappa di ricombinazione, per cui le frequenze di ricombinazione intragenica sono più grandi della somma di quelle osservate tra le singole coppie di mutanti considerati.
R. H. Pritchard nel 1955 e M. B. Mitchell nel 1956 incrociarono dei mutanti allelici auxotrofici rispettivamente di Aspergillus nidulans e Neurospora crassa e osservarono che la progenie di tipo normale formatasi per ricombinazione intragenica poteva avere ognuno dei 4 possibili genotipi per quanto riguardava i marcatori esterni delimitanti il gene in questione. Questa scoperta significava che si verificava una ricombinazione aggiuntiva oltre a quella che veniva recuperata mediante la selezione. La correzione delle basi mal appaiate nel DNA ibrido ci dà una possibile spiegazione per queste ulteriori rotture e fusioni.
Un processo abbastanza simile alla conversione delle basi non corrispondenti nel DNA ibrido è quello della riparazione del danno causato nel DNA dalla luce ultravioletta. P. Howard-Flanders e collaboratori dimostrarono nel 1966 che in Escherichia coli un sistema di enzimi determinato dai geni denominati uvrA e uvrB è responsabile della rimozione dei dimeri della pirimidina indotti dagli UV e della loro sostituzione con nucleotidi complementari a quelli nell'altra catena nucleotidica (v. fig. 11). Vengono così rimosse alcune centinaia di nucleotidi e non solamente i dimeri incriminati. W. D. Rupp e Howard-Flanders nel 1968 trovarono che quando un DNA contenente dimeri di pirimidina si duplica, nel filamento di nuova sintesi si viene a formare una discontinuità proprio di fronte al dimero. Questo precluderebbe la riparazione ad opera del sistema uvr, che richiede una catena complementare intatta come modello. Rupp, C. E. Wilde, D. L. Reno e Howard-Flanders dimostrarono nel 1971 per mezzo di esperimenti con marcatori di densità che un sistema enzimatico specificato dal gene recA permette la riparazione da ricombinazione anche quando il sistema uvr è inattivo: un tratto di catena nucleotidica appartenente alla molecola sorella viene inserito per riempire la discontinuità che si trova di fronte al dimero di pirimidina (v. fig. 11). Si ritiene che la riparazione del danno da UV mediante il processo di ricombinazione possa verificarsi anche negli eucarioti. Una delle caratteristiche peculiari della ricombinazione negli eucarioti è che il processo mostra una polarità - un gradiente regolare nella frequenza di conversione dipendente dalla posizione del sito mutante entro il gene. Questo fu osservato per la prima volta da P. Lissouba e G. Rizet nel 1960 utilizzando mutanti per il colore delle spore in Ascobolus immersus e da N. F. Murray con mutanti a richiesta di metionina in Neurospora. La polarità può essere interpretata ammettendo che vi siano dei punti di apertura fissati, al di fuori dei geni, dai quali inizi la formazione del DNA ibrido. Catcheside e collaboratori, sin dal 1965, scoprirono in Neurospora diversi repressori dominanti nella ricombinazione. Ciascun repressore altera la ricombinazione in una o più regioni specifiche del cromosoma, apparentemente prevenendo l'apertura di particolari punti di inizio per la ricombinazione.
È possibile che i punti fissi di apertura da cui si ritiene che inizi la ricombinazione negli eucarioti siano proprio le estremità dei repliconi. Se poi la ricombinazione si originassenon in seguito a rottura, ma per il mancato congiungimento della catena nucleotidica proprio ai punti di congiunzione tra un replicone e l'altro dopo la replicazione del cromosoma, questo potrebbe spiegare perché il crossing-over si verifichi sempre dopo la duplicazione (v. Whitehouse, 1971).
Nonostante le numerose recenti scoperte e i modelli del DNA ibrido proposti per interpretarle, molti dettagli del processo di ricombinazione negli eucarioti, quali l'eccesso di ricombinanti non reciproci all'interno dei geni, sono ancora del tutto oscuri.
Una struttura notevole, denominata ‛complesso sinaptinemale', si è certamente evoluta in relazione all'appaiamento cromosomico e alla ricombinazione negli eucarioti. Questo complesso fu scoperto nel 1956 da M. J. Moses e sembra che accompagni invariabilmente lo stadio di pachitene alla meiosi, a meno che il crossing-over sia assente (v. Moses, 1968). Il complesso sinaptinemale sembra sia composto da proteine ed appare al microscopio elettronico come due elementi paralleli distanti 120-150 nm che si trovano sullo stesso piano tra i cromosomi appaiati e che contengono un elemento centrale più sottile interposto. Filamenti sottilissimi trasversali riempiono lo spazio tra i due elementi paralleli. Sono state fatte molte ipotesi sulla relazione tra il complesso sinaptinemale e la ricombinazione, ma è difficile che si facciano dei passi avanti per capire questa relazione, a meno che non si trovino dei mutanti caratterizzati da un complesso difettivo.
c) Struttura e organizzazione del cromosoma
I modelli a DNA ibrido per spiegare il meccanismo della ricombinazione assumono che l'asse di un cromosoma consista di una singola molecola di DNA, cioè di due catene complementari di nucleotidi, ma vi sono state notevoli discussioni circa l'esattezza di questo assunto. Prove a favore dell'ipotesi dell'unica molecola di DNA sono state offerte da Taylor che, con esperimenti di marcatura al trizio del tipo già descritto, dimostrò come potessero verificarsi scambi di parti tra cromatidi fratelli. Questi scambi si rendevano evidenti dopo la seconda duplicazione, quella cioè verificatasi in assenza di 3H. I grani di argento nell'autoradiografia si trovavano in corrispondenza di un cromatidio per un tratto della sua lunghezza e sul cromatidio fratello per il tratto rimanente. Alcune volte inoltre i cromatidi fratelli presentavano uno scambio in posizione corrispondente. Ci si attende che tali scambi gemelli siano relativamente più frequenti di quelli singoli qualora le subunità longitudinali del cromatidio siano strutturalmente di due tipi, in maniera da imporre una restrizione alla loro riunione in nuovi modi dopo la rottura (v. fig. 5 g, i). Conteggi nella frequenza relativa di scambi gemelli e singoli, eseguiti dapprima da Taylor nel 1958 nella liliacea Bellevalia romana e più estesamente da B. Herreros e F. Giannelli nel 1967 in colture di cellule di mammifero, hanno confermato che le subunità sono strutturalmente diverse. Questo è in accordo con la diversa polarità delle due catene nucleotidiche del DNA (i cui assi sono rivolti verso direzioni opposte), ma non esclude la possibilità che le subunità debbano la loro diversità strutturale a componenti diverse.
La somiglianza, tuttavia, tra le subunità di Taylor e le catene individuali di nucleotidi del DNA si è rivelata ancora più evidente: J. O. Brewen e W. J. Peacock nel 1969 hanno dimostrato che le differenze strutturali tra le due subunità di un cromatidio possedevano una loro polarità, in modo che una subunità era l'opposto dell'altra. Questo fu provato applicando la tecnica della marcatura con timidina-3H di Taylor ai riordinamenti strutturali indotti da raggi X in cellule di Cricetulus (hamster cinese) coltivate in vitro. Questi riordinamenti strutturali comprendevano rotture di ambedue i cromatidi, seguite da riunioni in maniera da formare un cromosoma doppio con due centromeri.
La marcatura dei cromosomi con la tecnica di Taylor ha, in alcuni casi, prodotto dei cromosomi con entrambi i cromatidi marcati (isomarcatura) in posizioni corrispondenti dopo la duplicazione in assenza di trizio. Queste osservazioni sono difficili da spiegarsi sulla base del modello ‛una molecola di DNA', a meno che non si ammetta, come suggerito da D. E. Comings nel 1971, che l'asse del DNA sia ripiegato longitudinalmente all'interno del cromosoma metafasico condensato in corrispondenza delle zone di isomarcatura. La marcatura dei due cromatidi in quelle che sembrano posizioni corrispondenti non significherebbe quindi marcatura di regioni corrispondenti lungo l'asse del DNA, e potrebbe spiegarsi con uno scambio tra cromatidi (v. fig. 5, h).
Gli esperimenti di marcatura con il trizio e i dati sulla ricombinazione forniscono un notevole appoggio all'ipotesi che l'asse del cromosoma consista di una sola molecola a doppia elica di DNA. Sembra infatti che non esistano in proposito prove contraddittorie, sebbene la variazione nella quantità di DNA nucleare osservata in cellule di specie affini sia stata spesso invocata come evidenza che i cromosomi di specie diverse differiscono nel numero di molecole di DNA che formano il loro asse. La diversità nella quantità del DNA fu scoperta da A. E. Mirsky e H. Ris nel 1951 in seguito a misurazioni effettuate sugli eritrociti dove misero in evidenza notevoli differenze. Essi trovarono infatti che nei Vertebrati le quantità variavano da 1 a oltre 150 pg per cellula diploide e, dieci anni dopo, J. McLeish e N. Sunderland trovarono simili variazioni tra le piante superiori.
H. G. Keyl (v., A demostrable..., 1965) misurò la quantità di DNA presente nelle bande trasversali dei cromosomi delle ghiandole salivari in due subspecie del dittero Chironomus thummi utilizzando un microspettrofotometro dopo una colorazione di Feulgen, specifica per il DNA. Trovò così che ciascuna banda di una sottospecie mostrava sempre del DNA in quantità 1, 2, 4, 8 o 16 volte maggiore della quantità presente nelle bande corrispondenti dell'altra sottospecie (v. fig. 12). Questo rapporto evidentemente era valido anche per i cromomeri - gli elementi corrispondenti alle bande trasversali - nei cromosomi normali, dato che una sottospecie possedeva un 27% di DNA in più dell'altra, sia che lo si misurasse nelle cellule delle ghiandole salivari che negli spermatociti. Keyl ne trasse la conclusione che la differenza nella quantità di DNA tra le due sottospecie si era originata attraverso il successivo raddoppiamento del DNA dei singoli cromomeri. Si preferì l'ipotesi del raddoppiamento a quella del dimezzamento, dato che la sottospecie con la quantità inferiore di DNA era più vicina al tipo ancestrale sulla base della sequenza delle bande trasversali nei cromosomi salivari. Questa sequenza va incontro a lenti cambiamenti dovuti all'insorgere di riordinamenti strutturali nei cromosomi, quali le inversioni di alcuni tratti.
La scoperta di Keyl della duplicazione durante l'evoluzione della quantità di DNA nei cromomeri offre un'alternativa alla variazione del numero di molecole di DNA nell'asse cromosomico per spiegare i cambiamenti nella quantità di DNA presente in specie affini. I suoi dati pongono inoltre il problema del significato di questo raddoppiamento. Numerosi indizi suggeriscono attualmente l'ipotesi che il cromomero sia l'unità funzionale per una serie di diverse attività del materiale genetico quali la duplicazione del cromosoma, la ricombinazione e l'attività del gene.
La prova che negli eucarioti il replicone corrisponde al cromomero fu ottenuta da Keyl e C. Pelling nel 1963. Con la marcatura dei cromosomi delle ghiandole salivari di Chironomus per mezzo di 3H e di 14C, essi dimostrarono che tutte le bande iniziano la loro duplicazione entro 20 minuti l'una dall'altra, ma che finiscono di duplicarsi impiegando tempi diversi, con tempi più lunghi per quantità maggiori di DNA. La scoperta di Keyl del raddoppia- mento durante l'evoluzione della quantità di DNA dei cr0momeri, in maniera indipendente l'uno dall'altro, avvalora l'idea che essi siano delle unità discrete del cromosoma, dal punto di vista della replicazione (v. Pelling, 1966).
Se il replicone corrisponde al cromomero, allora il cromomero può anche essere considerato l'unità funzionale del processo di ricombinazione, dato che, come abbiamo già detto, vi è la possibilità che la ricombinazione si inizi alle estremità del replicone.
Nei cromosomi degli oociti degli Anfibi quale Triturus (salamandra), da numerosi cromomeri si dipartono delle anse (v. fig. 13). Questo fa si che i cromosomi assumano un caratteristico aspetto sfrangiato che è all'origine del loro nome di cromosomi a spazzola: oggi si ritiene che tutti i cromosomi profasici abbiano una organizzazione di questo tipo (v. genetica: Citogenetica). I cromosomi a spazzola sono stati studiati a fondo da J. G. Gall e H. G. Callan (v. Callan, 1963). Trattamenti enzimatici hanno rivelato che le anse hanno un filamento centrale di DNA circondato da una matrice di RNA e proteine. Lo stiramento meccanico dei cromosomi a spazzola può provocare una rottura trasversale dei cromomeri e sarà allora l'ansa a tenere insieme i punti di rottura: questo ha dato luogo all'ipotesi che l'asse dell'ansa sia la continuazione dell'asse intercromomerico del cromosoma.
Callan ha suggerito nel 1967 che ciascun cromomero consista di una serie in tandem di copie duplicate del gene, e che ciascuna copia detta ‛schiava' (slave) abbia la stessa sequenza di nucleotidi della copia ‛padrona' (master) localizzata ad una estremità della serie: questo sarebbe il risultato di un processo di appaiamento consistente nella temporanea ricostituzione di catene nucleotidiche complementari, una di origine slave e l'altra master. A ciò seguirebbe l'eliminazione di tutti i nucleotidi slave che non siano complementari a quelli master e la loro sostituzione con nucleotidi sintetizzati utilizzando la catena master come modello. Le anse dei cromosomi a spazzola mostrano di possedere una struttura polarizzata con una quantità di matrice maggiore ad una estremità che all'altra ed esperimenti di marcatura hanno confermato che l'RNA della matrice si sposta lungo l'ansa partendo dall'estremità più sottile verso quella più spessa. Callan ha suggerito inoltre che anche l'asse di DNA dell'ansa sia in movimento e questo sarebbe una conseguenza del fatto che le copie slave emergono in successione dal processo di appaiamento. E aggiunse che la sintesi dell'RNA messaggero avverrebbe nell'ansa dopo il processo di appaiamento e che la copia master non verrebbe trascritta in RNA. Secondo l'ipotesi di Callan del master and slaves, il raddoppiamento durante l'evoluzione della quantità di DNA in un cromomero sarebbe dovuto al raddoppiamento delle copie slaves. E questo potrebbe rappresentare un vantaggio selettivo se permettesse di aumentare la velocità dell'azione genica.
Un'altra ipotesi per spiegare l'organizzazione ad ansa dei cromomeri nei cromosomi a spazzola fu proposta da W. Beermann nel 1965. Egli suggerì che l'RNA venisse sintetizzato alla base dell'ansa, dove si pensava fosse localizzato il gene, e che quindi questo RNA si spostasse lungo l'ansa dove veniva rivestito da proteine per il suo trasporto dal nucleo nel citosplama. Fu portato a formulare questa ipotesi sulla base degli studi che, insieme ai suoi collaboratori, aveva eseguito sui puffs (rigonfiamenti) dei cromosomi politenici (v. fig. 14). I puffs, nei cromosomi delle ghiandole salivari, sembrano essere la struttura corrispondente a quella delle anse dei cromosomi a spazzola degli oociti degli Anfibi (v. genetica: Citogenetica). Nel 1961 U. Clever trovò che in Chironomus, in seguito all'iniezione di ecdisone, una sostanza che induce il passaggio da larva a pupa (v. ormoni negli invertebrati), la formazione dei puffs avveniva con sequenze caratteristiche in particolari posizioni del cromosoma. J. E. Edström e Beermann nel 1962 dimostrarono che un RNA a composizione specifica è associato a ciascun puff come se questo corrispondesse all'attività di un particolare gene.
L'ipotesi di Beerman sull'impacchettamento, come pure l'idea di Callan sul master and slave sono un tentativo di spiegare l'esistenza di un filamento di DNA in un'ansa o puff più grande per diversi ordini di grandezza di un gene, senza abbandonare l'ipotesi ‛un gene-un cromomero' (v. Hess e Meyer, 1968). Le dimensioni approssimative di un gene sono note, dato che 3 nucleotidi, che occupano circa 1 μm di DNA misurato lungo l'asse dell'elica, sono necessari per specificare ciascun amminoacido. In altre parole in un μm di DNA potrebbero stare tre geni, ognuno dei quali in grado di specificare una catena polipeptidica contenente più di 300 amminoacidi. Le anse di un cromosoma a spazzola di Triturus possono avere una circonferenza di oltre 30 μm, una lunghezza sufficiente per circa un centinaio di tali geni.
La prova delle sequenze ripetute di nucleotidi, come era stato postulato da Callan, fu fornita da C. A. Thomas e collaboratori nel 1970. Questi usarono una tecnica di adesio-frizione per ottenere la frammentazione del DNA di eucarioti, un'esonucleasi per indurre la formazione di catene singole di nucleotidi alle estremità dei filamenti, ed un lento raffreddamento per permettere la ricostituzione di catene complementari; quindi esaminarono il DNA al microscopio elettronico. Si poterono allora osservare numerosi cerchi chiusi su se stessi, prova che le estremità opposte di un frammento avevano delle sequenze nucleotidiche complementari.
I lavori, a partire dal 1965, di F. M. Ritossa e Spiegelman in Drosophila e di J. B. Gurdon e altri in Xenopus hanno dimostrato che l'organizzazione nucleolare, che codifica l'RNA ribosomale, consiste di sequenze di nucleotidi ripetute diverse centinaia di volte. O. L. Miller e B. R. Beatty (v., 1969) hanno potuto fotografare al microscopio elettronico questi geni nucleolari in azione negli oociti di Triturus. Numerosi segmenti, lunghi ciascuno circa 2,5 μm, erano provvisti di una matrice di RNA e proteine attorno all'asse del DNA e si pensò che ciascuno di essi corrispondesse ad un gene nucleolare (v. fig. 15). Questi geni erano disposti in una sequenza ripetuta lungo il DNA ed erano separati uno dall'altro da tratti di DNA privi di matrice. Queste regioni inerti variavano da 1 a 25 μm di lunghezza. La matrice di RNA e proteine disposta attorno a ciascun gene nucleolare era più spessa ad una estremità che all'altra e l'estremità più sottile era quella da cui si pensava iniziasse la trascrizione. Dal numero di fibrille costituenti la matrice si stabili che erano attive circa 100 molecole di RNA polimerasi, distribuite uniformemente lungo ogni gene.
Un altro esempio di sequenze ripetute di nucleotidi è costituito dal DNA satellite riscontrato nei Mammiferi e studiato da R. J. Britten, P. M. B. Walker, M. L. Birnstiel ed altri. Questo consiste di una corta sequenza di nucleotidi ripetuta molte migliaia di volte ed è localizzato nelle regioni eterocromatiche dei cromosomi, attorno ai centromeri. La dimostrazione della sua localizzazione è stata data da K. W. Jones e da M. L. Pardue e J. G. Gall nel 1971 marcando il DNA satellite e preparando poi delle autoradiografie dopo aver fatto ricombinare il DNA satellite con dei cromosomi intatti. La funzione di questo DNA satellite non è ancora conosciuta.
Possiamo pertanto concludere operativamente, sulla base delle prove in nostro possesso, che l'asse di un cromosoma consiste di una molecola di DNA (due catene di nucleotidi) e che l'unità di replicazione, ricombinazione e attività genica nel cromosoma eucariotico è il cromomero. Questo può consistere di una copia master di un gene e di una serie di copie slaves disposte in sequenze a tandem, con un processo di riconoscimento per dare alle slaves le sequenze nucleotidiche del master, prima che l'RNA messaggero venga sintetizzato ad opera delle slaves. Si pensa che un processo di riconoscimento debba anche avvenire durante la ricombinazione, in seguito all'associazione di catene nucleotidiche complementari - o quasi complementari - da regioni corrispondenti di cromosomi paterni e materni.
d) Il controllo dell'attività genica negli organismi superiori
Uno degli aspetti della genetica meno conosciuti è il controllo dell'attività genica negli eucarioti. Spesso si è ritenuto che il controllo sia organizzato nella stessa maniera che nei procarioti (modello dell'operone per il lattosio in Escherichia coli), ma non vi è alcuna chiara corrispondenza derivata dagli studi sugli eucarioti (v. Harris, 19702).
Numerosi dati sperimentali consentono di ritenere che l'eterocromatina rivesta un suo ruolo nel controllo dell'attività genica degli eucarioti, ma nulla si conosce ancora di preciso, a livello molecolare, sulle funzioni dell'eterocromatina. Questa fu descritta da E. Heitz per primo nel 1928 come facente parte di regioni cromosomiche che si coloravano più intensamente nell'epatica Pellia. Adesso si è visto che le regioni eterocromatiche spesso si colorano più intensamente in una parte del ciclo mitotico e meno intensamente in un'altra, quando vengano confrontate con le regioni eucromatiche. L'idea che l'eterocromatina sia fuori fase rispetto all'eucromatina durante il ciclo mitotico è convalidata dalla scoperta, fatta da A. Lima-de-Faria nel 1959, che l'eterocromatina si replica più tardi dell'eucromatina. Al microscopio elettronico nessuna differenza fu messa in rilievo da H. Ris nel 1957 tra eterocromatina ed eucromatina, tranne che nel grado di compattezza dei filamenti nucleoproteici: anche dal punto di vista chimico non si è riusciti ad accertare una qualche diversità per i suoi componenti. D'altra parte alcune regioni eterocromatiche contengono con certezza del DNA satellite e Caspersson e collaboratori, nel 1968, trovarono che queste regioni davano una colorazione fluorescente se trattate con chinacrina, ciò che ha reso possibile il riconoscimento individuale di tutti i cromosomi umani per il loro caratteristico tipo di colorazione con la chinacrina. Il sicuro riconoscimento di ciascun cromosoma ha una notevole importanza da un punto di vista medico perché molte anomalie dello sviluppo nell'uomo sono correlate ad anomalie cromosomiche. L'esempio classico a questo proposito viene dalla scoperta fatta da J. Lejeune, M. Gautier e R. Turpin nel 1959 che il mongolismo è associato alla presenza nel corredo cromosomico delle cellule di un cromosoma n. 21 in più. L'anomalia dei trisomici, cioè di individui con un cromosoma rappresentato tre volte anziché due nei loro nuclei, è stata scoperta da A. F. Blakeslee sin dal 1922. Questi aveva dimostrato che in Datura stramonium le piante con uno dei cromosomi in triplice copia avevano un aspetto caratteristico che dipendeva da quale cromosoma si trovava in eccesso. (V. eugenica; v. genetica: Applicazioni della genetica e Citogenetica).
Le regioni eterocromatiche dei cromosomi delle ghiandole salivari non contengono bande trasversali. T. Caspersson e J. Schultz nel 1938 scoprirono che in Drosophila l'eucromatina, se trasportata in seguito a una alterazione strutturale nelle vicinanze dell'eterocromatina, perde le sue bande trasversali in alcune delle cellule. Schultz nel 1948 trovò che questo aspetto diffusivo dell'eterocromatina nell'eucromatina poteva spiegare il fenotipo variegato osservato spesso nei casi in cui un gene veniva trasferito, a causa di un riordinamento strutturale, nelle vicinanze di regioni eterocromatiche (v. Baker, 1968). Sembra inoltre che l'eterocromatina non contenga dei normali geni strutturali, dato che si conoscono pochi mutanti di tali geni, o non se ne conosce alcuno, localizzati nelle regioni eterocromatiche. Oli studi di Schultz inoltre suggeriscono che quando una regione eucromatica diviene eterocromatica i geni in essa contenuti divengono inattivi.
M. F. Lyon suggerì nel 1961 che nelle femmine dei Mammiferi uno dei due cromosomi X in ogni cellula possa diventare inattivo qualche giorno dopo la fecondazione e rimanere inattivo durante tutto lo sviluppo. Sin dal 1949 era stato scoperto da M. L. Barr e E. G. Bertram che nei Mammiferi uno dei due cromosomi X è eterocromatico (v. Lyon, 1970). Studi di B. M. Cattanach e altri che dal 1967 ebbero come oggetto il topo (Mus), in cui parte di un cromosoma X era attaccato ad uno degli altri cromosomi in seguito ad un riordinamento strutturale, hanno dimostrato l'esistenza di un effetto diffusivo dell'eterocromatina del cromosoma X nei confronti dell'altro cromosoma, simile a quello osservato da Caspersson e Schultz in Drosophila. Cattanach e altri hanno ipotizzato l'esistenza nel cromosoma X di un elemento di controllo responsabile di determinare la lunghezza della zona del cromosoma che viene inattivata per diffusione.
Il termine di elemento di controllo fu introdotto da B. McClintock nel 1957 per indicare i fattori da lei studiati nel granoturco (Zea) e che sembravano controllare l'azione dei geni. Questi fattori risultavano localizzati nelle regioni eterocromatiche dei cromosomi e potevano causare dei fenotipi variegati attraverso due processi: per un effetto di diffusione ed inducendo mutazioni con alta frequenza. Molti altri studi sono stati condotti negli ultimi anni sui geni mutabili, per esempio da R. A. Brink e collaboratori nel granturco e da B. J. Harrison e J. R. S. Fincham in Antirrhinum majus.
Le prove che l'eterocromatina non sia geneticamente inerte, come si è più volte sostenuto, ci provengono da esperimenti con diversi organismi. K. Mather nel 1941 trovò che diversi cromosomi Y, eterocromatici, in Drosophila potevano influenzare il numero delle setole dei moscerini; mentre H. Rees e altri hanno dimostrato che al variare del numero degli extracromosomi eterocromatici (denominati cromosomi B) può variare in diverse piante superiori l'epoca della fioritura, la fertilità e la frequenza dei chiasmi. Sembra anche abbastanza provato che i geni nell'eterocromatina possano influenzare i caratteri quantitativi: e questo è abbastanza spiegabile se si attribuisce a quei geni una funzione di regolatori.
Al DNA dei cromosomi di molti eucarioti sono associate delle proteine basiche denominate istoni che sembrano potersi raggruppare in almeno 5 tipi principali. J. Bonner e collaboratori nel 1963 scoprirono che l'associazione degli istoni con il DNA è organo-specifica e si mantiene anche nella cromatina isolata. Dopo aver estratto le nucleoproteme dagli abbozzi fiorali e dai cotiledoni di piante di pisello (Pisum sativum) essi paragonarono la loro capacità di sintetizzare proteine in vitro usando il sistema per la sintesi proteica messo a punto da Nirenberg e Matthaei (v., 1961) a partire da estratti di Escherichia coli. Bonner trovò che le nucleoproteine dei germogli, a differenza di quelle provenienti dai cotiledoni, non riuscivano a sintetizzare una globulina caratteristica dei cotiledoni a meno che gli istoni non fossero stati rimossi dal DNA.
Nel 1968 J. Paul e R. S. Gilmour scoprirono che la specificità nel mascherare il DNA risiedeva in una frazione proteica che non era un istone. Con l'uso di nucleoproteine estratte da tessuti di mammifero trovarono che l'RNA specifico del tessuto sintetizzato dalle nucleoproteine dipendeva dalla presenza di queste proteine acide che prevenivano l'azione di mascheramento degli istoni. Se le proteine acide venivano allontanate dal DNA, anche la specificità di tessuto veniva a perdersi: l'aggiunta di istoni inoltre mascherava il DNA in modo completo. Il fatto che le proteine acide siano responsabili dell'attivazione di geni specifici è di particolare interesse in quanto si è accertato che anche le proteine che controllano l'attività dell'opero- ne lattosio in Escherichia coli e quelle del fago λ sono proteine acide.
Un'ulteriore prova che l'associazione regolativa del DNA con gli istoni non è specifica deriva dalle osservazioni di R. J. De Lange, D. M. Fambrough, E. L. Smith e Bonner nel 1969 che dimostrarono come l'istone IV di Pisum sativum sia quasi identico strutturalmente a quello di Bos taurus, con la sola differenza di due su 120 amminoacidi, e con una metilazione di più in un terzo. Per spiegare la conservazione quasi completa della sequenza degli amminoacidi nell'istone IV per centinaia di milioni di anni dal momento in cui pisello e bove si diversificarono nell'evoluzione, De Lange e collaboratori conclusero che la molecola degli istoni interagisce con l'impalcatura elicoidale del DNA e non ha rapporti con le basi responsabili della specificità dei geni (v. De Lange e Smith, 1971).
Sebbene vi sia qualche evidenza, come quella fornita da Paul e Oilmour, che indica come negli eucarioti il controllo dell'attività genica avvenga a livello della trascrizione, cioè a quello della sintesi dell'RNA messaggero dal DNA del gene, il controllo al momento della trascrizione non sarebbe efficace nel determinare con precisione l'ambito di attività del gene a meno che, come nel caso di Escherichia coli, l'RNA messaggero non venga rapidamente depolimerizzato. T. Spencer e H. Harris scoprirono nel 1964 che nell'alga verde Acetabularia la sintesi di un enzima specifico poteva verificarsi ancora per molti giorni dopo la rimozione del nucleo della cellula. Questa e altre osservazioni convaliderebbero invece l'ipotesi che il controllo dei livelli enzimatici negli eucarioti possa aver luogo nel citoplasma, presumibilmente a livello della traduzione dell'RNA messaggero in polipeptidi. Come abbiamo già indicato, la traduzione è un processo complesso ed è abbastanza probabile che il suo controllo possa avvenire in numerose e diverse maniere.
7. Le basi dell'ereditarietà non-mendeliana
Il controllo citoplasmatico dell'attività di un gene nucleare non è tuttavia la spiegazione dell'ereditarietà citoplasmatica di cui abbiamo già dato qualche esempio. J. T. O. Kirk nel 1963 scopri che i cloroplasti di Vicia faba contenevano del DNA con una quantità di guanina-citosina di circa il 37,5%, cioè più bassa in maniera significativa di quella del DNA nucleare (v. Kirk, 1971). L'ammontare del DNA in un cloroplasto è risultato essere tra i 2 e i 10 fg. La rapidità con cui le catene di nucleotidi separate si ricostituiscono in seguito a lento raffreddamento suggerisce che vi siano delle ripetizioni piuttosto estese di sequenze nucleotidiche. Le stime suggeriscono che vi siano 20-30 ripetizioni, il che implicherebbe 0,2-0,3 fg di DNA a sequenze specifiche, sufficienti a codificare 200-300 geni di 1.000 coppie nucleotidiche. Con la scoperta del DNA nei cloroplasti è stato naturale attribuire l'eredità citosplasmatica dei caratteri dei plastidi a questo DNA; tuttavia sarebbe sbagliato supporre che tutti i caratteri dei plastidi siano ereditati su base citoplasmatica. Infatti si conoscono molti casi, per esempio in Zea, di caratteri dei plastidi determinati da geni nucleari. In accordo con questo è stato dimostrato che geni nucleari controllano alcune reazioni per la sintesi della clorofilla a e b, dei carotenoidi e della catena di trasporto degli elettroni nella fotosintesi. D'altra parte esistono delle prove sulla sintesi dell'RNA ribosomale dei cloroplasti ad opera del DNA cloroplastico. Il che non era inatteso, dato che i ribosomi dei cloroplasti sono composti da un RNA di dimensioni inferiori a quello che si trova negli altri ribosomi cellulari a localizzazione citoplasmatica. I ribosomi dei cloroplasti somigliano a quelli dei batteri e questo, unitamente alla natura autonoma autoduplicantesi dei plastidi e al loro contenuto di DNA, ha fatto formulare l'ipotesi che i plastidi si siano potuti evolvere da organismi procariotici che hanno invaso la cellula eucariotica - una associazione simbiontica di due organismi piuttosto diversi (v. cellula: Fisiologia della cellula).
Sin dal 1968 gli studi di U. W. Hallier e collaboratori e di F. Herrmann e collaboratori sui mutanti per eredità plastidica rispettivamente di Oenothera e di Antirrhinum majus, hanno suggerito che questi mutanti potessero essere dovuti ad un difetto nelle proteine della membrana tilacoide del cloroplasto (v. Kirk, 1971). R. Sager e Z. Ramanis inoltre avevano trovato sin dal 1965 che i mutanti di Chlamydomonas reinhardii ad eredità non-mendeliana per la resistenza alla streptomicina e la richiesta di acetato mostravano associazione e ricombinazione ed erano riconducibili ad una struttura in DNA. Vi è quindi un'alta probabilità che questi caratteri siano determinati dal DNA del plastidio e questo è convalidato dalla scoperta nel 1970 di N. W. Gillham, J. E. Boynton e B. Burkholder che una mutazione ad eredità non-mendeliana per la resistenza alla streptomicina alterava i ribosomi dei cloroplasti.
B. Ephrussi nel 1958 scoprì che in un ceppo a lenta crescita di Saccharomyces cerevisiae (lievito), denominato petite, questa caratteristica era ereditata citoplasticamente e consisteva nella mancanza di un certo numero degli enzimi respiratori trovati nel mitocondrio. Nel 1965 fu dimostrato che i mitocondri contenevano una quantità di DNA non inferiore a 0,02 fg con una composizione in basi alquanto caratteristica: nel 1966 J. C. Mounolou e collaboratori trovarono che i mutanti citoplasmatici petite presentavano un DNA mitocondriale di densità anormale. Studi compiuti in altri mutanti mitocondriali da P. P. Slonimski e collaboratori e da D. Wilkie e collaboratori hanno fornito le prove di associazione e ricombinazione tra questi mutanti. Come per i plastidi, anche i mitocondri hanno dei ribosomi di tipo procariotico e vi sono alcuni dati che suggeriscono come l'RNA ribosomale dei mitocondri possa essere codificato dal DNA mitocondriale (v. Ashwell e Work, 1970).
La maggior parte degli esempi noti di ereditarietà citoplasmatica riguarda i plastidi o i caratteri respiratori. Questi casi trovano adesso una spiegazione plausibile in termini di DNA dei cloroplasti e dei mitocondri, rispettivamente, anche se soltanto in un numero piuttosto scarso di si tuazioni questo rapporto è stato accertato definitivamente. Ciò non di meno, l'aver realizzato sin dal 1965 che questa spiegazione era possibile ha avuto una notevole influenza sulle teorie circa i meccanismi ereditari.
8. Conclusioni
Per lo studio dei meccanismi ereditari è necessario trovare le differenze per un carattere tra due individui e poter studiare questi caratteri nella loro progenie. Questo automaticamente restringe lo studio dell'ereditarietà ad organismi relativamente affini che possano essere incrociati. Un corollario di questa situazione è che non è possibile studiare l'ereditarietà della grande gamma di caratteri che gli organismi affini hanno in comune. Si potrebbe così pensare che l'ereditarietà di tipo mendeliano spieghi le differenze tra specie affini, mentre l'ereditarietà di tutti i caratteri che rendono un organismo, per così dire, un animale invece che una pianta, risiederebbe nel citoplasma, come è stato dimostrato per alcuni caratteri di organismi strettamente affini. Oggi questa supposizione sembra molto meno plausibile di quanto non fosse qualche anno fa, in quanto è possibile che tutte le differenze ereditarie tra gli organismi simili siano dovute a differenze tra i loro acidi nucleici, siano essi nel nucleo o negli organi citoplasmatici quali i plastidi o i mitocondri. Sembra pertanto abbastanza ragionevole supporre che anche le differenze tra organismi meno affini possano avere una causa simile. Nonostante tutto questo il dubbio rimarrà sino a quando non si giungerà a dimostrare che l'informazione, sotto forma di sequenze nucleotidiche negli acidi nucleici, è in grado di specificare lo sviluppo di un'organizzazione tridimensionale straordinariamente complessa, come quella del cervello umano.
Gli studi di R. H. Epstein, R. S. Edgar, W. B. Wood e collaboratori a partire dal 1964 sulla sintesi del virus T4 di Escherichia coli suggeriscono che la sequenza con cui i vari componenti sono messi insieme è determinata non dall'attivazione sequenziale dei loro geni strutturali, ma dalle caratteristiche strutturali degli stessi componenti. In altre parole il montaggio è automatico, con la parte a che si adatta alla parte b e con il loro insieme che si combina con un complesso delle parti c e d, e così via. Un'analogia potrebbe essere rappresentata da uno di quei giochi di pazienza a incastro che possono essere messi insieme solo con una sequenza particolare. Può essere che anche negli organismi superiori il processo per lo sviluppo di particolari strutture si attui in maniera similare con le varie sequenze imposte dalle caratteristiche strutturali dei componenti.
I rimanenti decenni del XX secolo molto probabilmente vedranno un notevole progresso verso la comprensione di questo aspetto essenziale della materia vivente, le relazioni tra i meccanismi dell'ereditarietà e il controllo dello sviluppo.
Bibliografia
Ashwell, M., Work, T. S., The biogenesis of mitochondria, in ‟Annual review of biochemistry", 1970, XXXIX, pp. 251-290.
Auerbach, C., Robson, J. M., The production of mutations by chemical substances, in ‟Proceedings of the Royal Society of Edinburgh" (Section B), 1947, LXII, pp. 271-283.
Avery, O. T., MacLeod, C. M., McCarty, M., Studies on the chemical nature of the substance inducing transformation of pneumococcal types. Induction of transformation by a desoxyribonucleic acid fraction isolated from pneumococcus Type III, in ‟Journal of experimental medicine", 1944, LXXIX, pp. 137-158.
Baglioni, C., Correlations between genetics and chemistry of human haemoglobins, in Molecular genetics (a cura di J. H. Taylor), vol. I, New York 1963, pp. 405-475.
Baker, W. K., Position effect variegation, in ‟Advances in genetics", 1968, XIV, pp. 133-169.
Bateson, W., Mendel's principles of heredity, Cambridge 1909.
Baur, E., Das Wesen und die Erblichkeitsverhältnisse der varietates albomarginatae von Pelargonium zonale, in ‟Zeitschrift für Vererbungslehre", 1909, I, pp. 330-351.
Beadle, G. W., Genetics and metabolism in Neurospora, in ‟Physiological reviews", 1945, XXV, pp. 643-663.
Beadle, G. W., Tatum, E. L., Genetic control of biochemical reactions in Neurospora, in ‟Proceedings of the National Academy of Sciences", 1941, XXVII, pp. 499-506.
Benzer, S., Fine structure of agenetic region in bacteriophage, in ‟Proceedings of the National Academy of Sciences", 1955, XLI, pp. 344-354.
Brachet, J., Biochemical cytology, New York 1957.
Burgess, R., RNA polymerase, in ‟Annual review of biochemistry", 1971, XL, pp. 711-740.
Cairns, J., The bacterial chromosome and its manner of replication as seen by autoradiography, in ‟Journal of molecular biology", 1963, VI, pp. 208-213.
Callan, H. G., The nature of lampbrush chromosomes, in ‟International review of cytology", 1963, XV, pp. 1-34.
Chargaff, E., Chemical specificity of nucleic acids and mechanism of their enzymatic degradation, in ‟Experientia", 1950, VI, pp. 201-209.
Correns, C., Vererbungsversuche mit blass (gelb) grünen und buntlättrigen Sippen bei Mirabilis Jalapa, Urtica pilulifera und Lunaria annua, in ‟Zeitschrift für Vererbungslehre", 1909, I, pp. 291-329.
Crick, F. H. C., Barnett, L., Brenner, S., Watts-Tobin, R. J., General nature of the genetic code for proteins, in ‟Nature", 1961, CXCII, pp. 1227-1232.
De Lange, R. J., Smith, E. L., Histones. Structure and function, in ‟Annual review of biochemistry", 1971, XL, pp. 279-314.
Dounce, A. L., Duplicating mechanism for peptide chain and nucleic acid synthesis, in ‟Enzymologia", 1952, XV, pp. 251-258.
Fincham, J. R. S., Genetic complementation, New York 1966.
Gamow, G., Possible relation between desoxyribonucleic acid and protein structures, in ‟Nature", 1954, CLXXIII, p. 318.
Garrod, A. E., Inborn errors of metabolism, London 1909.
Harris, H., Nucleus and cytoplasm, Oxford 19702.
Hess, O., Meyer, G. F., Genetic activities of the Y chromosome in Drosophila during spermatogenesis, in ‟Advances in genetics", 1968, XIV, pp. 171-223.
Jacob, F., Monod, J., Genetic regulatory mechanisms in the synthesis of proteins, in ‟Journal of molecular biology", 1961, III, pp. 318-356.
Johannsen, W., Elemente der exakten Erblichkeitslehre, Jena 1909.
Keyl, H. G., A demonstrable local and geometric increase in the chromosomal DNA of Chironomus, in ‟Experientia", 1965, XXI, pp. 191-193.
Keyl, H. G., Duplikationen von Untereinheiten der Chromosomalen DNS während der Evolution von Chironomus thummi, in ‟Chromosoma", 1965, XVII, pp. 139-180.
Khorana, H. G., Büchi, H., Ghosh, H., Gupta, N., Jacob, T. M., Kössel, H., Morgan, R., Narang, S. A., Ohtsukà, E., Wells, R. D., Polynucleotide synthesis and the genetic code, in ‟Cold Spring Harbour symposia on quantitative biology", 1967, XXXI, pp. 39-49.
Kirk, J. T. O., Chloroplast structure and biogenesis, in ‟Annual review of biochemistry", 1971, XL, pp. 161-196.
Knapp, E., Reuss, E., Risse, O., Schreiber, H., Quantitative Analyse der mutationsauslösenden Wirkung monochromatischen UV-Lichtes, in ‟Naturwissenschaften", 1939, XXVII, p. 304.
Knapp, E., Schreiber, H., Quantitative Analyse der mutationsauslösenden Wirkung monochromatischen UV-Lichtes in Spermatozoiden von Shaerocarpus, in Proceedings of the 7th international genetics congress (a cura di R. C. Punnett), Edinburgh 1939, pp. 175-176.
Kornberg, A., Biologic synthesis of DNA, in ‟Science", 1960, CXXXI, pp. 1503-1508.
Lederberg, J., Tatum, E. L., Gene recombination in Escherichia coli, in ‟Nature", 1946, CLVIII, p. 558.
Lucas-Lenard, J., Lipmann, F., Protein biosynthesis, in ‟Annual review of biochemistry", 1971, XL, pp. 409-448.
Lyon, M. F., The activity of the sex chromosomes in mammals, in ‟Science progress", 1970, LVIII, pp. 117-130.
Meselson, M., Stahl, F. W., The replication of DNA in Escherichia coli, in ‟Proceedings of the National Academy of Sciences", 1958, XLIV, pp. 671-682.
Miller, O. L., Beatty, B. R., Visualization of nucleolar genes, in ‟Science", 1969, CLXIV, pp. 955-957.
Morgan, T. H., Sturtevant, A. H., Muller, H. J., Bridges, C. B., The mechanism of Mendelian heredity, New York 1915.
Moses, M. J., Synaptinemal complex, in ‟Annual review of genetics", 1968, II, pp. 363-412.
Muller, H. J., Artificial transmutation of the gene, in ‟Science", 1927, LXVI, pp. 84-87.
Nirenberg, M. W., Matthaei, J. H., The dependence of cell-free protein synthesis in E. coli upon naturally occurring or synthetic polyribonucleotides, in ‟Proceedings of the National Academy of Sciences", 1961, XLVII, pp. 1588-1602.
Pelling, C., A replicative and synthetic chromosomal unit, the modern concept of the chromomere, in ‟Proceedings of the Royal Society" (Series B), 1966, CLXIV, pp. 279-289.
Taylor, J. H., Woods, P. S., Hughes, W. L., The organization and duplication of chromosomes as revealed by autoradiographic studies using tritium-labeled thymidine, in ‟Proceedings of the National Academy of Sciences", 1957, XLIII, pp. 122-128.
Watson, J. D., Crick, F. H. C., A structure for deoxyribose nucleic acid, in ‟Nature", 1953, CLXXI, pp. 737-738.
Watson, J. D., Crick, F. H. C., Genetical implications of the structure of deoxyribonucleic acid, in ‟Nature", 1953, CLXXI, pp. 964-967.
Whitehouse, H. L. K., Chromosomes and recombination, in ‟Brookhaven symposia in biology", 1971, XXIII, pp. 293-325.
Citogenetica
di Claudio Barigozzi
Sommario: 1. Introduzione. 2. Il nucleo interfasico: a) i nuclei politenici; b) eucromatina ed eterocromatina; c) la replicazione del DNA; d) la cromatina sessuale; e) i cromosomi ‛a spazzola'. 3. I cromosomi durante il ciclo mitotico: a) il differenziamento longitudinale dei cromosomi; b) la ripartizione dei cromosomi nelle cellule figlie della mitosi; c) l'endomitosi e la C-mitosi. 4. I cromosomi nella meiosi: a) l'appaiamento; b) la ricombinazione (crossing-over); c) i chiasmi; d) i cromosomi sessuali; e) anomalie strutturali e di distribuzione; f) appaiamento incompleto ed eliminazione di cromosomi. 5. I cromosomi nel differenziamento somatico e nelle colture in vitro. □ Bibliografia.
1. Introduzione
I cromosomi sono oggetto di studio da oltre un secolo. A partire dall'inizio di quello in corso, dopo che fu dimostrato che essi sono la sede di quel patrimonio ereditario che si trasmette secondo le leggi di Mendel, l'interesse nei loro confronti si è enormemente accresciuto. Queste leggi, che permettono di prevedere con quali frequenze si trasmettono i caratteri ereditari, postularono fin da quando furono formulate (1865) l'esistenza di strutture regolari costanti per numero e per composizione, trasmissibili da una generazione all'altra. Le osservazioni citologiche però avevano già fornito la dimostrazione della costanza numerica e strutturale di corpi colorabili (visibili durante la divisione cellulare) appunto chiamati cromosomi, che, col loro comportamento, soddisfacevano pienamente alle esigenze della trasmissione mendeliana. Si formò così, dopo la riscoperta delle leggi di Mendel (1900), un sistema di nozioni che rappresentò da un lato il corpo dottrinale di una citologia estremamente specializzata, dall'altro la base direttamente osservabile dei fenomeni ereditari. Nacque così la citogenetica, branca della genetica, il cui dominio tende, col raffinarsi delle tecniche, a diventare sempre più ampio, avvicinandosi all'analisi della struttura molecolare del cromosoma. A questo punto appare chiara la confluenza dell'analisi fine di certi cromosomi con quella della struttura del gene, e i risultati tendono a sovrapporsi. Lo studio dei cromosomi ha rivestito, col tempo, anche un'importanza pratica, perché contribuisce spesso al miglioramento delle colture vegetali.
Di recente, infine, i cromosomi umani hanno assunto grande interesse anche in clinica, per la diagnosi di certe sindromi e in riferimento alla loro possibile trasmissione; un profondo rinnovamento tecnico ha portato a finissime distinzioni strutturali entro il cromosoma, ignote fino a pochissimi anni fa. Lo scopo di questo articolo è di presentare una descrizione concisa, ma la più attuale possibile, della morfologia e della struttura dei cromosomi e dei fenomeni della loro trasmissione attraverso la divisione cellulare.
2. Il nucleo interfasico
Il nucleo cellulare degli eucarioti, nel periodo compreso tra una mitosi e la successiva, viene definito interfasico o in riposo. In realtà, questa seconda espressione è poco felice, perché, anche quando la cellula non è in fase di divisione, il nucleo non si trova in riposo: infatti i geni, come si può osservare al microscopio in condizioni particolarmente favorevoli, sono attivi e inoltre ha luogo, in questo periodo, la sintesi dell'acido desossiribonucleico (DNA). Tali fenomeni, che contraddistinguono dunque una fase di intensa attività, hanno come conseguenza la duplicazione del contenuto cellulare di DNA, quindi la replicazione dei geni o il raddoppio del numero di cromosomi in caso di poliploidia e sono l'espressione della preparazione della cellula alla successiva fase mitotica.
Principale costituente del nucleo è la cromatina, una sostanza morfologicamente caratterizzata da una struttura filamentosa o a zolle, nella quale sono chimicamente individuabili elevate quantità di acidi nucleici (DNA e RNA), proteine, istoni e numerosi ioni. La particolare ricchezza in acidi del nucleo ne rende possibile la colorazione con i coloranti basici, mentre con la reazione di Feulgen, specifica per la dimostraziqne dell'acido desossiribonucleico, le strutture contenenti DNA si colorano elettivamente in rosso-viola. Il DNA, che come è noto rappresenta il materiale genetico, è chimicamente legato agli istoni; l'RNA, cioè l'acido ribonucleico, è presente nel nucleo in due tipi, il ribosomale o r-RNA, localizzato nei nucleoli, e il DNA simile o D-RNA o c-RNA (chromosomal RNA), associato alla cromatina e probabilmente identificabile in una forma di RNA forse in grado di reprimere la funzione del DNA.
Il complesso descritto non occupa interamente il nucleo nel quale è presente infatti anche il cosiddetto succo nucleare, assai ricco di proteine e contenente a sua volta DNA e vari tipi di RNA. Tutto il nucleo, infine, è avvolto dalla membrana nucleare (v. acidi nucleici; v. biologia; v. cellula: Fisiologia della cellula).
Come si è detto, questo nucleo ‛tipo', che si differenzia in varie forme, è quello degli eucarioti (v. fig. 1); i procarioti (Batteri e virus), infatti, non hanno un vero nucleo ma un cromosoma formato, come risulta dalle ricerche di genetica, da solo acido nucleico (sempre DNA nei Batteri, talvolta RNA nei virus) e organizzato in un filamento di solito anulare nei Batteri (v. fig. 2), anulare o aperto nei virus.
Si comprende facilmente, da quanto si è detto, che i cromosomi sono presenti anche nel nucleo interfasico; né potrebbe essere diversamente. Occorre infatti tener presente che, poiché i geni spesso funzionano in rapporto alla loro posizione così che l'allineamento ne costituisce la premessa indispensabile al modo di funzionare, un cromosoma, ridotto alla sua struttura essenziale, può essere considerato come un puro allineamento di geni.
Nei diversi tessuti dell'individuo e nelle diverse specie il nucleo interfasico presenta una fisionomia molto tipica, anche se quasi sempre nsulta impossibile precisame l'esatta struttura: infatti i filamenti di cromatina sono spesso così sottili e aggrovigliati da non svelare neppure la loro presenza. Una spiegazione che viene data di tale fenomeno è che durante l'interfase i cromosomi sono scarsamente fissabili e colorabili e tendono a formare piccole masse che alterano notevolmente la morfologia nucleare nella cellula viva; d'altro canto, senza fissazione e colorazione, il nucleo appare spesso otticamente quasi vuoto. Un valido ausilio alla comprensione della struttura del nucleo interfasico è offerto dallo studio del cosiddetto nucleo politenico, da cui si possono trarre numerose informazioni riguardanti anche la natura del gene e la struttura cromosomica.
a) I nuclei politenici
Si definisce politenico un nucleo nel quale, in seguito a un processo di accrescimento detto ‛endomitosi', di cui si parlerà in seguito, ogni filamento cromosomico è presente molte volte (decine o centinaia) e i vari filamenti cromosomici uguali tendono a rimanere accostati per tutta la loro lunghezza, costituendo grossi fasci. I nuclei politenici, tranne che nel gruppo dei Ditteri Cecidomudi, non danno mai luogo a divisioni, sono cioè in stato di interfase permanente. Dato il numero elevato di copie uguali di cromosomi che contengono, essi sono assai voluminosi e tale caratteristica costituisce già una condizione assai favorevole all'osservazione; inoltre, non solo si fissano e si colorano con grande facilità, ma in molti casi sono analizzabili agevolmente anche a fresco.
Finora i nuclei politenici sono stati rinvenuti in un limitato numero di casi: nelle cellule del sospensorio dell'embrione di certe piante (per es. di Phaseolus coccineus), in quasi tutte le cellule somatiche dei Ditteri e dei Collemboli. I nuclei politenici delle cellule vegetali non hanno dato luogo, almeno fino a oggi, ad analisi genetiche importanti; la loro struttura, molto semplice, è caratterizzata dalla disposizione dei filamenti cromosomici in grossi fasci che non presentano differenziamenti longitudinali a bande.
I nuclei politenici che hanno assunto una vera importanza generale, e sui quali ci intratterremo in modo particolare, sono invece quelli dei Ditteri, specialmente dei Chironomidi e del genere Drosophila: i loro cromosomi sono anche indicati come ‛cromosomi giganti' o ‛cromosomi salivari', denominazione quest'ultima derivata dal fatto che l'organo maggiormente utilizzato per studiarli è la ghiandola salivare della larva (v. fig. 3).
I cromosomi salivari hanno in comune una struttura trasversale a strie, dette bande, alternativamente colorabili e non colorabili, così da dare l'impressione di essere formati da dischi di cromatina intercalati a dischi non cromatinici; inoltre, i cromosomi omologhi, cioè quelli che nello zigote provengono uno da un genitore e uno dall'altro, sono in appaiamento stabile, designato come sinapsi somatica. In realtà i filamenti cromosomici sono continui, cioè ognuno è formato da due semieliche di DNA (più le altre sostanze che si trovano nei cromosomi degli eucarioti) prolungantisi senza interruzione da un estremo all'altro del cromosoma, le quali in punti costanti, intercalati da tratti estesi, presentano tratti disposti in stretto avvolgimento: in tal modo questi formano una specie di granuli, detti cromomeri, simili a quelli che si riscontrano nello stadio pachitene della meiosi (v. sotto), e raggiungono uno spessore tale da poter essere individuati al microscopio ottico.
Poiché tutti i cromosomi uguali formano fasci regolari, i loro cromomeri risultano situati agli stessi livelli, e da ciò deriva l'aspetto di banda o di disco. I tratti nei quali i filamenti cromosomici non sono avvolti su se stessi sono così sottili da essere individuabili con chiarezza solo mediante il microscopio elettronico. La successione delle bande è estremamente costante in ogni specie, a eccezione dei casi di alterazioni mutazionali di cui parleremo in seguito, e il loro spessore e le distanze che le separano - oltre a strozzature e rigonfiamenti dell'intero cromosoma - ne permettono un'individuazione precisa, che ha dato luogo a un minuzioso simbolismo per la loro identificazione (v. fig. 4).
Nello studio dei cromosomi salivari il problema fondamentale consiste nell'individuare la posizione occupata su di essi dai geni.
Mediante l'utilizzazione di mutazioni cromosomiche (delezioni, inversioni, traslocazioni o duplicazioni) furono identificate, con varia approssimazione, le sedi di moltissimi geni; in alcuni casi fortunati è stato possibile localizzare un gene in una singola banda, ma per lo più la sede di un gene viene indicata entro un complesso di alcune bande, anche se ciò non significa che un gene comprenda più bande. Il caso analizzato più in profondità è quello del locus complesso o cistrone white in D. melanogaster, che controlla il colore dell'occhio, collocato a 1,5 unità di ricombinazione dall'estremità libera (convenzionalmente la sinistra) del cromosoma X. Questo cistrone comprende una serie di siti (sequenze di nucleotidi) che possono mutare indipendentemente ed essere separati per ricombinazione; esso sembra essere localizzato in una singola banda, la cui struttura è stata anche analizzata molto a fondo mediante micrografie elettroniche. Si è fatta dunque strada l'idea che in ogni cromomero sia localizzato un gene o cistrone, o al massimo due cistroni. Il numero di bande note supera quello dei geni conosciuti, ma è oggi opinione diffusa che questi siano ancora pochi in confronto a quelli realmente esistenti: si consideri infatti, ad esempio, che fra il locus yellow (corpo giallo) situato all'estremità del cromosoma X e il locus prune (occhio color prugna secca), tra loro assai vicini (distanza calcolata sulla ricombinazione: 0,8 unità), vi sono ben 57 bande. Secondo l'interpretazione che oggi trova maggior credito, fra i due loci noti debbono esservi almeno 57 cistroni (v. fig. 4). Recentemente è stato prospettato il problema dell'effettiva sede dei cistroni: infatti, in contrasto con la tradizionale teoria secondo la quale il locus sarebbe collocato nella banda cromatinica, e cioè nel cromomero, è stata formulata l'ipotesi che questa potrebbe in realtà essere la sede di strutture capaci di regolare la funzione del gene strutturale, il quale sarebbe invece collocato nella banda non cromatinica. Questa ipotesi, che chiaramente estende ai geni della drosofila il modello dell'operone della β-galattosidasi di E. coli, non può essere oggi né accolta né respinta, e non si può escludere categoricamente che i dischi incolori possano essere sede di loci genici.
Dallo studio dei nuclei politenici dei Ditteri ci proviene, inoltre, la maggior parte delle informazioni sull'attività dei geni: i loro cromosomi, infatti, mostrano rigonfiamenti temporanei specifici, detti puffs, che indicano l'entrata in funzione di singoli cistroni oppure di operoni (v. fig. 5). Si conoscono due tipi di puffs: uno, riscontrabile specialmente nel gruppo degli Sciaridi, è rappresentato dai rigonfiamenti che sono sede di sintesi di DNA; l'altro, più generalmente diffuso fra i Ditteri, comprende invece i rigonfiamenti nei quali ha luogo la sintesi di RNA messaggero (m-RNA). Questo secondo gruppo, proprio per la natura multipla dei cromosomi di cui si parla, viene considerato il modello ingigantito della funzione genica visualizzata. Il prodotto primario dei geni che danno luogo a questo tipo di puffs è infatti, come s'è detto, m-RNA, e tale interpretazione risulta convalidata da varie osservazioni. Prescindendo dalla possibilità di induzione sperimentale di puffs mediante l'impiego di numerose sostanze, anche costituite da molecole di piccole dimensioni, è noto che molti puffs insorgono in momenti tipici dello sviluppo: è stato ad esempio dimostrato che in D. virilis il trattamento con ecdisone, cioè con l'ormone che controlla l'impupamento (v. ormoni negli invertebrati), è in grado di indurre un puff la cui comparsa è seguita dall'inizio della formazione dell'enzima dopa-carbossilasi, preposto alla tannificazione della cuticola che diventerà pupario: è questo il caso più manifesto di parallelismo fra formazione di un puff ed entrata in funzione di un gene.
L'elemento tuttora non chiarito del quadro, così presentato, rimane pur sempre la reale estensione dei geni nei puffs; tuttavia, malgrado questa lacuna, appare del tutto logico che i geni funzionino in questo o in analogo modo alterando la struttura del cromosoma durante il periodo della loro funzione, e che d'altra parte il fenomeno passi inosservato nel caso di cromosomi non giganteschi come quelli dei nuclei politenici. Questi ultimi sono invece meno utili come modello generalizzabile della replicazione del DNA in quanto, per la loro struttura a numerosi filamenti paralleli, si distaccano troppo dai cromosomi di tipo ordinario.
Mediante l'impiego della timidina triziata è comunque facile dimostrare che i cromosomi politenici sono sede di replicazione di DNA: tale attività biosintetica è in perfetto accordo coi fenomeni di endoreduplicazione di quei filamenti, detti cromatidi (v. sotto), originariamente presenti in numero di due per ciascun cromosoma, fenomeni che avvengono per mezzo di un processo di divisioni ripetute all'interno del nucleo, cui si dà il nome di endomitosi. Si può aggiungere, a proposito della replicazione, che le bande non replicano sincronicamente, ma alcune mostrano una costante precedenza su altre; ogni banda, infine, va considerata come un elemento della replicazione, comunemente designato come ‛replicone'.
b) Eucromatina ed eterocromatina
Nel complesso cromosomico politenico, così come in generale in tutti i nuclei interfasici degli eucarioti e nei cromosomi mitotici, è molto importante la distinzione della cromatina in eucromatina ed eterocromatina. La prima corrisponde a tutte le zone cromosomiche a bande, ed è quindi quella di cui ci siamo occupati finora, e riguarda la maggioranza dei cromosomi; essa, inoltre, nel genere Drosophila corrisponde alle zone nelle quali sono localizzati i geni finora mappati, salvo poche eccezioni. L'eterocromatina, sempre nel genere Drosophila, non presenta invece struttura a bande, ma forma un ammasso di cromomeri che unisce tutte le parti eucromatiche in una zona di confluenza comune, alla quale si dà il nome di cromocentro. Questo, che dopo trattamento con chinacrina appare fortemente fluorescente, è di dimensioni assai modeste, malgrado corrisponda in realtà a una cospicua parte dei cromosomi mitotici (v. fig. 6): si consideri, ad esempio, che in D. melanogaster esso comprende tutta la cromatina che forma, nella mitosi, l'intero cromosoma Y, metà circa del cromosoma X, determinate parti, dette adiacenti al centromero (v. sotto), delle due coppie dei grandi autosomi denominati 2 e 3 e quasi per intero la coppia dei piccoli autosomi denominati 4. La ragione di questo divario dimensionale fra le due entità sta nel loro diverso ritmo di replicazione del DNA: l'eterocromatina, infatti, rispetto all'eucromatina si replica meno velocemente, cosicché la sua quantità rimane molto più vicina a quella inizialmente presente nel nucleo.
L'eterocromatina, per molti anni ritenuta funzionalmente inerte, è sede di attività importanti, anzi essenziali: tale è la sintesi dell'RNA ribosomale, che normalmente confluisce poi, per formare i ribosomi, nel nucleolo, presente in questi nuclei sotto forma di una massa globosa debolmente colorabile con l'orceina. Oggi sappiamo che tale attività sintetica è concentrata almeno in un punto del cromosoma X e del cromosoma Y, cioè in quella zona che agisce come organizzatore del nucleolo. Tale zona, che è stata localizzata nel cromocentro, appare costituita dall'allineamento di porzioni uguali ripetute molte volte; il DNA che la forma è dunque una lunga sequenza di cistroni uguali, caratterizzato dalla prevalenza di basi guanina-citosina. Per concludere, è certo che nel cromocentro dei nuclei politenici di D. melanogaster si trovano zone di DNA cui compete, tra varie funzioni (v. sotto), la sintesi dell'RNA ribosomale, attività che, in ogni caso, consente di considerare il nucleolo quale luogo di maturazione dei ribosomi. L'esistenza del cromocentro non è però prerogativa dei nuclei politenici di tutti i Ditteri: infatti nei Chironomidi manca completamente la tendenza all'attrazione fra tutte le parti eterocromatiche - nelle quali le bande appaiono poco chiare o sono del tutto assenti - che rimangono pertanto distinte per ogni cromosoma.
Dopo questa descrizione analitica del nucleo politenico, resta da vedere quanto questo modello, che consideriamo il più ricco di informazioni fra i nuclei interfasici, sia generalizzabile. Anzitutto possiamo affermarne la validità per quanto riguarda i nuclei interfasici di drosofila - così come si possono studiare nelle cellule dei gangli nervosi e in quelle coltivate in vitro - nei quali non esiste la politenia, essendo l'assetto cromosomico diploide, cioè uguale a quello dello zigote. Questi nuclei, che a loro volta non differiscono dalla generalità dei nuclei di qualsivoglia specie, mostrano all'osservazione al microscopio ottico una sottilissima trama di filamenti al limite della risolvibilità, un nucleolo e spesso un cromocentro fluorescente, formato da alcuni blocchi: appare chiaro che quest'ultima struttura corrisponde all'eterocromatina, mentre la parte filamentosa è identificabile con l'eucromatina. Sempre nel genere Drosophila, osservazioni di notevole interesse sono state compiute in D. hydei da Hess e Meyer (v., 1968) sugli spermatidi in via di differenziarsi in spermatozoi: nei nuclei ditali elementi è stato possibile mettere in evidenza formazioni a nastro o ad ansa, sporgenti dal cromosoma Y eterocromatico, le quali ne esprimono la funzione nei riguardi del differenziamento delle suddette cellule e costituiscono anche la prova di un altro aspetto dell'attività funzionale dell'eterocromatina. Tali formazioni possono senz'altro venire assimilate ai puffs osservabili nei cromosomi dei nuclei politenici, cosicché è possibile dimostrare ancora una volta che l'attività dei geni può manifestarsi con variazioni strutturali del cromosoma morfologicamente apprezzabili. Quasi sempre nell'eterocromatina è presente, in una o più forme di differente peso molecolare, e in esigua quantità, un DNA del quale non si conoscono le funzioni: questo è costituito da brevissime sequenze di basi, anche solo sette, e viene designato come DNA satellite, mentre l'altro DNA, quantitativamente prevalente, è definito principale.
c) La replicazione del DNA
I nuclei interfasici comuni, generalmente diploidi, ci forniscono, in misura notevolmente maggiore rispetto ai nuclei politenici, informazioni su due particolari aspetti della replicazione del DNA: il numero di eliche di DNA effettivamente presenti in un cromosoma di eucariote, problema della massima importanza dal momento che è dimostrato che nei procarioti il cromosoma consta di una sola doppia elica, e le modalità secondo le quali si svolge il processo di replicazione (v. acidi nucleici; v. biologia; v. cellula: Fisiologia della cellula). Nei Batteri la dimostrazione dello schema di replicazione del DNA, detto semiconservatore o semiconservativo, è basata sulla possibilità di valutare mediante un gradiente di densità il peso di doppie eliche, cioè di cromosomi, nelle quali il processo abbia avuto luogo con l'utilizzazione di 14N o di 15N. Senza entrare nei dettagli di questo esperimento, basterà riassumerne i risultati nel modo seguente: la replicazione di un cromosoma prima contenente solo 14N, avvenuta utilizzando 15N, conferisce al nuovo cromosoma un peso maggiore, e ancora più pesante risulterà il cromosoma contenente, a seguito di un'ulteriore replicazione avvenuta con 15N, solo 15N. Se il cromosoma con peso intermedio ha un'elica di DNA con 14N e una con 15N, il cromosoma con peso leggero sarà tutto con 14N e quello più pesante tutto con 15N: ciò significa che ogni nuova elica è copia di una vecchia, e in tale fenomeno consiste la semiconservatività del processo di replicazione. La dimostrazione che in un cromosoma di eucariote (a replicazione avvenuta) sono presenti due doppie eliche come nei procarioti, a eccezione di casi meno frequenti nei quali il loro numero può salire a quattro o eventualmente a otto, è fornita dai risultati di ricerche condotte sulle modalità di incorporazione di timidina triziata nei nuclei in fase di sintesi premitotica e sulle due mitosi seguenti tale fase. Durante una replicazione interfasica, infatti, la doppia elica presente viene copiata per formarne due nuove, una per ogni elica vecchia: se nello svolgimento di questo processo viene utilizzata timidina triziata, le metà longitudinali di ciascun cromosoma saranno costituite rispettivamente da DNA sintetizzato utilizzando l'isotopo radioattivo e da DNA preesistente non radioattivo. Nella mitosi successiva, studiata durante la metafase, che è la fase più adatta per compiere l'osservazione, ogni cromosoma apparirà diviso nelle due metà, o cromatidi, delle quali una sola radioattiva. Successivamente, eliminando dal mezzo di coltura la timidina triziata, dopo un secondo ciclo di replicazione sia le metà radioattive sia quelle non radioattive appariranno completate con metà non radioattive: risulteranno così cromosomi con un cromatidio radioattivo e cromosomi totalmente privi di radioattività. Queste osservazioni dimostrano da un lato che la replica del DNA avviene anche negli eucarioti, al pari dei procarioti, secondo il cosiddetto schema semiconservativo, dall'altro che le doppie eliche corrispondono ciascuna a un cromatidio, cioè a mezzo cromosoma (J. H. Taylor, 1958). Negli eucarioti, dunque, il cromosoma, costruito secondo questo piano strutturale, è doppio di quello dei procarioti. Tuttavia, si ha ragione di ritenere che questo piano strutturale non sia l'unico: nei Rincoti, infatti, si osservano anche mezzi cromatidi (v. fig. 7).
Il secondo aspetto della replicazione riguarda il tempo assoluto e relativo del suo svolgimento, studiato sempre mediante autoradiografia dopo incorporazione di timidina triziata. Per quanto riguarda il tempo assoluto, i risultati di tali ricerche hanno consentito di stabilire che in generale la replicazione, cosiddetta fase di sintesi o fase 5, occupa un periodo di poche ore - 1-2 o meno, con variazioni anche notevoli da caso a caso - che rappresenta un intervallo fra altri due periodi detti G1 e G2, sensibilmente più lunghi, fino a 18-20 ore, durante i quali non vi è sintesi. Durante la fase S la sintesi non è contemporanea o casualmente distribuita in tutti i punti dei cromosomi: è stato infatti dimostrato in molte specie animali e vegetali (A. Lima-de-Faria e Jaworska, 1968) che l'eterocromatina si replica nettamente più tardi dell'eucromatina, fenomeno questo che costituisce un altro elemento essenziale di distinzione tra le due cromatine, anche se ne sfugge tuttora il significato (v. fig. 8).
Non è stato ancora possibile delineare la struttura microscopica dei cromosomi nell'interfase, malgrado i numerosi tentativi compiuti per isolarli in tale stadio, in considerazione della notevole difficoltà rappresentata dalla loro risoluzione nei nuclei interi. In diverse specie è stato osservato che i cromosomi interfasici non sembrano differire troppo da quelli mitotici (v. Mirsky e Ris, 1947): per esempio negli Anfibi Urodeli i cromosomi interfasici, più lunghi e spessi di quelli di certi pesci, presentano una notevole corrispondenza con i rispettivi cromosomi mitotici. Tuttavia, attualmente non è facile stabilire con certezza in quale misura questi dati morfologici siano in realtà attribuibili ad artefatti. Recentemente (Bonner e Wu, 1973) si è osservato che il DNA proveniente da cromosomi isolati da nuclei interfasici di ratto è costituito da frammenti di molecole, di aspetto filamentoso, osservabili solo al microscopio elettronico. Sembra quindi più aderente alla realtà l'ipotesi che il DNA si presenti nella forma di molecole estese o estensibili piuttosto che in stato compatto, tanto più che la struttura molecolare del cromosoma interfasico del ratto sembra essere assai simile a quella dei cromosomi politenici di drosofila. Gli istoni, ai quali, come s'è detto, nel nucleo degli eucarioti è legato chimicamente il DNA, vengono sintetizzati subito dopo la replica.
Il nucleo interfasico come è stato fin qui descritto è così ampiamente rappresentato, almeno nelle cellule che vanno incontro a mitosi, da poter essere considerato un modello valido per quasi tutti gli eucarioti; fanno eccezione i Protozoi e forse molti vegetali inferiori, in realtà ancora scarsamente conosciuti, il cui nucleo (micro- e macronucleo) appare così intensamente compatto da non consentire quasi alcun grado di risoluzione all'osservazione microscopica.
d) La cromatina sessuale
Un'importante eccezione, anche se a prima vista potrebbe non apparire tale, è costituita dai nuclei interfasici di cellule somatiche di Mammiferi, in particolare dell'uomo. Nei nuclei di numerose cellule sia dell'embrione sia dell'aduito sono chiaramente visibili un insieme di filamenti sottili, un nucleolo e, nel sesso femminile, un cromocentro particolare, detto corpo di Barr: quest'ultima formazione presenta per lo più (nelle cellule delle mucose, per esempio) l'aspetto di una zolla angolosa addossata alla parete nucleare ed è intensamente colorabile e fluorescente. In un certo numero di leucociti polinucleati (5%) il corpo di Barr è sostituito da una masserella a forma di manubrio o di mazza di tamburo (drumstick in inglese). Secondo l'interpretazione comunemente accettata questa porzione di cr0matina, detta anche cromatina sessuale, rappresenta uno dei due cromosomi X della femmina che diventa compatto, cioè eterocromatico (eterocromatina facoltativa). Esistono prove genetiche a favore dell'inattivazione casuale di uno dei cromosomi X nel sesso femminile (cosiddetta teoria di M. Lyon), e la dimostrazione di tale fenomeno è fornita dalla comparsa di caratteri recessivi negli eterozigoti: per esempio, nelle donne eterozigoti per il gene che controlla l'enzima G6PD, alcune cellule sono G6PD positive e altre G6PD negative, in quanto l'allele attivo è presente solo nel cromosoma X che non si è eterocromatinizzato.
La suddetta interpretazione consente di differenziare nettamente il corpo di Barr - che nell'uomo e in altri Mammiferi costituisce soltanto l'espressione di uno stato funzionale - dal cromocentro dei nuclei politenici e di tutti gli altri finora considerati, che è invece costituito da eterocromatina stabile, cioè costitutiva. La dimostrazione del particolare significato funzionale dell'eterocromatinizzazione costituisce un importante elemento a favore della tesi secondo la quale quando un cromosoma permane compatto durante l'interfase, i suoi geni non funzionano: considerando costantemente valido questo principio ci si dovrebbe attendere il blocco funzionale in tutti i casi in cui la cromatina appare addensata o, come anche si dice, picnotica (v. fig. 9).
A prescindere dal suo aspetto teorico, la scoperta del corpo di Barr è comunque risultata importante dal punto di vista pratico per la diagnosi cromosomica del sesso. Infatti, la presenza di un corpo equivale alla costituzione XX, cioè al sesso femminile, e la sua assenza a quello maschile (l'inattivazione dell'unico X nel maschio avrebbe come conseguenza il blocco di troppi geni, e ne deriverebbe una condizione di letalità); la presenza di due corpi corrisponde a una costituzione anormale, XXX, e quella del corpo di Barr in un individuo di aspetto maschile è espressione della costituzione XXY, o sindrome di Klinefelter; l'assenza del corpo di Barr in individui di aspetto femminile corrisponde alla costituzione XO, caratteristica della sindrome di Turner. Lo studio su singole cellule della formula dei cromosomi sessuali non solo fornisce preziose indicazioni per la diagnosi di sindromi interessanti dal punto di vista clinico, ma permette anche la diagnosi prenatale del sesso: infatti mediante l'amniocentesi, consistente nel prelievo di liquido amniotico, è possibile ottenere cellule del feto e procedere agevolmente alla loro analisi (v. eugenica).
e) I cromosomi ‛a spazzola'
Ai nuclei interfasici si avvicinano molto i nuclei degli oociti di Anfibi (e probabilmente di Pesci), i quali vanno incontro a un periodo di interruzione della meiosi allo stadio diplotene (v. sotto): in essi, durante questa pausa che può durare anche mesi o anni, vengono sintetizzate abbondanti quantità di proteine per il deutoplasma dell'uovo ed entrano in funzione alcuni geni. Nel corso di questa particolare forma di interfase, di notevole importanza per lo studio della funzione genica, i cromosomi presentano una struttura del tutto particolare, per molti anni descritta come un asse longitudinale con ‛barbe' laterali a terminazione libera: di qui il nome loro assegnato di cromosomi a spazzola, o cromosomi piumosi, o lamp-brush chromosomes (v. fig. 10). In realtà le ‛barbe' laterali sono anse di varia ampiezza, ma costanti nella successione, come le bande dei cromosomi politenici. Il confronto con le anse dei filamenti politenici nei puffs è molto suggestivo, ma non può reggere alla critica: non sembra infatti possibile attribuire a ogni ansa laterale il significato di banda funzionante, perché indubbiamente la grande maggioranza dei geni in questi cromosomi è repressa durante lo stadio diplotene. La funzione è particolarmente vivace solo in alcune zone, variamente localizzate in specie diverse, ove ha luogo la sintesi di quell'RNA ribosomale destinato a formare un grande numero di piccoli nucleoli (100 o più nell'axolotl), che rapidamente si portano alla periferia del nucleo ove si dissolvono emettendo il loro contenuto nel citoplasma. Si ritiene che tale processo sia necessario alla sintesi delle proteine del deutoplasma che, come già si è detto, viene prodotto proprio durante questo periodo. L'elevato numero dei nucleoli indica già che il loro organizzatore è ripetuto molte volte, cioè - come si suol dire - amplificato, e ciò conferma quello che si è già detto a proposito dell'organizzatore del nucleolo della drosofila. Nei cromosomi a spazzola le sequenze ripetute sono state identificate come porzioni di filamento di DNA da ciascuna delle quali sporge lateralmente una serie ordinata di molecole di RNA, il quale - se realmente tale acido nucleico è, come si crede, del tipo ribosomale - sarebbe l'espressione della trascrizione in atto di geni che codificano l'RNA stesso. Nello Xenopus (anfibio anuro) è stato anche osservato che ogni piccolo nucleolo si forma da un anello di DNA staccatosi dal cromosoma. Gli oociti degli Anfibi sarebbero dunque caratterizzati da un aumento straordinario di ripetizioni di sequenze codificanti r-RNA, che si staccherebbero durante la sintesi di proteine al momento dell'accrescimento dell'uovo.
Possiamo dire, come conclusione, che i componenti del nucleo (cromatina, distinta in eu- ed eterocromatina, e nucleolo) sono costanti in tutti gli eucarioti, mentre nell'unico cromosoma dei procarioti è presente solo DNA o, limitatamente ad alcuni virus, RNA. L'eterocromatina, poi, può essere distinta, in base al suo assetto strutturale, in costitutiva (tipo Ditteri) e facoltativa (tipo cromosoma X dell'uomo). Per quanto riguarda la possibilità che la funzione dei geni si renda manifesta in modo tale da risultare morfologicamente riconoscibile, i dati sono finora limitati a pochi esempi riscontrabili in specie assai lontane, dei quali i meno controversi sono quelli dei puffs nei cromosomi politenici dei Ditteri e quello dei cistroni ripetuti che codificano per RNA ribosomale. È innegabile che la tendenza alla ripetizione di sequenze uguali prevalga nell'eterocromatina, ma sarebbe erroneo escludere, benché le osservazioni siano ancora scarse, la ripetitività di sequenze anche nell'eucromatina.
3. I cromosomi durante il ciclo mitotico
I singoli cromosomi durante il ciclo mitotico (distinto in profase, prometafase, metafase, anafase e telofase) vanno incontro a una serie di trasformazioni morfologico-strutturali culminanti nella loro ripartizione in metà, ciascuna delle quali è destinata a far parte del corredo cromosomico di ognuna delle due cellule figlie.
L'aspetto più appariscente delle trasformazioni osservabili nei cromosomi nel corso della mitosi, durante la quale la funzione dei geni in gran parte si arresta, può essere sinteticamente descritto come una modificazione delle caratteristiche di compattezza e colorabilità, in graduale incremento dalla profase alla metafase, seguita da un decremento altrettanto graduale dall'anafase alla telofase fino a che, a processo ultimato, appaiono ripristinate le stesse condizioni del nucleo interfasico da cui ha inizio il ciclo. In questa trattazione ci occuperemo solo dei fenomeni rilevabili nei cromosomi, mentre quelli che hanno luogo nel citoplasma saranno ricordati solo se strettamente necessari a spiegare i primi. Come abbiamo in precedenza descritto, il cromosoma interfasico dell'eucariote già replicato risulta costituito da almeno quattro eliche di DNA, due per ogni mezzo cromosoma o cromatidio; s'è inoltre detto che il filamento di DNA si considera come legato solo agli istoni, i quali insieme ad altri componenti entrano pure nella costituzione dei cromosomi. Dal momento che non siamo ancora in possesso di informazioni più dettagliate in proposito, la migliore esemplificazione che possiamo fornire delle modificazioni strutturali del cromosoma nel ciclo mitotico è la seguente.
Durante la profase e la prometafase esso si accorcia e s'ispessisce, molto verosimilmente mediante un meccanismo di avvolgimento a spirale o ‛spiralizzazione' (v. fig. 11). La formazione di spirali, talvolta di due ordini (una spirale minore che si avvolge in una spirale maggiore), visibili al microscopio ottico, è stata osservata e descritta soprattutto in Tradescantia, ma dimostrazioni simili sono possibili su numerose altre specie, anche animali. Non conosciamo il fattore o i fattori in grado di promuovere la spiralizzazione, nè d'altra parte ci sono note le modalità secondo le quali la componente proteica entra a far parte della struttura del cromosoma: l'esame di numerose micrografie elettroniche, mettendo in evidenza una serie di avvolgimenti complicatissimi, ha condotto alla conclusione che non è ancora possibile stabilire come la doppia elica di DNA sia disposta nel cromatidio.
L'aspetto del cromosoma metafasico, invece, può essere descritto nei suoi minuti particolari e rappresenta lo stato più idoneo per le ricerche morfologiche e strutturali. Durante la metafase, infatti, scomparsi la membrana nucleare e il nucleolo nel corso della prometafase, i cromosomi si dispongono nella piastra equatoriale mediante contatti fisici con le fibre del fuso e si realizzano quindi le migliori condizioni per il loro conteggio e per la loro risoluzione in parti. La possibilità così offerta di definire nei vari casi il numero e la morfologia dei cromosomi, sia pure prescindendo dalla loro organizzazione molecolare, si è rivelata di enorme importanza in patologia umana per la diagnosi di varie sindromi.
Alcune tecniche hanno contribuito in modo essenziale al progresso della conoscenza del cromosoma metafasico: la fissazione con acido acetico, che dissolve il citoplasma; l'impiego di soluzioni ipotoniche, che allontanano tra loro i singoli cromosomi e divaricano i due cromatidi di ciascuno di essi; le colorazioni specifiche per il DNA (colorazione di Feulgen e colorazione di Giemsa) e il trattamento coi derivati fluorescenti delle acridine, quale la chinacrina cui si è già fatto cenno. La grande massa di dati raccolta in questi ultimi anni (la fluorescenza fu introdotta da T. Caspersson e collaboratori nel 1970) è derivata da osservazioni condotte su cellule coltivate in vitro, ed è pertanto riferibile a cromosomi metafasici soprattutto di cellule somatiche (linfociti, ad esempio) oppure di cellule embrionali e di gangli nervosi della drosofila e di altri insetti.
Tali studi hanno anzitutto dimostrato, confermando i risultati di ricerche compiute già da molti anni con differenti tecniche, che nella quasi totalità dei casi ogni cromosoma consta di due bracci (v. fig. 12). Questi possono essere di lunghezza uguale, nel qual caso il cromosoma è detto mediocentrico, o di lunghezza diversa, e allora il cromosoma viene detto submediocentrico, subtelocentrico o telocentrico, in ragione della differenza progressivamente crescente fra i due bracci (v. fig. 13). L'ultima denominazione (sinonimo, per alcuni, di acrocentrico) è riferita a un braccio così breve da risultare quasi invisibile. Nel punto che separa i due bracci - un istmo sottilissimo detto strozzatura primaria o cinetocoro - si collocano due granuli o un solo granulo al limite della visibilità all'osservazione al microscopio ottico, il cosiddetto centromero, che rappresenta l'organulo deputato a prendere contatto con le fibre del fuso. In pratica, la distinzione fra cinetocoro e centromero spesso non è agevole, data l'impossibilità di discernere il granulo o i granuli centromerici. Il cinetocoro è l'unico punto di connessione dei cromatidi nella metafase. L'effetto divaricante esercitato sui cromatidi dallo shock da soluzione ipotonica, cui s'è gia accennato, è responsabile della forma con la quale il cromosoma si manifesta nelle comuni preparazioni: quando tale divaricazione è massima, i cromosomi medio e submediocentrici appaiono a croce o a X con braccia più o meno uguali, mentre i telocentrici e i subtelocentrici assumono forme a V o a Y. Questi aspetti hanno completamente sovvertito la morfologia dei cromosomi delle vecchie descrizioni, nelle quali i mediocentrici apparivano conformati a V. Di questa situazione estremamente generalizzata dei normali aspetti cromosomici, quando si prescinda dalle variazioni mutazionali, cioè da casi in cui sono presenti due centromeri (cromosomi dicentrici) o manca il centromero (frammenti acentrici) di cui si dirà più avanti, sono note solo poche eccezioni: le condizioni caratterizzate dalla mancanza di un centromero differenziato e quelle in cui sono stati descritti mezzi cromatidi e persino quarti di cromatidi. Esempi del primo tipo di eccezione sono rappresentati da Parascaris equorum e da alcuni Rincoti, fra gli animali, e dal genere Luzula fra le piante; in questi casi i cromosomi sono privi di una strozzatura primaria, o cinetocoro, e presentano uno spessore costante per tutta la lunghezza, sicché è impossibile distinguere in ognuno di essi due bracci. La presenza di mezzi cromatidi deriva dall'esistenza, in ogni mezzo cromosoma, di una spaccatura ortogonale a quella che separa i due cromatidi: non è ancora noto, tuttavia, se tale osservazione, in verità compiuta non molto recentemente nei Rincoti e in alcune piante, sia stata convalidata dall'applicazione di tecniche più moderne, tanto più che i dati genetici sembrano escludere l'esistenza di mezzi cromatidi.
a) Il differenziamento longitudinale dei cromosomi
L'aspetto più interessante del cromosoma, specialmente in considerazione degli ultimi sviluppi delle ricerche, è rappresentato dal suo differenziamento longitudinale. Anzitutto sono noti differenziamenti dovuti a strozzature, dette secondarie, che ripartiscono le braccia in porzioni minori. Talvolta si tratta di blocchi appena accennati, come nel cromosoma Y di D. melanogaster, oppure di lunghi assottigliamenti, che appaiono quasi come interruzioni del cromosoma, i quali separano una parte principale da una breve e terminale, simile a una piccola sfera, cui si dà il nome di ‛satellite'. In ogni cromosoma le strozzature secondarie sono generalmente caratterizzate da numero e posizioni fissi: di esse una almeno corrisponde all'organizzatore del nucleolo, che attualmente, generalizzando le osservazioni compiute su D. melanogaster, è identificabile in una sequenza di cistroni codificanti per l'RNA ribosomale, di cui abbiamo ripetutamente parlato. Le differenziazioni che non interessano lo spessore del cromosoma riguardano piuttosto la sua natura, cioè la compattezza, la colorabilità e il grado di fluorescenza. Nella terminologia di uso corrente si parla generalmente di eucromatina e di eterocromatina, secondo la stessa distinzione adottata a proposito dei nuclei interfasici; occorre tuttavia tener presente che gli unici dati certi sono quelli descrittivi, mentre i concetti di eucromatina e di eterocromatina possono includere una corrispondenza con precisi dati genetici, esistente solo in pochissime specie, in particolare in D. melanogaster. In altre parole, prima di descrivere i risultati delle indagini condotte coi metodi accennati, è opportuno ricordare che mentre in D. melanogaster e in poche altre specie eucromatina ed eterocromatina hanno a un tempo significato citologico e genetico, nell'uomo, per esempio, i due termini debbono essere impiegati solo in senso citologico: in tali casi, poiché si prescinde da implicazioni funzionali, i termini di eu- e di eterocromatina assumono un valore del tutto descrittivo se non addirittura provvisorio, in considerazione dei risultati di recentissime osservazioni che inducono a ritenere possibile in un prossimo futuro un radicale cambiamento ditale situazione.
Nell'uomo e in molti altri Mammiferi le varie tecniche, come per esempio la fluorescenza, consentono la distinzione strutturale tra cromosomi morfologicamente molto simili, e offrono pertanto la possibilità di procedere a più sicure diagnosi di anomalie cromosomiche.
Per valutare la rivoluzione suscitata dall'impiego di queste nuove tecniche nel riconoscimento dei cromosomi umani è sufficiente appena considerare che questi erano prima generalmente classificabili in gruppi contrassegnati da lettere alfabetiche (A, B, C, D, E, F, G), nel cui ambito le ulteriori classificazioni con numeri progressivi da 1 a 23 erano da molti ritenute incerte: ne risultava la difficoltà di distinguere certe paia di piccoli cromosomi, per esempio il 21° e il 22°, dei quali è ben noto il significato diagnostico nel mongolismo e nella leucemia mieloide cronica. Oggi invece, in base alla differente distribuzione della fluorescenza che consente di operare la distinzione fra 21° e 22° paio, si è in grado di correlare il mongolismo con un'anomalia del 21° consistente nell'eccesso di una dose, per esempio un terzo cromosoma di questo tipo o ‛trisomia 21°', e la leucemia mieloide cronica con la perdita di una porzione del 22°, detta ‛delezione Philadelphia', più recentemente interpretata come traslocazione.
L'introduzione di varie modifiche al metodo di Giemsa ha permesso di mettere in evidenza nei cromosomi la presenza di un certo numero di grosse strutture trasversali, dette bande (bandeggiatura o banding è il termine usato per indicare il fenomeno nel suo insieme), in posizione fissa, che vengono designate con lettere alfabetiche: le bande C sono quelle situate vicino al cinetocoro, le G sono intercalate lungo i bracci, le T sono in posizione terminale, le R sono responsabili di un banding rovesciato; con la lettera Q sono invece indicate le bande che appaiono nei preparati trattati con le tecniche della fluorescenza. Il riconoscimento della posizione di queste bande risulta di estrema importanza, in quanto consente di operare la distinzione fra paia di cromosomi e di individuare le variazioni di struttura di ongine mutazionale (perdite o delezioni, inversioni, traslocazioni, duplicazioni), acquisizioni che nell'uomo assumono un significato pratico fondamentale; molto incerto, invece, è il significato funzionale, cioè genetico, del banding. A parte il fatto che le tecniche impiegate non hanno dato finora risultati rigorosamente costanti e sovrapponibili e che una revisione critica non è stata ancora compiuta, va comunque ricordato che, come meglio vedremo poi, il significato stesso sia della fluorescenza sia della colorazione col Giemsa a livello molecolare non è ancora ben noto; si ritiene attualmente che solo le bande C siano quasi certamente eterocromatiche (v. fig. 13).
Nella drosofila, come si è detto, la distribuzione dell'eucromatina e dell'eterocromatina corrisponde a sicuri dati genetici. Conviene qui ricordare che nei cromosomi politenici l'eterocromatina appare come un elemento unitario, cioè come l'esigua massa del cromocentro nel quale confluiscono tutte le parti eterocromatiche che sono visibili con grande chiarezza nella metafase e che sono state indicate nel cap. 2, È b. Riassumento i dati di ordine genetico, si ricorda ancora che l'eterocromatina contiene geni che codificano RNA ribosomale; aggiungiamo che vi sono anche geni che agiscono in gruppo (poligeni), con effetti assai minuti, e geni che agiscono sulla maturazione degli spermi (i cosiddetti fertility genes). È probabile che le nostre conoscenze possano in futuro ampliarsi notevolmente, ma già ora è certo che il contenuto di geni mappabili (geni principali) dell'eterocromatina è minimo in confronto a quello dell'eucromatina, che ne contiene la quasi totalità. Ancora non sappiamo se questa situazione sia generalizzabile, e nemmeno il mais, ottimo materiale per ricerche genetiche e citologiche, finora è stato così ben esplorato a questo proposito come la drosofila.
Alla luce di questi dati si può ora esaminare la distribuzione della fluorescenza e della colorazione col Giemsa nei cromosomi mitotici di D. melanogaster. In questa specie, secondo la descrizione tradizionale basata sui risultati ottenuti con l'impiego di particolari coloranti come l'orceina, tutte le zone eterocromatiche sopra elencate appaiono compatte e fortemente colorabili; la colorazione con Giemsa dà risultati completamente sovrapponibili a questi. Non altrettanto può dirsi, invece, della distribuzione della fluorescenza: questa è infatti assai intensa a livello di blocchi del cromosoma Y, del cromosoma 4 e di zone adiacenti al cinetocoro del cromosoma 3; al contrario, il cromosoma X, telocentrico, è fluorescente solo in una breve porzione terminale vicino al cinetocoro, mentre per il resto della sua lunghezza la fluorescenza appare debolissima, al pari di quella rilevabile nelle zone adiacenti al cinetocoro del cromosoma 2 (G. Mosna e S. Faccio-Dolfini, 1974; v. fig. 14). Una prima semplice considerazione sulla differente intensità della fluorescenza mostrata dall'eterocromatina, intesa in senso genetico e citologico, potrebbe indurre a ritenere che la sua struttura molecolare non sia costante. Tuttavia, per quanto riguarda il problema del significato della fluorescenza a livello molecolare, occorre subito dire che nessuna spiegazione è finora apparsa del tutto convincente: l'ipotesi che la sua intensità dipenda dalla struttura del DNA - una forte fluorescenza essendo dovuta a una prevalenza nell'eterocromatina di basi citosina e guanina - è però difficilmente accettabile in Drosophila, in cui le diverse parti dell'eterocromatina del cromosoma X, pur caratterizzate da differenti fluorescenze, non sembrano differire funzionalmente. Per poter accettare tale ipotesi, infatti, si dovrebbe ammettere anche che funzioni simili siano correlate a una diversa struttura molecolare. In base alle dimostrazioni fornite da recenti ricerche, si ritiene oggi che le parti più fluorescenti siano particolarmente ricche di basi A e T o G e C, condizioni queste che caratterizzano due tipi di DNA, rispettivamente il satellite leggero e il satellite pesante. L'ipotesi alternativa, che attribuisce la fluorescenza agli istoni, non ha ancora ricevuto alcuna dimostrazione.
La colorabilità col Giemsa è generalmente interpretata come l'espressione di alta ripetitività nel DNA di sequenze di basi uguali, prevalentemente guanina-citosina: la dimostrazione migliore di ciò è stata fornita dai cromosomi mitotici del topo, studiati con le tecniche dell'ibridazione molecolare, nei quali è stato possibile accertare che nelle adiacenze del centromero è presente DNA ripetitivo e inoltre sono ben evidenti le caratteristiche dell'eterocromatina. Tuttavia sembra oggi ancora prematuro ammettere l'esistenza di un parallelismo generale fra eterocromatina geneticamente intesa, alta ripetitività di DNA e alta fluorescenza; d'altra parte, la stessa ipotesi di una corrispondenza rigorosa fra colorabilità col Giemsa e ripetitività del DNA è stata recentemente rimessa in questione. Comunque, è innegabile che in D. melanogaster tale corrispondenza appaia consistente, malgrado la comparsa, nell'ambito delle porzioni fortemente colorate col Giemsa, di un particolare banding al quale attualmente non è ancora possibile attribuire un significato genetico. La disparità fra i dati rilevabili rispettivamente in Drosophila e nei Mammiferi dovrebbe consigliare una notevole prudenza circa l'opportunità di interpretare come eterocromatina e indicazione di ripetitività del DNA tutte le zone positive alla colorazione col Giemsa e fortemente fluorescenti.
b) La ripartizione dei cromosomi nelle cellule figlie della mitosi
La mitosi tipica ripartisce l'assetto cromosomico, il cui DNA si è replicato durante la fase 5, in modo identico nelle cellule figlie. I meccanismi mediante i quali si attua tale processo sono: a) la separazione longitudinale di ogni cromosoma in parti uguali; b) il mantenimento del centromero indiviso fino alla fine della metafase; c) la disposizione dei cromosomi su un unico piano, la cosiddetta piastra equatoriale, in modo che un cromatidio sia rivolto verso un polo del fuso e l'altro verso il polo opposto; d) la divisione del centromero seguita dalla repulsione dei due cromatidi, che ormai equivalgono ciascuno a un cromosoma, e successiva migrazione fino al proprio polo del fuso. I fenomeni molecolari implicati nelle varie fasi di questo complicato processo sono ancora in gran parte sconosciuti; è certo comunque che la loro azione non può modificare l'aspetto genetico fondamentale, in quanto tutti i geni debbono essere ugualmente rappresentati nelle due cellule figlie.
Quanto si è sommariamente descritto avviene nella maggior parte delle cellule dei tessuti differenziati ove abbiano luogo mitosi, oltre che nelle cellule embrionali e negli abbozzi, sia animali sia vegetali. Le differenze nelle strutture del fuso sono abbastanza notevoli, ma non hanno rilevanza per i fenomeni cromosomici. Questi però, per cause varie, possono svolgersi in maniera diversa da quella descritta e conseguentemente possono realizzarsi assetti genici diversi nelle due cellule figlie. Le varianti verranno elencate a seconda del momento della mitosi nel quale si realizzano.
Un complesso di fenomeni, cui si dà il nome di mutazioni cromosomiche (v. fig. 15), che può aver luogo durante l'interfase, prima o dopo la replicazione del DNA, e manifestarsi nella successiva mitosi, è caratterizzato da un meccanismo comune, schematicamente rappresentabile come la rottura di un cromosoma seguita o no dal riattacco delle parti rotte, tale da dare origine a cromosomi dalla struttura anche notevolmente abnorme. Se l'energia richiesta dalla realizzazione di queste rotture è fornita da fattori esterni come raggi X, luce ultravioletta, metilsulfonato di etile ecc., le mutazioni che ne conseguono sono designate come indotte; spontanee, invece, sono definite quelle, in generale non differenti dalle prime, causate da fattori endogeni ignoti, e in parte anche dovute a errori di replica. Le rotture semplici, dette delezioni, causano la perdita di quella parte di cromosoma che viene a trovarsi priva di centromero: infatti, poiché questo non si riforma ex novo, i frammenti acentrici che non migrano ai poli non potranno far parte del patrimonio ereditario delle cellule figlie. Nelle delezioni che si trasmettono, i frammenti che si distaccano sono sempre di piccole dimensioni: distacchi di frammenti più grandi, infatti, sono causa di letalità cellulare. Se la rottura ha luogo prima della replica del DNA, i due cromatidi che successivamente si formano risultano identici anche per quanto riguarda la rottura, cosicché non vi sarà disparità nelle cellule figlie: è questo il caso della mutazione cromosomica. Invece, se la rottura ha luogo a replicazione avvenuta, è estremamente improbabile che si estenda ai due cromatidi o cada nello stesso punto di ciascuno di essi: ne risulta allora una differenza fra i due cromatidi del cromosoma, che si renderà evidente nella ripartizionè fra le due cellule figlie.
La rottura cromatidica o cromosomica può essere seguita da riattacco sicuramente operato da enzimi, anche se questi sono ancora pressoché sconosciuti; in questo caso si formerà una porzione acentrica, che andrà perduta (delezione), e un cromosoma con due centromeri, o dicentrico, che darà origine a una caratteristica figura, il cosiddetto ponte, e causerà un'ulteriore rottura all'anafase e una differente ripartizione nel patrimonio ereditario delle cellule figlie. Questo fenomeno si verifica spontaneamente e ripetutamente nelle cellule dell'endosperma di mais, dando luogo ai cosiddetti cicli di rottura-fusione-ponte. Quanto si è detto è appena sufficiente a dare un'idea delle numerose possibilità di mutazioni cromosomiche, con rotture multiple e cointeressamento di più cromosomi.
Le rotture cromatidiche e cromosomiche, d'altro canto, possono anche ‛guarire', cioè saldarsi nello stesso punto, cosicché la mutazione non è più riconoscibile; la frequenza con cui possono verificarsi queste ‛guarigioni' è in rapporto alle condizioni fisiologiche della cellula.
Quando i cromosomi si sono replicati, possono avvenire fenomeni di appaiamento e scambio fra cromatidi omologhi che, come vedremo, costituiscono la norma nella meiosi. Questo appaiamento, ben documentato in D. melanogaster, ha come risultato uno scambio di parti omologhe, cioè una ricombinazione. Si consideri ad esempio il caso di un genotipo
(yellow = corpo giallo; singed = setole contorte) cui corrisponde un fenotipo normale (++), in quanto i caratteri yellow e singed sono recessivi: a seguito di processi di appaiamento e successiva ricombinazione fra singed e il centromero si formeranno cellule yellow e cellule singed (essendo le prime
e le seconde
la cui successiva moltiplicazione darà luogo alla comparsa sul corpo dell'insetto di due zone adiacenti, dette macchie gemelle, l'una gialla con setole normali e l'altra bruno-chiaro (il colore tipico della drosofila selvatica) con setole contorte (v. fig. 16). Il fenomeno dell'appaiamento è noto nelle cellule dei miceli di Ascomiceti, ove surroga parzialmente l'effetto genetico della meiosi e della fecondazione che mancano in varie specie di questo gruppo. Quando i cromosomi sono ordinati nella piastra equatoriale può ancora accadere che il centromero di qualcuno di essi non si divida: si realizza in tal caso la non-disgiunzione, per effetto della quale a un polo, e quindi a una cellula figlia, migrerà indiviso un cromosoma, di cui risulterà ovviamente priva l'altra cellula figlia. È questo il fenomeno che dà luogo nell'uomo alla trisomia del 21° paio, a cui corrisponde la totale assenza dei cromosomi 21, che determinano rispettivamente la condizione di cellule dotate di 47 cromosomi e quella, letale, di cellule con 45 cromosomi, in luogo del normale numero di 46.
La mitosi più drasticamente alterata è quella nella quale il fuso è fornito di più poli: in questi casi, frequenti in cellule patologiche come ad esempio le neoplastiche, l'assetto cromosomico viene ripartito in modo estremamente abnorme e generalmente letale.
Per concludere, si può affermare che, se è vero che la mitosi consiste normalmente in una distribuzione in parti uguali del patrimonio ereditario, esistono tuttavia non pochi casi nei quali le cellule figlie risultano dotate di assetti cromosomici differenti.
c) L'endomitosi e la C-mitosi
Diversi tipi di cellule, quali nei Ditteri le cellule somatiche destinate a divenire politeniche, le cellule somatiche di certi Rincoti, alcune cellule del fegato dei Mammiferi ecc., si accrescono mediante il raddoppiamento dell'assetto cromosomico: questo si realizza per mezzo della cosiddetta mitosi interna o endomitosi, consistente tipicamente in una replicazione del DNA non seguita da divisione mitotica. L'endomitosi, scarsamente visibile nei nuclei politenici, è comunque caratterizzata da un certa spiralizzazione dei cromosomi e dalla divisione dei centromeri. In questi casi pertanto si raddoppia il numero dei cromosomi e, se il fenomeno si ripete, compaiono cellule con 4n, 8n, 16n ecc. cromosomi, dette cellule poliploidi.
Il raddoppio del numero dei cromosomi può essere provocato sperimentalmente mediante l'impiego di sostanze diverse, fra le quali la più nota e più usata è la colchicina, la cui azione si esplica essenzialmente durante la metafase sulle fibre del fuso, che ne risulta praticamente disfatto. I cromosomi in mitosi, già divisi, si riorganizzano allora entro una nuova membrana nucleare (nucleo di restituzione), e il loro numero risulta quindi raddoppiato.
Dal momento che l'induzione di poliploidi è un mezzo sempre più sfruttato per migliorare molti prodotti agricoli, il cui valore è in relazione diretta alla mole delle foglie, del frutto ecc., ed essendo la coichicina ben tollerata dalle cellule vegetali, a differenza di quelle animali, si comprende facilmente come la C-mitosi abbia assunto una notevole importanza pratica nel miglioramento genetico vegetale (v. fig. 17).
4. I cromosomi nella meiosi
La meiosi è quel processo durante il quale avviene la riduzione a metà dell'assetto cromosomico e si verifica quindi il passaggio dalla condizione diploide, 2n, a quella aploide, n. Il fine di questo processo è quello di mantenere costante il numero dei cromosomi nelle forme sessuate, nelle quali altrimenti in ogni fecondazione il patrimonio ereditario si raddoppierebbe. La meiosi è stata definita (C. D. Darlington, 1938) una successione di due divisioni del nucleo e delle cellule, durante le quali i cromosomi si dividono una sola volta: da queste hanno quindi origine 4 nuclei, ognuno dei quali con un numero di cromosomi pari alla metà di quello del nucleo primitivo. La meiosi è strettamente connessa con la riproduzione sessuale e presenta una notevole uniformità nella sequenza delle sue fasi proprio nelle specie diploidi e bisessuate, che sono le più note dal punto di vista genetico; è invalsa perciò l'abitudine di considerare il tipo di meiosi in esse riscontrabile come l'unico esistente. È questa, tuttavia, una semplificazione alquanto scolastica che non prende in considerazione i numerosi casi, assai più frequenti negli animali che nelle piante, nei quali la meiosi si attua in modo diverso da quello più generalmente riscontrabile.
I fenomeni cromosomici della meiosi non sono esclusivi di un solo tipo di cellula e possono verificarsi in un qualunque momento del ciclo riproduttivo, costante però in ogni specie. Negli animali, infatti, la meiosi si svolge durante la gametogenesi (spermatogenesi e ovogenesi) e viene pertanto detta gametica o terminale; nelle piante superiori è invece sporica o intermedia, cioè ha luogo in cellule dello sporofito che daranno origine, a meiosi ultimata, alla spora, dalla quale poi deriverà il gametofito; si realizza così un'alternanza fra generazioni aploidi e diploidi. In molti Funghi e in molte Alghe, infine, la meiosi è definita zigotica o iniziale in quanto ha luogo immediatamente dopo la formazione dello zigote, che per breve tempo risulta pertanto diploide prima di divenire aploide.
Dal momento che in questa trattazione i fenomeni cromosomici vengono esaminati dal punto di vista dell'interpretazione genetica, tralasceremo in linea di massima di parlare dei processi che riguardano il citoplasma. La descrizione generale della meiosi sarà riferita ai soli autosomi, che sono i cromosomi comuni ai due sessi; in seguito parleremo anche degli eterocromosomi o cromosomi sessuali, quelli cioè che - quando sono individuabili - differiscono nei due sessi.
L'analisi dei complessi fenomeni che si svolgono durante la meiosi ha consentito di individuare varie fasi, che verranno descritte più in dettaglio, la cui successione può essere esemplificata nel seguente schema (v. figg. 18 e 19): 1) profase, a sua volta distinta in: a) leptotene, in cui i cromosomi divengono individuabili in forma di filamenti; b) zigotene, in cui inizia l'appaiamento dei cromosomi; c) pachitene, in cui i cromosomi appaiati si ispessiscono e si duplicano; d) diplotene, in cui i cromosomi bipartiti di ogni paio si allontanano l'uno dall'altro; e) diacinesi, in cui i cromosomi sono contratti e mostrano i punti di incrocio spostati verso l'estremità; 2) metafase I, caratterizzata dalla posizione dei bivalenti diacinetici in piastra equatoriale sul fuso; 3) anafase I, in cui i centromeri si allontanano verso i poli opposti traendo ognuno un elemento del bivalente; 4) telofase I, in cui avviene la costituzione del nucleo figlio, con i centromeri ancora indivisi; 5) metafase II, in cui gli elementi di ciascun nucleo aploide si dispongono in piastra equatoriale; 6) anafase II, caratterizzata dallo spostamento dei centromeri ai poli opposti del fuso; 7) telofase II, caratterizzata dalla ricostituzione della membrana in ciascuno dei quattro nuclei figli, aploidi, che entrano in quiescenza.
a) L'appaiamento
La meiosi comincia con una profase mitotica particolare, durante la quale ha luogo l'appaiamento fra cromosomi omologhi. Questo talvolta avviene solo in un sesso, ad esempio in D. melanogaster solo nel sesso femminile, cioè nell'ovogenesi, il cui studio, peraltro, presenta notevoli difficoltà. Lo schema della meiosi ‛normale' in entrambi i sessi, perciò, è più facilmente riconoscibile nel mais e negli Ortotteri. Nel primo stadio, detto leptotene, i cromosomi divengono ben visibili, in forma di filamenti molto sottili e assai sinuosi dei quali riesce assai arduo seguire il percorso. Il vero problema prospettato dallo studio di questo primo stadio è rappresentato dalla struttura dei singoli filamenti che, a differenza di quelli osservabili nelle mitosi somatiche, all'osservazione al microscopio ottico appaiono semplici, cioè non costituiti da due cromatidi. Si è quindi convenuto di interpretare questi sottili filamenti come ancora non replicati, anche se in alcuni tratti ne appare chiaramente la struttura duplice. Si potrebbe pertanto concludere che la replicazione del DNA cominci nello stadio leptotene, per proseguire poi, come vedremo, fino al pachitene. Nei filamenti leptotenici sono presenti, in forma di granuli distaccati uno dall'altro, i cromomeri, che si manterranno visibili anche negli stadi successivi di zigotene e pachitene. L'eterocromatina costitutiva è già ben individuata come un blocco compatto. Nello stadio zigotene ha inizio l'appaiamento dei cromosomi, prima per brevi tratti e poi per tutta la loro lunghezza, e diviene completo nel successivo stadio pachitene; a questo punto i cromosomi omologhi, cioè uno di origine paterna e l'altro di origine materna, sono così strettamente appaiati da dare origine a corpi apparentemente indivisi, detti bivalenti, il cui numero, pari a metà di quello originario dei cromosomi, caratterizza il nuovo assetto aploide della cellula (v. fig. 20).
Allo stadio pachitene la struttura di ogni bivalente è però tetrapartita, perché ogni cromosoma si è ormai duplicato. Esistono di ciò prove genetiche irrefutabili, dedotte dalla ricombinazione (crossing-over), o scambio omologo fra cromatidi omologhi, che avviene appunto durante il pachitene (v. fig. 21). Infatti, senza appaiamento, almeno rapido e fugace, non vi può essere scambio fra cromosomi omologhi; i risultati dello scambio debbono essere riconoscibili quando i filamenti cromosomici del pachitene, dopo essersi disgiunti, si ricostituiscono in nuclei di cellule separate, la cui analisi sarà poi resa possibile dallo studio dei caratteri e delle manifestazioni dei loro discendenti, siano questi miceli di Funghi o sporofiti di piante superiori o, negli animali, individui provenienti da zigoti. Tutti gli esperimenti eseguiti sulle specie più diverse provano che da una cellula meiotica maschile, detta anche meiocita, derivano quattro cellule aploidi mature. Quando è possibile riconoscere, come in Neurospora crassa, i singoli prodotti della meiosi, mediante la cosiddetta analisi delle tetradi, si osserva che, anche dove è avvenuta la ricombinazione, due cellule su quattro non sono ricombinate. Esemplificando: se due caratteri diversi noti, A e B (detti marcatori), di un cromosoma sono presenti in una cellula fino alla meiosi in condizione eterozigote
le cellule derivate saranno quattro (salvo la possibilità esclusiva dei Funghi, che qui non interessa ricordare, di un ulteriore raddoppiamento per una divisione) con le seguenti costituzioni genetiche o genotipi: A B e a b (senza ricombinazione), A b e a B (ricombinanti). Queste osservazioni dimostrano chiaramente che la ricombinazione, come del resto si verifica nelle cellule somatiche, avviene fra cromatidi e non fra cromosomi e dunque che ogni filamento del pachitene, che deriva dall'appaiamento di due cromosomi omologhi, al momento della ricombinazione o scambio consta in effetti di quattro elementi: questi vengono denominati cromatidi omologhi in quanto fanno parte di due cromosomi omologhi, mentre si designano come fratelli i due cromatidi di uno stesso cromosoma (v. fig. 18).
Il fenomeno della ricombinazione o scambio consiste dunque nella formazione di combinazioni nuove e reciproche fra geni situati sullo stesso cromosoma: esso avviene più o meno uniformemente lungo il cromosoma e la sua frequenza, in riferimento a due determinati geni, è direttamente proporzionale alla distanza che intercorre fra i geni stessi.
Questa rappresentazione formale del fenomeno mostra tuttavia una fondamentale carenza, in quanto non spiega affatto la causa e il meccanismo dell'appaiamento e consente solo alcune ipotesi circa quelli della ricombinazione.
L'appaiamento è un fenomeno estremamente specifico per ogni punto dei cromosomi omologhi, vale a dire che l'accostamento è rigoroso fra loci geneticamente omologhi; poiché l'unità visibile più vicina al gene è il granulo di cromatina denominato cromomero, si può anche dire che l'appaiamento consiste in un accostamento fra cromomeri omologhi. Dimostrazioni in tal senso possono essere fornite sia dallo studio di materiale, come gli Ortotteri o il mais, in cui i cromomeri siano particolarmente ben visibili, sia dall'osservazione di quei casi nei quali la struttura dei due cromosomi omologhi non è identica, perché uno dei due ha subito una mutazione che altera l'ordine dei cromomeri. Si conoscono varie mutazioni - la delezione (mancanza di un segmento in uno dei due cromosomi omologhi), l'inversione (rotazione di 180° di un segmento di un cromosoma), la traslocazione reciproca (scambio di posto di due segmenti appartenenti allo stesso cromosoma o a cromosomi diversi), la duplicazione (presenza di un segmento supplementare in un cromosoma), la frammentazione (i frammenti cromosomici privi di centromero vengono riassorbiti nel citoplasma), la fusione (unione di due cromosomi) - alcune delle quali possono essere schematizzate come segue:
È evidente che il semplice accostamento non è sufficiente a consentire l'appaiamento di due cromosomi diversamente conformati, onde è necessaria la formazione di anelli, occhielli ecc. che caratterizzano il pachitene (v. fig. 22). Tanto per dare un esempio, l'appaiamento fra un cromosoma normale e uno deleto assumerà la seguente configurazione:
In questi ultimi anni l'impiego della microscopia elettronica ha permesso di individuare nella fessura interposta fra i cromosomi in appaiamento una formazione, cui è stato dato il nome di ‛complesso sinaptinemico' (synaptinemic complex), consistente in due cilindri paralleli nei quali è possibile distinguere una zona esterna e una interna. Gli stretti rapporti esistenti tra complesso sinaptinemico e appaiamento, dimostrati dal fatto che soltanto in coincidenza di tale fenomeno è possibile osservare la suddetta formazione, non consentono tuttavia di individuare i fattori responsabili dell'appaiamento stesso. E comunque certo che questo fenomeno si svolge sotto il controllo genetico, dal momento che nella drosofila e nel mais sono state dimostrate alcune mutazioni per effetto delle quali esso non ha luogo.
L'appaiamento meiotico trova riscontro nell'appaiamento somatico, detto più precisamente sinapsi somatica, già descritto a proposito dei cromosomi politenici dei Ditteri, con un perfetto parallelismo nelle configurazioni fra cr0mosomi a struttura deleta, invertita ecc.
b) La ricombinazione (crossing-over).
Durante lo stadio pachitene, come già si è detto, ha luogo la ricombinazione, mediante un meccanismo di doppia rottura a uguale livello con riattacco scambiato, secondo lo schema:
Questo meccanismo, designato con il termine di crossing-over, pur se recentemente sottoposto a varie critiche, resta in definitiva il modello più attendibile, malgrado sia esclusivamente meccanico e privo di quella corrispondenza a livello molecolare che è oggi un'esigenza assoluta nella spiegazione di ogni fenomeno biologico. Ne deriva una comprensibile situazione di lacune e di incertezze, la quale ha comunque reso possibile la formulazione di due ipotesi (v. fig. 23) che saranno qui brevemente riferite. Loro base comune è l'ammissione dell'esistenza in ogni cromatidio di una doppia elica di DNA e dell'intervento di un enzima che, ad appaiamento avvenuto, rompe allo stesso punto due eliche appartenenti a cromatidi omologhi; a ciò può seguire, se vi è riattacco (cioè ricombinazione, sempre per effetto di azioni enzimatiche) fra eliche di DNA da cromatidi omologhi, la formazione di un'elica di DNA ibrido. Secondo la prima ipotesi, o modello di Whitehouse, le eliche che si rompono avrebbero polarità opposta (v. acidi nucleici), cioè i terminali di ciascuna elica che si ricombina sarebbero rispettivamente ′3-′5 e 5′-3′; la seconda ipotesi, o modello di Holliday, suppone invece che le eliche che si scambiano abbiano la stessa polarità. Conseguentemente, nel primo caso, a differenza del secondo, sarebbe necessaria una nuova sintesi di DNA a ricombinazione avvenuta, cioè nel pachitene, sintesi che in certe condizioni, come già si è detto, è stata effettivamente dimostrata; sicché anche se è ancora presto per accordare la preferenza all'una o all'altra delle due ipotesi, l'accettazione di quella di Whitehouse non sembra incontrare serie difficoltà.
Buona parte di questa impostazione teorica è desunta dall'osservazione dei fenomeni di ricombinazione che hanno luogo nei procarioti, regolati da enzimi, molti dei quali sono stati isolati e studiati. Negli eucarioti l'azione enzimatica è soprattutto inferita dall'esistenza di mutanti deficienti per l'appaiamento (come nel caso del mais), nei quali sicuramente vi è carenza di un enzima specifico. Tale ricostruzione della ricombinazione rappresenta dunque un'estensione a livello molecolare del crossing-over.
Nell'esposizione che si è fatta si è trascurata la possibilità di ricombinazione fra cromatidi fratelli, la cui esistenza può però essere dimostrata indirettamente con metodi citogenetici piuttosto complicati e, secondo studi molto recenti, con osservazioni citologiche dirette.
c) I chiasmi
La ricombinazione è un fenomeno obbligatorio nella maggior parte delle specie, sebbene spesso limitata, come già si è detto, a un solo sesso; essa può essere localizzata in determinati tratti lungo l'asse del cromosoma, come nell'ortottero Mecosthetus, oppure casualmente distribuita lungo tutto il cromosoma, come in D. melanogaster e nel mais.
In alcuni casi sembra che una ricombinazione avvenuta in un braccio del cromosoma inibisca il realizzarsi di un'altra nell'altro braccio (interferenza oltre il centromero).
Quando il pachitene volge alla fine l'appaiamento diventa meno intenso: compaiono allora chiaramente i quattro cromatidi, che a due a due, cioè fra omologhi, mostrano caratteristici incrociamenti a X, detti chiasmi. Lo stadio in cui questi appaiono chiaramente visibili è il diplotene. E dimostrato che ogni chiasma corrisponde a un'avvenuta ricombinazione: in altre parole i chiasmi rappresentano l'espressione morfologica del crossing-over che ha avuto luogo nello stadio pachitene. Pertanto, conoscendo il numero dei chiasmi, è possibile stimare la frequenza con cui è avvenuta la ricombinazione; va tuttavia ricordato che alla fine del diplotene i chiasmi scompaiono per un fenomeno detto ‛terminalizzazione'. Riprendiamo ora in considerazione un aspetto della meiosi cui si è prima accennato, cioè il destino cui andranno incontro le cellule da essa prodotte che, rappresentando il risultato di due divisioni cellulari, devono essere quattro. Negli organismi nei quali non si ha sessualità morfologicamente espressa, le quattro cellule finali prenderanno tutte parte o alla fecondazione o alla formazione di sistemi pluricellulari aploidi, come nel caso dei miceli dei Funghi. Invece, nelle specie in cui esiste la differenziazione sessuale, come gli animali e le piante superiori, le quattro cellule saranno tutte ugualmente impiegate nel processo fecondativo (animali) o nella produzione del gametofito (piante) solo nel sesso maschile, mentre nei processi di ovogenesi e di megasporogenesi solo una delle 4 cellule, la cui scelta è casuale, evolverà a formare la cellula uovo. Tale risultato della meiosi nel sesso femminile, nelle piante è in relazione a una semplice degenerazione di tre su quattro cellule; negli animali, invece, sia dalla prima sia dalla seconda divisione hanno origine due prodotti disuguali per quello che concerne il citoplasma e alla fine si formeranno una sola grossa cellula uovo e due piccolissimi globuli polari, rispettivamente I e II, questi ultimi destinati a degenerare. Siccome il fenomeno è, come si è detto, casuale, l'eliminazione di 3 su 4 cellule significa solo una riduzione a 1/4 delle cellule utilizzate nella riproduzione, ma non un'eliminazione sistematica di genotipi: pertanto se una primitiva cellula femminile (ovogonio) è eterozigote
le probabilità che nel globulo polare vada A o a sono le stesse e alla fine dell'ovogenesi le uova mature saranno 1/2 A e 1/2 a, in analogia a quanto si verifica negli spermatozoi (v. fig. 24).
Infine, un altro problema inerente alla meiosi, del quale abbiamo in precedenza prospettato i termini, è la possibilità di individuare il momento in cui i cromosomi paterni si separano da quelli materni. In relazione ai due classici termini di divisione riduzionale e divisione equazionale, usati per indicare rispettivamente la divisione che determina la separazione dei cromatidi paterni da quelli materni e quella nella quale i cromatidi fratelli si separano fra loro, occorre tener conto dell'intervento o meno della ricombinazione: se questa ha luogo, le divisioni non possono risultare totalmente riduzionali o equazionali; in caso contrario invece - come si verifica nel maschio di D. melanogaster - risulta riduzionale la prima divisione ed equazionale la seconda. Appare infatti chiaro che in ogni cromosoma per effetto della ricombinazione la prima divisione è riduzionale solo in corrispondenza della zona del centromero, in quanto tale formazione si divide nella seconda divisione, la quale a sua volta è riduzionale a livello di tutti i tratti successivi a un crossing-over (v. fig. 25).
d) I cromosomi sessuali
Il numero e la struttura dei cromosomi sessuali, o eterocromosomi, variano molto nelle diverse specie e talvolta anche nei gruppi di specie congeneri; tuttavia le trattazioni elementari si limitano di solito a considerare il caso in cui gli eterocromosomi sono due e uguali in un sesso (XX o ZZ) e due e differenti oppure uno solo nell'altro (XY o ZW o rispettivamente X o Z; v. fig. 26). Quest'ultimo è il caso tipico degli Uccelli, dei Lepidotteri, di molti Crostacei e dell'anfibio urodelo axolotl.
La condizione più frequente è rappresentata dal sesso XX, detto anche omogametico, nella femmina e da quello XY o X, detto eterogametico, nel maschio: in questi casi, che potremmo dire classici, il comportamento dei due cromosomi uguali X o Z non differisce da quello degli altri cromosomi (autosomi), mentre i due cromosomi X e Y, essendo diversi quasi sempre per forma, struttura e dimensioni, danno luogo durante l'appaiamento a figure caratteristiche (v. fig. 27). Nell'uomo e in molti mammiferi non solo l'Y è assai più piccolo dell'X, così che la parte che si può appaiare è breve, ma manca anche l'omologia per tutta la lunghezza appaiabile. Se esistesse appaiamento vero e proprio fra X e Y in D. melanogaster (come alcuni autori hanno affermato, malgrado la notevole difficoltà tecnica presentata dallo studio della meiosi in questa specie) esso si dovrebbe limitare al tratto eterocromatico dell'X, corrispondente a metà circa dell'intera lunghezza, mentre l'Y è tutto eterocromatico. La condizione di un solo X in un sesso, frequente in molti insetti (Rincoti e Ortotteri; v. fig. 26), è caratterizzata dal risultato delle due divisioni: infatti, poiché durante l'appaiamento l'unico X non può prendere parte al fenomeno, l'eterocromosoma sarà presente in una sola delle due cellule figlie dopo la prima divisione, mentre nella seconda avrà luogo la ripartizione dei due cromatidi dell'X in seguito alla quale due dei quattro spermatozoi (il fenomeno è tipico della spermatogenesi) risulteranno forniti dell'eterocromosoma e due ne saranno privi. Conseguenza della meiosi è pertanto il mantenimento della differenza di un'unità fra maschio e femmina, perché nella fecondazione gli spermatozoi provvisti dell'X danno origine alla formula XX (femmina) e quelli che ne sono privi alla formula X (maschio). Questi brevi cenni sono sufficienti a mettere in evidenza sia l'importanza del ruolo svolto dai cromosomi sessuali nella determinazione del sesso, sia la difficoltà di valutare esattamente l'andamento del fenomeno. Una logica interpretazione del caso in cui un solo X caratterizza il sesso maschile e due danno luogo a quello femminile consente di ammettere che il sesso dipende da uno sbilancio tipico fra geni contenuti negli autosomi e geni contenuti negli eterocromosomi: infatti, considerando i fattori autosomici come favorenti la mascolinità, appare chiaro che occorrono due eterocromosomi, cioè due X, perché si determini lo sbilancio necessario alla manifestazione della femminilità. Questa situazione per la quale X è responsabile della femminilità si verifica in D. melanogaster (non però in altre specie dello stesso genere), in cui il cromosoma Y non prende parte alla determinazione del sesso, ma solo collabora nel differenziamento degli spermatozoi: sono infatti maschi gli individui X e quelli XY, i primi però sono sterili. Invece nell'uomo, nel topo e probabilmente in molti altri mammiferi il cromosoma Y è portatore dei geni della mascolinità e l'X di quelli della femminilità: è noto infatti che in queste specie gli individui XY sono maschi e quelli X sono femmine, sia pure sterili.
Il comportamento dei cromosomi sessuali X e Y nelle due divisioni meiotiche non è sempre uguale a quello degli autosomi: si conoscono molti casi in cui gli eterocromosomi si dividono equazionalmente nella prima divisione, così che ogni cellula figlia riceve un cromatidio dell'X e uno dell'Y, e riduzionalmente nella seconda, durante l'anafase della quale a un polo andrà il cromatidio dell'X e all'altro il cromatidio dell'Y (v. fig. 25). La frequenza con cui si verificano i diversi tipi di divisione è differente nelle varie specie: per esempio la prima divisione è riduzionale nel ratto nel 9o% dei casi, nel criceto dorato nell'81% circa. Per brevità non teniamo conto della ricombinazione, che avviene comunque, come di norma, nel segmento omologo regolarmente appaiatosi allo stadio pachitene.
Quella fin qui esposta è la situazione tipica dei casi più comuni, ritenuti classici: non è tuttavia possibile, a meno di non cadere in un eccesso di semplificazione, non tener conto dell'esistenza di altre condizioni, invero meglio conosciute dal punto di vista citologico che non da quello genetico, contrassegnate da formule eterocromosomiche differenti da quelle precedentemente descritte. Ad esempio, si conoscono varie specie di Rincoti con numeri elevati di X e di Y, i quali non vanno neppure incontro a un vero appaiamento, ma si avvicinano semplicemente per separarsi nella prima divisione; nel genere Drosophila, è conosciuto il caso di D. miranda, in cui le femmine hanno due coppie di X diversi (X1X1X2X2) e i maschi un solo X per sorte (X1X2Y). Va infine ricordata l'esistenza di specie nelle quali i cromosomi del sesso sono morfologicamente poco o nulla distinguibili dagli autosomi, pur essendo sempre responsabili della determinazione del sesso.
e) Anomalie strutturali e di distribuzione
Lo schema della meiosi che abbiamo descritto può andare incontro a diverse varianti, dovute ad anomalie della struttura o a errori di distribuzione dei cromosomi.
Le anomalie strutturali più importanti sono rappresentate dalle inversioni allo stato eterozigote. Nel corso dell'appaiamento zigotenico-pachitenico si formano le configurazioni particolari che abbiamo precedentemente descritto; orbene, se avviene una ricombinazione entro l'inversione, ne derivano cromatidi ricombinanti letali, onde in pratica è come se le inversioni eterozigoti inibissero la ricombinazione. Le traslocazioni reciproche possono dar luogo a combinazioni letali, se ai poli della prima divisione vanno porzioni cromosomiche ripetute o risultano carenze. L'errore di distribuzione più conosciuto è la non disgiunzione, consistente nella mancata separazione dei coniuganti alla prima divisione e, eventualmente, nella mancata divisione dei centromeri alla seconda. In D. melanogaster il fenomeno della non disgiunzione degli X è più o meno frequente in alcuni ceppi e determina la formazione di uova XX e uova senza X; la correzione conseguente alla fecondazione dà luogo alle seguenti combinazioni:
Le conseguenze di tale fenomeno si manifestano nella trasmissione di quei geni che sono collocati nel cromosoma X e che si dicono legati al sesso: prescindendo dalla dettagliata esposizione dello schema di questa trasmissione, basterà qui ricordare che nel caso della non disgiunzione gli individui maschi X derivati da un uovo senza X ricevono tale cromosoma dallo spermatozoo, mentre normalmente i maschi (XY) ricevono l'X dalla madre. Nell'uomo è classico il caso di non disgiunzione autosomica del 21° paio cromosomico, che dà luogo alla sindrome di Down o mongolismo: quando in una donna si forma un uovo con due cromosomi di questo paio anziché con uno solo come di norma, la sua successiva fecondazione da parte di uno spermatozoo normale dà origine a uno zigote con 47 cromosomi, più precisamente a un fenotipo caratterizzato da tre elementi 21.
Una femmina mongoloide a sua volta potrà produrre uova con due o con un solo cromosoma 21, che daranno origine, rispettivamente, le prime a individui mongoloidi, le seconde a individui normali. La presenza di un cromosoma soprannumerario, molto diffusa nelle piante nelle quali caratterizza frequenti varianti nell'ambito di molte specie, è detta trisomia.
Abbiamo illustrato il processo meiotico quale senza dubbio si svolge nella maggior parte degli eucarioti, anche se fra le specie più studiate prevalgono quelle che si prestano bene allo studio citologico ma sono poco note geneticamente: è questo il caso, fra gli Insetti, degli Ortotteri e dell'ascaride del cavallo (Parascaris equorum) e, fra le piante, di Fritillaria, Tradescantia e Trillium. Esistono tuttavia anche situazioni opposte: i lieviti, ad esempio, geneticamente analizzati con grande precisione, sono tuttora indecifrabili dal punto di vista citologico, almeno con le tecniche attualmente disponibili; la stessa D. melanogaster, l'organismo animale geneticamente più noto, non è un buon materiale citologico e la sua ovogenesi è praticamente ancora inesplorata. Questa specie presenta inoltre una meiosi che differisce non poco dalla descrizione generale in precedenza fornita: infatti, dopo l'appaiamento dello stadio pachitene (durante il quale soltanto nella femmina ha luogo la ricombinazione, mentre nel maschio probabilmente non avviene neppure un vero appaiamento), nella diacinesi si svolge un secondo appaiamento anche fra cromosomi non omologhi cui si dà il nome di ‛secondario' o ‛distributivo', in quanto è responsabile della distribuzione dei cromosomi alla prima divisione. La dimostrazione di questo fenomeno è stata resa possibile dall'impiego di marcatori in genotipi caratterizzati da aberrazioni numeriche: ad esempio la formazione, in una femmina con un Y soprannumerario (cioè XXY) e un solo cromosoma 4, di circa un 8o% di uova o con il 4 o con l'Y si può spiegare solo con un appaiamento distributivo fra questi due cromosomi, il quale può a sua volta essere spiegato con l'attrazione entro l'eterocromatina, in quanto come si è detto l'Y è tutto eterocromatico e il cromosoma 4 è quasi tutto eterocromatico. Il mais è l'unica specie nella quale la validità dello schema generale della meiosi può essere dimostrata sia citologicamente sia geneticamente. In molte specie, invece, per ragioni diverse la meiosi segue obbligatoriamente, e non solo in casi particolari, vie molto peculiari: è questo un aspetto del processo solitamente trascurato dai manuali scolastici, ma del quale si vuole qui dare un'informazione sufficiente a dimostrare come la riduzione del numero dei cromosomi possa venir conseguita con una notevole varietà di meccanismi o, nelle non poche specie monosessuate, in quelle cioè nelle quali manca il sesso maschile, non realizzarsi affatto o realizzarsi solo parzialmente.
La forma di meiosi particolare più simile allo schema generale è quella che si svolge nelle piante poliploidi; con il termine poliploidia si indica una condizione in cui il numero dei cromosomi è superiore a quello normale diploide o 2n, ed è identificabile con il prodotto di n per un numero intero (per esempio, nella triploidia il numero dei cromosomi è 3n). Nel genere Trillium (Liliiflore) la specie T. praecox presenta una grande varietà di tipi di appaiamento, dovuta al fatto che vi sono tre elementi omologhi anziché due, e l'accostamento avviene peraltro sempre fra due elementi omologhi in tratti diversi: il cromosoma che chiameremo 1 si appaia infatti per un tratto con quello che chiameremo 2, e questo per un altro tratto si appaia con quello che chiameremo 3; si viene così a realizzare una situazione non molto differente dall'appaiamento vero e proprio. I corpi che ne risultano, formati da tre elementi omologhi, assumono forme caratteristiche e sono detti trivalenti (polivalenti, in generale, in condizioni di poliploidia diverse dalla triploidia). In alcune specie, come appunto in T. praecox, la frequenza dei polivalenti varia molto anche da cellula a cellula; in altre, come ad esempio nella congenere T. lanatum, è invece uniforme; in altre ancora, infine, non vi è formazione alcuna di polivalenti e la meiosi non differi sce da quella tipica dei diploidi. I polivalenti, specie se dispari (trivalenti, pentavalenti ecc.), tendono a distribuirsi in modo non simmetrico alla prima divisione: per esempio nella triploidia si ha la formazione di un monovalente e di un bivalente, onde la meiosi di un trivalente dà chiaramente luogo a instabilità genotipica. Inoltre, l'appaiamento è spesso largamente incompleto e la ricombinazione ne risulta limitata. Nella fig. 28 è illustrato lo schema di una meiosi con quadrivalenti.
Una situazione assai complessa è poi quella nota come allopoliploidia, in cui la poliploidia ha origine da una precedente ibridazione interspecifica (alla poliploidia entro la specie si dà invece il nome di autopoliploidia): un esempio molto importante è offerto dal grano (Triticum vulgare), esaploide con 42 cromosomi derivati dalla successiva ibridazione fra T. monococcum (genoma A), Aegilops speltoides (genoma B) e Aegilops squarrosa (genoma D). Nella meiosi di T. vulgare si formano solo bivalenti, cioè A con A, B con B e D con D, essendo ciascun genoma duplicato (3×2=6, onde l'esaploidia).
Per finire, noteremo che la poliploidia costituisce il caso limite della presenza di cromosomi soprannumerari, di cui la trisomia già considerata è l'altro estremo.
Alcune specie poi (animali, questa volta, e Insetti Imenotteri in particolare) sono caratterizzate da una singolare determinazione del sesso per cui le uova fecondate danno luogo a femmine diploidi, le non fecondate, che si sviluppano con corredo aploide, a maschi: in tali casi è facilmente prevedibile che la maturazione delle cellule germinali maschili non comporterà una riduzione cromosomica e perciò neppure una vera meiosi.
Un secondo gruppo di meiosi particolari è quello, piuttosto eterogeneo, delle specie o forme che si riproducono per sviluppo della cellula uovo non fecondata e le cui femmine sono dette partenogenetiche negli animali e apomittiche o partenogenetiche (a seconda dei casi) nelle piante (v. riproduzione). Molteplici sono negli animali, soprattutto negli Artropodi, le soluzioni al problema generale rappresentato dall'esigenza di evitare la riduzione del numero cromosomico, onde evitare, per la mancanza della fecondazione, il dimezzamento del patrimonio ereditario a ogni generazione: in alcune specie del genere Solenobia (lepidottero psichide) ha luogo una meiosi completa compensata alla fine, cioè nelle segmentazioni dell'uovo aploide, da una fusione fra i due nuclei di due cellule, cosicché gli individui sono obbligatoriamente omozigoti; nel crostaceo Artemia salina possono realizzarsi diverse condizioni, dalla meiosi limitata alla sola prima divisione fino alla completa assenza di meiosi; in certe piante (Erigeron) si ha pure una sostanziale mancanza di meiosi, mentre in Taraxacum a un fugace appaiamento seguono deconiugazione e una sola mitosi; ancora in altre piante (Hieracium) l'embrione deriva da una cellula diploide e non dalla megaspora. Come si vede, in sostanza, tutti questi meccanismi sono impiegati per conservare il numero cromosomico e ridurre o annullare totalmente la ricombinazione, tranne che nel caso di Solenobia, in cui appunto la meiosi, completa e normale, è compensata dalla fusione nucleare.
f) Appaiamento incompleto ed eliminazione di cromosomi
Un terzo gruppo di meiosi particolari, differenti in modo ancora più radicale dallo schema generale, si riscontra quasi esclusivamente nei Ditteri e nei Coccidi (Rincoti), con una distribuzione assai irregolare dal punto di vista sistematico; in tali casi il processo è caratterizzato, in momenti molto diversi da caso a caso, da appaiamento incompleto o da mancanza di appaiamento e da eliminazione di cromosomi, e rappresenta probabilmente il risultato di varie evoluzioni delle meiosi di significato ancora oscuro.
Nei Ditteri, gli Sciaridi presentano tale tipo di meiosi: in Sciara coprophila, che può servire d'esempio per l'intero gruppo, il processo è normale nella femmina con riduzione del numero dei cromosomi dal diploide 10 all'aploide 5 nelle cellule-uovo mature, mentre nel maschio si svolge senza appaiamento e la prima divisione è sostituita dall'eliminazione di 4 cromosomi di origine paterna, fra cui un X. Dei 6 cromosomi residui, poi, durante l'intervallo fra prima e seconda divisione un solo X si raddoppia, onde alla fine della seconda divisione i cromosomi risulteranno differentemente ripartiti ai due poli, 6 in uno e 7 nell'altro, che è quello destinato a fecondare l'uovo; il numero dei cromosomi dello zigote sarà perciò 5+7, cioè 12. Una successiva eliminazione di cromosomi durante lo sviluppo embrionale, come meglio vedremo più avanti, consente poi di ripristinare il normale numero diploide 10. Il processo è in realtà alquanto più complesso del quadro riassuntivo che abbiamo illustrato e presenta alcune varianti.
Nei Coccidi, Phenacoccus, Gossyparia ecc. (v. fig. 29), si osserva, durante la spermatogenesi, la trasformazione strutturale dei cromosomi di origine paterna, che divengono compatti, ossia eterocromatici, ricordando morfologicamente il corpo di Barr dei Mammiferi (v. fig. 10), e vanno quindi a formare due dei quattro spermatozoi, che poi degenerano: da una cellula diploide (spermatogonio) si formano così due soli spermatozoi, anziché quattro come avviene nello schema generale.
La riduzione cromosomica che avviene nella meiosi, secondo lo schema generale, è compensata nelle piante e negli animali dal processo fecondativo in cui avviene la fusione dei due nuclei maschile e femminile. Appaiamento e riduzione garantiscono la ricombinazione e la segregazione mendeliana dei geni: appare quindi facilmente comprensibile come la meiosi non sia riduttiva nelle forme unisessuate, mentre sostanzialmente inspiegate rimangono ancora quelle varianti del processo contrassegnate dall'eliminazione di cromosomi interi. Questa carenza interpretativa dipende anche dalle ancora scarse conoscenze della natura dell'appaiamento cromosomico e della fisiologia della meiosi in generale.
Da quanto è stato esposto risulta chiaramente che l'analisi genetica degli eucarioti, basata sulle leggi di Mendel, trova una possibilità di applicazione solo nei casi nei quali esistono bisessualità e diploidia e la meiosi si svolge secondo lo schema generale; in tutti gli altri, invece, in cui manca il sesso maschile, oppure è presente un numero cromosomico diverso dal diploide, o ancora il processo meiotico differisce in qualche modo dallo schema generale, per procedere all'analisi genetica occorre applicare schemi diversi da quelli delle leggi mendeliane, come è ben dimostrabile in Sciara coprophila e in qualche altra specie dello stesso genere, in diversi poliploidi e in Imenotteri con maschi aploidi. In altre parole, la genetica classica è riferita alle forme diploidi e bisessuate in cui la meiosi non differisce dallo schema generale, mentre nelle altre forme la trasmissione dei caratteri legati a geni localizzati nei cromosomi avviene secondo schemi diversi; nei casi in cui non è stato ancora possibile procedere all'analisi genetica, la conoscenza dei fenomeni cromosomici consente di prevedere il tipo di trasmissione. L'importanza preponderante attribuita alla genetica mendeliana classica dipende dal fatto che le specie degli eucarioti, che forniscono i materiali di studio più utilizzati, sono nella maggior parte dei casi diploidi e bisessuate, anche se non è giustificabile ignorare gli altri orgapismi che non rientrano nello schema generale. Una dimostrazione della molteplicità dei meccanismi cromosomici di trasmissione è offerta dai procarioti, nei quali si riscontra una divisione equazionale che determina gli stessi effetti della mitosi: mancano in tali organismi processi corrispondenti alla meiosi e alla fecondazione, mentre hanno luogo la coniugazione e altri fenomeni dei quali non vi sono corrispondenti negli eucarioti. Insomma, all'universalità del codice genetico non corrisponde un'universalità di meccanismi cromosomici di trasmissione, i quali sono invece molteplici, vari, diffusissimi alcuni, altri rari o rarissimi e tipici di gruppi sistematici anche limitati.
5. I cromosomi nel differenziamento somatico e nelle colture in vitro
Si è molto spesso affermato, e ancora adesso alcuni autori continuano a sostenere semplicisticamente, che nei diplonti il numero cromosomico delle cellule somatiche è il doppio di quello delle cellule germinali mature (gameti) e delle spore: se questa asserzione corrispondesse alla realtà, assumerebbe il valore di una legge fondamentale della biologia, dal momento che sono diplonti tutti i Metazoi e le piante superiori.
In realtà sono invece noti alcuni casi, molto probabilmente tutt'altro che infrequenti, nei quali le cellule somatiche non sono sempre diploidi: la triploidia dell'endosperma delle Fanerogame ne costituisce già una prova. Del resto, trattando della mitosi si è fatta già menzione delle mutazioni somatiche e dell'endomitosi: le variazioni del numero dei cromosomi somatici, infatti, nel senso di un aumento o di una diminuzione, possono essere rispettivamente di tipo moltiplicativo o poliploide oppure di tipo aneuploide. Daremo qualche indicazione di variazioni cromosomiche in sede somatica, che presentano una relazione fra divario dalla diploidia e differenziamento.
I casi più conosciuti riguardano la poliploidizzazione somatica nei Ditteri e in altri insetti e in molte piante, la diminuzione di cromatina nell'ascaride del cavallo (Parascaris equorum), la perdita sistematica di alcuni cromosomi durante l'embriogenesi degli Sciaridi e di alcuni mammiferi. La poliploidia somatica più appariscente, pur con i suoi aspetti peculiari, è rappresentata dalla politenia dei Ditteri, che abbiamo già trattato nel capitolo sul nucleo interfasico; aggiungeremo qui che questo fenomeno è assai precoce, perché nelle larve appena schiuse è già possibile osservare nuclei politenici, ed è comune a tutte le cellule somatiche a eccezione di quelle nervose, degli emociti e degli enociti. Peraltro non sono note le ragioni di questa ripartizione né la natura del rapporto tra politenia e differenziamento.
La poliploidizzazione costituisce la regola in molti altri insetti, per esempio nei Rincoti (nei quali però non si osserva politenia), soprattutto in cellule specializzate nella secrezione, come quelle dei tubi malpighiani, i cui nuclei acquisiscono numeri cromosomici estremamente elevati e divengono a poco a poco ampiamente e tortuosamente ramificati.
Nelle piante superiori la proprietà delle cellule meristematiche di diventare spesso poliploidi è ben nota e sfruttata in agricoltura: per esempio, la tecnica più rapida per ottenere tetraploidi nel pomodoro consiste nel cimare un germoglio e lasciare che il meristema tetraploide ne riformi uno nuovo, le cui cellule saranno appunto tetraploidi. Tra i casi di perdita di cromosomi, meno numerosi ma assai interessanti anche se di significato ancora oscuro, il più classico è quello di Parascaris equorum (v. fig. 30): in questa specie, durante la prima divisione dei biastomeri, inizia, nelle sole cellule che daranno luogo al soma, una perdita delle parti estreme dei quattro cromosomi e la frammentazione in molti piccoli corpi delle parti che rimangono, mentre la cellula madre di quelle germinali mantiene l'assetto intatto. L'ipotesi da alcuni formulata che questa perdita per rottura interessi zone eterocromatiche dei cromosomi prive o quasi di geni principali non sembra sostenuta da verifiche genetiche. Questo è comunque uno dei casi più documentati di differenziamento cromosomico fra linea somatica e linea germinale.
Il genere Sciara (e ci riferiremo alla specie Sciara Coprophila) presenta un esempio di perdita di cromosomi interi: in questo caso, infatti, due cromosomi mediocentrici di dimensioni cospicue, detti L (cioè limitati alla linea germinale), dotati però di scarsa attività genica ed eteropicnotici, vanno perduti per esclusione dal fuso durante le mitosi di segmentazione dell'uovo, insieme a un cromosoma X della femmina e a un X del maschio. Ne consegue una differenza numerica di cromosomi fra cellule germinali e cellule somatiche. Differenze simili esistono anche nei Marsupiali Isodon e Perameles, in cui le cellule di alcuni tessuti somatici posseggono un solo X, mentre le cellule sessuali immature maschili sono XXY.
Sembrerebbe possibile dedurre da questi esempi che le differenze del numero dei cromosomi fra cellule di tessuti diversi o fra cellule somatiche e cellule germinali sono limitate a casi particolari, mentre la poliploidizzazione dei meristemi rappresenta con ogni probabilità un fenomeno più generale anche se limitato a pochi gruppi. Il significato evolutivo di tali variazioni cromosomiche rimane oscuro, anche se appare logico ritenere che a esse possa comunque essere attribuito non già il valore di causa, ma piuttosto quello di conseguenza del differenziamento: si tratterebbe cioè di un fenomeno secondario anziché primario. In questi ultimi trent'anni sono stati condotti numerosi studi sulle variazioni dell'assetto cromosomico in relazione a stati patologici della cellula, con speciale riferimento alle neoplasie maligne, ma i risultati sono molto incerti: infatti, se è vero che molte cellule neoplastiche presentano anomalie numeriche e strutturali dei cromosomi, non si può tuttavia affermare che queste rappresentino un reperto obbligatorio nè tanto meno che siano interpretabili come causa piuttosto che come semplici manifestazioni secondarie della trasformazione neoplastica (v. neoplasie).
Ancor più recentemente, cioè durante l'ultimo decennio allo studio del decorso della mitosi in assenza di differenziamento si è aperto il nuovo campo di ricerca delle popolazioni cellulari coltivate in vitro. Com'è noto, si tratta di popolazioni derivate da cellule sia di embrioni sia di adulti, che dapprima si moltiplicano piuttosto stentatamente o tendono a esaurire le proprie capacità di moltiplicazione (colture primarie) e successivamente, dopo il verificarsi di un evento di natura ignota, acquistano capacità di moltiplicazione indefinita (linee stabilizzate), mostrando qualche volta variazioni dell'assetto cromosomico che spesso diventa diploide.
I dati raccolti sulle variazioni dell'assetto cromosomico di cellule in coltura sono numerosissimi e si possono raggruppare in quelli che si riferiscono ai Mammiferi (uomo, topo, criceto, ecc.) e quelli che riguardano D. melanogaster. Oggi si coltivano cellule delle specie più diverse di Vertebrati e di alcuni Invertebrati (Lepidotteri, Ditteri, Blatte), ma più spesso i risultati sono utilizzati per l'analisi dell'assetto cromosomico della specie o per lo studio dell'effetto di mutageni sui cromosomi piuttosto che per uno studio sistematico della dinamica dell'assetto cromosomico in una popolazione cellulare di colture continue. Manca ancora, inoltre, un tentativo sistematico di comparazione fra i risultati ottenuti in osservazioni condotte su specie disparate, come su alcuni mammiferi e sulla drosofila. Tuttavia si può considerare dimostrato che nelle popolazioni cellulari stabilizzate in vitro compaiono mutazioni cromosomiche e genomiche con una frequenza certamente rilevante, anche se non è possibile effettuare alcun paragone con le variazioni che spontaneamente si manifestano nelle cellule dei vari tessuti; rispetto a quanto accade in condizioni naturali, inoltre, l'ambiente di coltura è sottoposto a una selezione assai più debole, tanto che, dopo anni di stabilizzazione, le cellule coltivate possono presentare un assetto cromosomico molto diverso da quello normale. In generale, le cellule di linee stabilizzate mostrano una cospicua tendenza a poliploidizzare (che tuttavia non consente di ammettere in ogni caso l'esistenza di una correlazione fra stabilizzazione e moltiplicazione del numero dei cromosomi) e una grande abbondanza di mutazioni cromosomiche, responsabili dell'elevata frequenza degli assetti diploidi e delle profonde alterazioni morfologiche dei cromosomi, che spesso non sono più comparabili con quelli normali. Inoltre, la poliploidizzazione è generalmente seguita da perdita di cromosomi. In D. melanogaster (v. fig. 31) il confronto fra le variazioni mutazionali spontanee compatibili con la vita, e perciò trasmissibili, e quelle presenti in linee stabilizzate ha consentito di dimostrare che alcune sono comuni alle due condizioni (per esempio la presenza di un solo cromosoma del 4° paio o di un soprannumerario frammentato), altre (per esempio la sostituzione di due acrocentrici a uno dei grandi metacentrici del 2° paio) sono esclusive delle cellule coltivate in vitro: sembrerebbe pertanto accertato che questi elementi sono in grado di tollerare variazioni del cariotipo certamente insopportabili per le cellule di un organismo vivente, cosicché appare chiara la necessità di un controllo abbastanza rigido, durante lo sviluppo dei tessuti, delle variazioni mutazionali spontanee del cariotipo.
Bibliografia
Barigozzi, Cl., Ghidoni, A., Cromosomi e meccanismi ereditari, Torino 1976.
Beermann, W., Riesenchromosomen, in Protoplasmatologia, vol. VI-D, Berlin 1962.
Caspersson, T., Zech, L., Johannson, C., Modest, E. J., Identification of human chromosomes by DNA-binding fluorescent agents, in ‟Chromosoma", 1970, XXX, pp. 215-227.
Darlington, C. D., Cytology, London 1965.
Du Praw, E. J., Dna and chromosomes, New York-Chicago 1970.
Fréderic, J., Distèche, C., Quelques données sur la structure et l'identification des chromosomes, in ‟Archives of biology", 1973, LXXXIV, pp. 115-145.
Geitler, L., Endomitose und endomitotische Polyploidisierung, in Protoplasmatologia, vol. VI-C, Berlin 1953.
Hess, O., Meyer, F., Genetic activity of the chromosomes in Drosophila during spermatogenesis, in ‟Advances in genetics", 1968, XIV, pp. 171-225.
Lima-de-Faria, A. (a cura di), Handbook of molecular cytology, Amsterdam-London 1969.
Mirsky, A. E., Ris, H., Isolated chromosomes. The chemical composition of isolated chromosomes, in ‟Journal of general physiology", 1947, XXXI, pp. 1-19.
Mittwoch, U., Genetics of sex-differentiation, New York-London 1973.
Purdom, C. E., Genetic effects of radiations, London 1963.
Stern, C., Genetic mosaics and other essays, Cambridge, Mass., 1968.
Swanson, C. P., Cytology and cytogenetics, Englewood Cliffs, N. J., 1957.
Swanson, C. P., Merz, T., Young, W. J., Cytogenetics, Englewood Cliffs, N. J., 1967.
Taylor, J. H., The structure and duplication of chromosomes, in Genetic organization (a cura di E. W. Caspari e A. W. Ravin), New York-London 1969, pp. 163-216.
Tschermak-Woess, E., Strukturtypen der Ruhekerne von Pflanzen und Tieren, in Protoplasmatologia, vol. V-I, Berlin 1963.
Turpin, R., Lejeune, J., Les chromosomes humains, Paris 1965.
White, M. J. D., Animal cytology and evolution, Cambridge 19733.
Evoluzione e ontogenesi
di Leslie C. Dunn
Sommario: 1. Aspetti della teoria della selezione naturale. 2. Il sistema genico nei processi evolutivi. 3. Controllo genico dello sviluppo. 4. La conservazione dei sistemi viventi. □ Bibliografia.
1. Aspetti della teoria della selezione naturale
La formulazione della teoria darwiniana del meccanismo dell'evoluzione fu uno dei grandi avvenimenti del sec. XIX. La teoria affermava che la vita attuale, sia dell'uomo sia degli altri esseri viventi, è una continuazione della vita del passato e da quella deriva, per cambiamenti, attraverso processi naturali. Oggi l'enunciato di tale teoria è stato verificato, con l'approfondimento dei processi naturali e la dimostrazione ch'essi si basano sul materiale genico che è ovunque costituito dalle stesse strutture elementari, cioè dalle basi nucleotidiche che formano le unità che si ripetono nella molecola polimerica degli acidi nucleici. Questa prova della unicità della natura vivente non ha ancora prodotto il suo pieno effetto né nel campo filosofico e morale, né in quello scientifico. Risulta evidente che la scoperta del meccanismo con cui tale universalità si realizza ha posto una quantità di problemi relativi alla vita e al destino dell'uomo. Alcuni di questi saranno discussi alla fine della rassegna storica.
La teoria della selezione naturale fu presentata in forma succinta a una riunione della Linnean Society di Londra il 1° luglio 1858 da Charles Darwin e Alfred Russel Wallace. Ognuno dei due autori era giunto indipendentemente dall'altro alla formulazione della medesima idea generale, ma fu Darwin che nel 1859, nel suo libro On the origin of species by means of natural selection on the preservation of favoured races in the struggle for life, espresse in forma definitiva la teoria come meccanismo fondamentale dell'evoluzione, traendone le prove da dati ricavati dalla storia naturale. Le prove fondamentali furono fornite dagli effetti della selezione artificiale sulle variazioni degli animali e delle piante in domesticazione (che furono poi oggetto di un altro libro di Darwin, Variation of animals and plants under domestication, London 1868). Gli argomenti di natura storica, come quelli addotti da Darwin e dai suoi successori, non costituiscono prove altrettanto definite quanto quelle che si ottengono da un'analisi sperimentale. Soltanto dopo la scoperta della struttura particolata del materiale ereditario e delle sue applicazioni alle variazioni delle frequenze geniche in popolazioni naturali o sperimentali, si poté costruire una teoria sintetica dell'evoluzione sulle basi stabilite da Darwin. E perciò il valore della teoria di Darwin si poté apprezzare soltanto dopo la nascita e lo sviluppo della genetica delle popolazioni che avvennero nel periodo 1920-1940.
Tra la storia della genetica e quella della biologia evoluzionistica vi è uno stretto parallelismo. L'idea centrale di ciascuna di esse fu concepita verso la metà del XIX secolo e lunga fu la gestazione di entrambe. La teoria dell'evoluzione subì un lungo ritardo, probabilmente a causa della sua dipendenza dalla teoria del gene. Se situiamo nel 1900 la nascita della genetica, possiamo far risalire la nascita della teoria genetica dell'evoluzione agli anni 1930-1932.
L'opera di Darwin, che produsse effetti profondi sul pensiero scientifico e filosofico fin dal 1859, non ebbe alcuna influenza diretta sulla scoperta di Mendel delle leggi della ereditarietà. La teoria dell'ereditarietà di Darwin (la sua cosiddetta ‛ipotesi provvisoria della pangenesi') fu pubblicata nel 1868, due anni dopo la pubblicazione del lavoro di Mendel. Gli esperimenti di Mendel erano incominciati tre anni prima della pubblicazione della Origin of species, e non vi è traccia di connessione tra l'opera di Mendel e la teoria di Darwin prima dell'anno della riscoperta, il 1900. E così due teorie, che oggi consideriamo complementari, restarono isolate l'una dall'altra per due generazioni (v. evoluzione).
2. Il sistema genico nei processi evolutivi
L'estensione della teoria del gene all'interpretazione dell'evoluzione fu piuttosto tardiva e non seguì direttamente al riconoscimento dei principi che stanno alla base dell'equilibrio mendeliano (1908). Fino a circa la metà degli anni venti perdurarono fra i biologi dubbi sulla validità della teoria della selezione naturale. La dimostrazione da parte di Muller e della scuola di Morgan che in Drosophila la mutazione genica è un processo ricorrente che fornisce la variabilità su cui la selezione naturale può agire, segnò la svolta per un nuovo orientamento generale. La prima esplicita formulazione di tale indirizzo fu fatta dal biologo russo S.S. Četverikov nel 1926. Il suo lavoro innovatore fu pubblicato in lingua russa e le difficoltà che a quel tempo esistevano nelle comunicazioni tra Est e Ovest causarono un ulteriore ritardo alla sua diffusione, ma non vi è dubbio che Četverikov abbia individuato i principi fondamentali della genetica evoluzionistica: l'esistenza di geni mutanti in popolazioni naturali; la possibilità che questa variabilità si mantenga per segregazione e ricombinazione mendeliane; l'opera di fattori d'isolamento e fortuiti che intervengono nel processo della selezione naturale come forze evolutive tendenti a rovesciare la condizione di equilibrio delle frequenze geniche. Nel 1927 Četverikov pubblicò osservazioni sperimentali su popolazioni naturali di Drosophila che convalidavano la sua teoria.
Nel frattempo, in Inghilterra e negli Stati Uniti, era stato costruito un edificio teorico, chiaramente espresso dai titoli di tre pubblicazioni che segnarono la nascita d'un nuovo campo della biologia: la genetica delle popolazioni e dei processi dell'evoluzione. Le tre opere erano le seguenti: Genetical theory of natural selection di R.A. Fisher, 1930; Evolution in mendelian populations di S. Wright, 1931; The causes of evolution di J.B.S. Haldane, 1932. I tre autori, pur discordando in alcuni punti, concordavano sulla teoria fondamentale, secondo cui il meccanismo dell'evoluzione è condizionato dal sistema genico e il sistema stesso è il risultato dei processi dell'evoluzione. Tutti davano rilievo alla mutazione come fonte di variabilità e alla selezione naturale come processo primario del cambiamento delle frequenze geniche che sta alla base del divergere delle linee evolutive.
Wright pose l'accento sul fatto che la condizione di una popolazione rappresenta un equilibrio fra pressioni sistematiche e dirette di mutazione, selezione e isolamento, e fluttuazioni casuali di frequenze geniche. Egli inoltre attribuì ai cambiamenti dell'equilibrio dinamico nell'ambito di questi parametri della popolazione un carattere di prova ed errore e ciò lo condusse a considerare l'evoluzione come un processo opportunistico.
Si poté così giungere a una classificazione, che consente una verifica sperimentale, delle forze evolutive che tendono a far variare le frequenze geniche: 1) pressione di mutazione, cioè cambiamenti di A in a o di a in A con frequenze diseguali; 2) selezione, cioè perpetuazione differenziale di alleli alternativi, A o a, o di gruppi associati di geni; 3) migrazione differenziale nella popolazione o dalla popolazione di portatori di forme differenti di geni, A o a; 4) eventi casuali dovuti al campionamento in piccole popolazioni (deriva genetica casuale).
Negli anni 1930 e 1940 la teoria, sottoposta a prove sperimentali, progredì. Una prima strutturazione delle nuove conoscenze così raggiunte fu espressa nel libro di Th. Dobzhansky Genetics and the origin of species (1937), che segnò la nascita della genetica sperimentale delle popolazioni e suggerì la via per l'unificazione della genetica, della sistematica, della paleontologia e di altri approcci allo studio dell'evoluzione entro il quadro più generale della biologia evoluzionistica.
Tale campo di studio e i risultati conseguiti sono troppo vasti per poter essere trattati brevemente (v. evoluzione). Nella genetica emersero nuovi orientamenti e punti di vista, come il considerare la formazione delle razze un cambiamento microevolutivo dovuto a differenze locali delle frequenze geniche in una popolazione nell'ambito di una specie; o il considerare una specie come un'unica popolazione mendeliana entro la quale i geni possono circolare per riproduzione sessuale, però separata dal punto di vista riproduttivo da altre popolazioni. Un aspetto comune della maggior parte delle popolazioni che si incrociano fra di loro è il polimorfismo, ossia la presenza, entro una popolazione, di due o più alleli o di uno o più geni con frequenze inferiori a quelle che possono dipendere da mutazione. Questa riserva di eterogeneità genetica si mantiene per opera della selezione, che spesso è del tipo che favorisce gli eterozigoti. Ciò porta a una struttura della popolazione genetica in cui ‟non vi è alcun tipo normale, ma soltanto una norma adattativa composta da una serie di genotipi, che hanno la proprietà comune di presentare una idoneità (fitness) soddisfacente nella maggior parte degli ambienti che la popolazione incontra più frequentemente" (v. Dobzhansky, 1970, p. 198). Il contrasto di questa opinione con la visione classica precedente del ‛tipo' adattativo uniforme nella popolazione dà la misura dell'entità del cambiamento apportato dalla genetica.
3. Controllo genico dello sviluppo
Lo sviluppo di un organismo complesso, qual è quello di un Vertebrato, dall'uovo ha tenuto desto l'interesse dei naturalisti per molti secoli. A partire dalla fine del XIX secolo, quando si scoprì che un nuovo individuo ha origine da una singola cellula formata dall'unione di due nuclei (provenienti dall'uovo e dallo spermio), molti biologi supposero che il controllo dello sviluppo dovesse essere situato nel materiale che passa nella nuova cellula, per il tramite dei due nuclei (v. embriologia).
Nel 1883 A. Weismann si chiedeva come possa accadere che una cellula germinale contenga la Anlage per la formazione di un intero, complesso individuo. Gli embriologi del XIX secolo descrissero i processi dello sviluppo di molti tipi di animali e di piante e intrapresero l'analisi causale dei processi.
Con la scoperta del sistema mendeliano dell'ereditarietà particolata questi problemi furono visti in una nuova luce. Il materiale ereditario che passa attraverso le cellule germinali risultò costituito da un mosaico di elementi separati, i geni, i quali non contengono informazioni specifiche per una essenziale caratteristica di sviluppo: le trasformazioni continue determinano il differenziamento di cellule, tessuti e organi che esplicano funzioni diverse e tuttavia costituiscono un singolo individuo integrato, con caratteristiche sue proprie di forma e di comportamento.
Th. Boveri (1902, 1909), con brillanti esperimenti sulle uova di riccio di mare fecondate da più spermi, dimostrò chiaramente che lo sviluppo normale richiede che nel nuovo individuo siano presenti tutti i tipi di cromosomi parentali. Da qui derivò l'ipotesi che i caratteri ereditari che influenzano i processi dello sviluppo siano distribuiti in tutti i cromosomi e che ogni qualità di cromosoma sia una struttura unica. L'individualità dei cromosomi fu dimostrata morfologicamente anche da W.S. Sutton nel 1903.
La ‛teoria cromosomica' di Sutton e Boveri precorse la dimostrazione, data da Morgan e dalla sua scuola in base agli esperimenti di ibridazione in Drosophila, che il materiale ereditario è rappresentato da una sequenza lineare di geni nei cromosomi. Ben presto la teoria fu confermata ed estesa da genetisti che studiavano altri organismi animali e vegetali. I risultati di tali ricerche chiarirono i meccanismi della trasmissione ereditaria, ma non ebbero come scopo l'interpretazione dello sviluppo. Nel 1926 Morgan scriveva: ‟La teoria del gene, così formulata, non entra nel merito delle modalità del rapporto del gene con il prodotto finale, cioè il carattere" (Th. H. Morgan, The theory of the gene, New Haven 1926, p. 26). Tuttavia era chiaro a Morgan, come ad altri, che ‟un cambiamento in un gene produce effetti definiti sui processi dello sviluppo". Il punto essenziale era che una teoria del meccanismo della trasmissione poteva essere stabilita ‟senza cercare di spiegare la natura dei processi causali che collegano gene e carattere". Si stabilì in tal modo un nuovo atteggiamento di ricerca, tipico del XX secolo, consistente nel separare per scopi analitici un problema relativamente semplice, che si risolve in base al risultato numerico degli esperimenti di ibridazione, dal problema più complesso che coinvolge processi di regolazione che variano nel corso del tempo. Osservazioni sugli effetti di geni mutanti in numerosi animali e piante multicellulari rivelarono concordemente che esistono due relazioni generali tra i geni e i processi dello sviluppo. Si notò innanzitutto che ogni gene mutante, pur essendo stato identificato in prima istanza per qualche effetto vistoso, come un cambiamento del colore degli occhi, se studiato accuratamente dimostrava di avere effetti complessi su altri caratteri visibili, spesso su processi metabolici dimostrabili per via biochimica, talvolta su di un ormone capace esso stesso di produrre una quantità di effetti, e sul comportamento, come le risposte alla gravità, alla luce, al calore e ad altre variabili ambientali. Una seconda osservazione generale fu che molte delle qualità che distinguono gli individui di una specie, quali le dimensioni, la forma, la durata della vita, la produttività o capacità fisica o mentale, dipendono dalle interazioni tra numerosi geni. Dalle osservazioni sulla pleiotropia (effetti molteplici di geni distinti) e sulla interazione ditali effetti, sorse l'ipotesi che lo sviluppo di un individuo sia come una rete in cui si connettono gli effetti dei prodotti primari dei geni, condizionati dall'ambiente in cui essi si esplicano. Il problema di stabilire la relazione tra geni e sviluppo divenne così il problema del controllo della funzione individuale del gene e delle modalità dell'interazione dei geni.
Sebbene la struttura del gene, il modo in cui esso si reduplica e la sua funzione, o prodotto primario, siano ormai chiaramente stabiliti (v. sotto, cap. 4), non si è ancora fatta luce sul modo in cui i geni controllano lo sviluppo nel suo complesso. Si sa che il nucleo produce messaggi che possono sopravvivere al nucleo stesso per parecchie settimane e influenzare i processi morfogenetici a distanza (J. Hämmerling, 1933). Si è poi trovato che il prodotto primario di un gene è una trascrizione complementare della sua sequenza di nucleotidi nel DNA sotto forma di un RNA messaggero da cui il messaggio codificato viene tradotto in una sequenza di amminoacidi in un polipeptide. Quasi certamente le istruzioni provenienti dal nucleo assumono questa forma, ma resta da scoprire in quale modo i messaggi provenienti dai diversi geni sono comunicati al di là della cellula in cui prendono origine e come sono coordinate le reazioni delle diverse cellule.
Resta inoltre inesplicato il paradosso principale che lasciò perplessi i primi biologi. Sembra che tutti i nuclei del corpo degli animali e delle piante superiori contengano tutti i rappresentanti degli stessi geni, e tuttavia le cellule si differenziano tra loro per strutture e funzioni. È stato esaminato sperimentalmente il problema se i nuclei delle cellule differenziate conservino la totipotenza dei nuclei dell'uovo, la quale è in grado di produrre i vari tipi di cellule nel corso dello sviluppo.
Nelle piante superiori è evidente che una singola cellula di un tessuto differenziato può produrre l'intero corpo della pianta (lo sporofito). Negli animali i risultati di prove sperimentali sono ambigui; però, almeno in alcuni casi (J.B. Gurdon, 1963), un nucleo di una cellula intestinale, sostituito al nucleo dell'uovo, può conservare la capacità di dirigere lo sviluppo di un individuo normale. Quando sembrò evidente che tutti i nuclei di un individuo contenessero gli stessi geni, si pensò che il differenziamento cellulare potesse presentarsi secondo un'altra modalità, e precisamente che non tutti i geni fossero attivi in tutte le cellule e in ogni momento (v. embriologia).
Le prove di un'attività genica differenziale in cellule diverse si sono ottenute con lo studio dei cromosomi politenici giganti di Insetti Ditteri (Beermann, 1965). La successione di bande (cromomeri) di DNA lungo questi cromosomi corrisponde alla successione dei geni nella mappa di ricombinazione. Il cromomero stesso può essere considerato come un centro di sintesi (indicata dalla produzione di RNA) in certi tessuti e in certi momenti, e ciò può significare che nei diversi tessuti i geni possono essere ‛accesi' o ‛spenti'. In uno dei due cromosomi X delle femmine dei Mammiferi è stato dimostrato un interessante caso di attività differenziale dei geni (M.F. Lyon, 1968). Mentre nei primi blastomeri degli embrioni di sesso femminile entrambi i cromosomi X sono attivi, in uno degli stadi successivi, ma sempre precoci, uno di essi diventa inattivo. L'inattivazione (che si realizza con modalità sconosciute) colpisce a caso o l'X di provenienza materna o quello paterno. Le femmine dei Mammiferi, specie umana compresa, si sviluppano quindi come mosaici in cui i geni di origine paterna localizzati nel cromosoma X sono attivi in alcune cellule, quelli di origine materna in altre. Gli effetti di questa determinazione precoce si mantengono, a quanto pare, per tutta la durata della vita.
Nel moscerino dell'aceto, Drosophila, cellule che nell'embrione sono state determinate in un dato senso in alcuni casi sono capaci di ‛transdeterminazione', cioè possono produrre, nell'adulto, un altro tipo di cellula (E. Hadorn, 1968). Il cambiamento di destino o di direzione parrebbe associato a un aumento della sintesi del DNA, forse concomitante con lo spostamento dell'attività da un gruppo di geni a un altro.
Non è stata ancora formulata alcuna teoria circa l'attivazione o la soppressione dell'attività genica specifica negli organismi pluricellulari. Un modello, però, è rappresentato dalla teoria del circuito di regolazione o operon di F. Jacob e J. Monod (1961), che è stata applicata con successo all'interpretazione della regolazione genica nei Batteri. La teoria ammette l'esistenza di due tipi di geni sostanzialmente differenti. I geni ‛strutturali' controllano la formazione degli RNA messaggeri che determinano in modo specifico la sequenza degli amminoacidi nelle proteine. I geni ‛controllori' regolano il funzionamento del DNA e sono di due tipi, regolatori e operatori. Un operon è un' associazione di geni strutturali adiacenti controllati da un operatore. Quando questo è attivo, ogni gene strutturale sintetizza la sua proteina specifica. L'operatore può essere ‛spento', e con esso i geni strutturali, da una sostanza repressiva proveniente da un gene regolatore situato in un altro punto della cellula. Il regolatore è sensibile allo stato metabolico della cellula, così che le proteine specifiche, cioè gli enzimi, sono sintetizzate oppure no a seconda della condizione metabolica della cellula. Possono così insorgere differenze fra le cellule nei riguardi dell'attività genica. Fino a oggi non vi sono prove che dimostrino che tale meccanismo di differenziamento sia funzionante nelle cellule degli organismi superiori. Evidentemente esso permette di formulare ipotesi molto utili per saggiare i sistemi di controllo all'interno delle cellule, ma non è ancora servito a suggerire sistemi di coordinamento delle attività fra gruppi di cellule in via di sviluppo. La regolazione temporale e la coordinazione costituiscono i due principali problemi insoluti della genetica dello sviluppo (v. embriologia).
4. La conservazione dei sistemi viventi
Verso la metà del sec. XX la storia della biologia fu scossa da eventi grandiosi. Essi consistono nella scoperta della struttura chimica e delle modalità di reduplicazione degli acidi nucleici, che costituiscono il sistema genetico di tutti gli organismi; nella scoperta della struttura e delle modalità della sintesi delle proteine, che costituiscono i sistemi effettori di tutti i processi della vita; nella ricostruzione del modo in cui il sistema genetico regola la sintesi degli enzimi e quella di altre proteine. Esula dagli scopi di questo articolo la trattazione dei particolari di tali scoperte, ma per l'importanza che esse hanno avuto nella trasformazione profonda delle conoscenze del XX sec. circa l'ereditarietà, la variabilità e le trasformazioni evolutive, non si potrà fare a meno di ricordare alcuni effetti che esse hanno prodotto sul pensiero biologico (v. biologia; v. biologia molecolare; v.acidi nucleici; v. proteine).
Un postulato fondamentale della disciplina che è stata denominata genetica molecolare o biologia molecolare è il cosiddetto ‛ centrale', stabilito da F. C. H. Crick nel 1958. Esso afferma che la sequenza delle basi nucleotidiche in un acido nucleico funziona come un semplice codice che controlla la sequenza degli amminoacidi di una particolare proteina, e che perciò l'informazione genetica si trasmette in una sola direzione: dal gene (DNA) alla proteina e mai nella direzione inversa. Si usa esprimere questo fatto con la formula DNA→RNA→proteina: la prima freccia indica la trascrizione di un codice dal DNA all'RNA, la seconda la traduzione della sequenza dell'RNA nella sequenza degli amminoacidi nella molecola proteica. La scoperta del DNA nel sec. XIX (J.F. Miescher, 1870) fu il primo passo verso la formulazione del dogma. Molto più tardi si identificò il DNA con il ‛principio trasformante' nei Batteri (O.T. Avery e altri, 1944), poi con il materiale genetico in quanto origine della sintesi proteica nel batteriofago (A.D. Hershey e M. Chase, 1952). La composizione del DNA a coppie di basi fu dimostrata da E. Chargaff nel 1950; la struttura e il modo di reduplicazione della molecola del DNA da Watson e Crick nel 1953 (v. Watson, 1965). Nel 1961 Brennen, Jacob e Meselson scoprirono l'RNA messaggero; la decifrazione del codice genetico fu opera di M. W. Nierenberg e J. H. Matthei nel 1961 e di Crick e altri, successivamente. Il coronamento di queste ricerche fu raggiunto con la sintesi chimica di un gene (H. G. Khorana, 1970).
Abbiamo semplicemente ricordato alcune tappe di questa sequenza di ricerche per poter dare un'idea della rapidità del loro progresso nonchè della natura dei metodi e delle conquiste teoriche. Lo studio delle molecole viventi è stato portato avanti con i metodi della fisica e della chimica: analisi chimiche, modelli della struttura dei cristalli ottenuti con la diffrazione con i raggi X, azione in vitro di catalizzatori per degradare e poi ricostruire porzioni delle catene del DNA e RNA; separazione di molecole e di loro parti con la cromatografia, la elettroforesi e la centrifugazione ad alta velocità; ricombinazione in vitro di frammenti sintetizzati di DNA usati come modello per la formazione di un enzima: cioè una vera e propria sintesi di un'unità genetica attiva.
Tali metodi sono stati applicati ai sistemi che l'analisi genetica aveva dimostrato potersi risolvere in sequenze lineari di elementi genetici. Si sono raggiunti questi risultati lavorando soprattutto con i Batteri e i virus, che si riproducono con grande rapidità, in quanto è stato possibile saggiare con metodi strettamente sperimentali teorie basate sulle stesse idee meccanicistiche che avevano guidato le scoperte della fisica e della biochimica. La teoria cromosomica della genetica classica, con i suoi sviluppi, aveva dimostrato che le più semplici ipotesi meccanicistiche sulla struttura del materiale genetico erano le sole che normalmente si dimostravano corrette. Le ricerche eseguite con porzioni progressivamente più piccole di acidi nucleici, ivi compreso il controllo da parte di una unità-codice di tre paia di nucleotidi contenenti poche dozzine di atomi, dimostrarono che era necessario applicare al livello molecolare gli stessi principi scientifici e gli stessi tipi di ragionamento di cui già si era riconosciuta la validità nella genetica classica. Per tenere unite coppie di basi complementari si rivelarono sufficienti i deboli legami idrogeno, senza dover ricorrere a nuove forze. Con queste esperienze si voleva provare sostanzialmente se, oltre alle istruzioni codificate nel DNA per la sintesi delle proteine, ne fossero richieste altre per la replica della conservazione della continuità genetica e per la ricostruzione dei modelli ereditari nei discendenti. Il risultato dimostrerebbe che le istruzioni ereditate sono sufficienti a determinare l'automontaggio di nuovi individui. Ciò si presenta con grande evidenza nella ricostituzione di un batteriofago a partire da parti incomplete che sono sintetizzate da genomi virali resi difettosi per mezzo di mutazioni che colpiscono geni diversi. Se si mescolano in vitro tutte le parti, il virus si ricostituisce senza ulteriore guida. Che questo aspetto misterioso del problema sia risolto da un meccanismo nei sistemi viventi meglio conosciuti; cioè i virus e i Batteri, ha determinato nei biologi di quest'ultimo periodo del XX secolo la fiducia che l'estensione dei principi meccanicistici potrà essere sufficiente a spiegare le operazioni che avvengono in sistemi affini degli organismi superiori, il controllo dello sviluppo e del differenziamento e le operazioni che stanno alla base delle funzioni nervose e mentali.
È interessante sottolineare che il risultato del processo unidirezionale del ‛dogma centrale', secondo cui le proteine ricevono ma non danno istruzioni, tende a smantellare l'ultimo rifugio di coloro che ancora credono, secondo la vecchia teoria del XIX secolo, nell'eredità dei caratteri acquisiti come fattore dell'evoluzione. I cambiamenti hanno inizio con errori nella replicazione del materiale genetico (gli acidi nucleici) e si manifestano in alterazioni delle proteine. Gli organismi sarebbero dunque costituiti in modo tale, al livello molecolare, che il processo inverso non può aver luogo; o per lo meno non se ne hanno prove. Tutto ciò aumenta la fiducia, generata dalla soluzione dei problemi della genetica al livello organismico e molecolare, che si possano risolvere anche quelli dell'organizzazione e del funzionamento degli organismi, nell'ambito di popolazioni, razze e specie e nella successione degli eventi evolutivi. E fa anche sperare che tali idee si possano applicare utilmente agli animali e - forse - all'organizzazione e al comportamento delle società umane.
Bibliografia
Beadle, G. W., Genetics and modern biology, Philadelphia 1963.
Benzer, S., On the topography of the genetic fine structure, in ‟Proceedings of the national Academy of Sciences", 1961, XLVII, pp. 403-416.
Brink, R. A. (a cura di), Heritage from Mendel, Madison 1965.
Dobzhansky, Th., Genetics of the evolutionary process, New York 1970.
Dunn, L. C., A short history of genetics, New York 1965 (tr. it.: Breve storia della genetica, Milano 1978).
Dunn, L. C., Genetics in historical perspective, in Genetic organization (a cura di E. W. Caspari e A. W. Ravin), vol. I, New York 1969, pp. 1-90.
Ebert, J. D., Sussex, I. M., Interacting systems in development, New York 19702.
Garrod, A. E., Inborn errors of metabolism, Oxford 19232.
Johannsen, W. L., Elemente der exakten Erblichkeitslehre, Jena 1909.
Křiženecký, J., Fundamenta genetica. The revised edition of Mendel's classic paper with a collection of 27 original papers published during the rediscovery era, Praha 1965.
Stubbe, H., Kurze Geschichte der Genetik bis zur Wiederentdeckung der Vererbungsregeln Gregor Mendels, Jena 19652.
Sturtevant, A. H., A history of genetics, New York 1965.
Taylor, J. H., Selected papers on molecular genetics, New York-London 1965.
Watson, J. D., The molecular biology of the gene, New York 1965, 19702, 19763 (tr. it.: Biologia molecolare del gene, Bologna 1967, 19722).
Wilson, E. B., The cell in development and heredity, New York 19283.
Applicazioni della genetica
di Giuseppe Montalenti
Sommario: 1. Applicazioni alla zootecnia e all'agricoltura. 2. Ingegneria genetica: a) ricombinazione del DNA; b) ibridazione di cellule somatiche. □ Bibliografia.
1. Applicazioni alla zootecnia e all'agricoltura
La coltivazione delle piante e la domesticazione degli animali d'interesse economico, o di valore estetico, ornamentale, sono pratiche che risalgono alla remota antichità e che sono state variamente sviluppate dalle diverse razze umane e nei diversi cicli culturali.
Probabilmente si può risalire a sette o otto millenni da oggi per trovare l'origine di queste pratiche nelle popolazioni dell'Asia centrale. Con il progredire della civiltà e il suo graduale spostamento dall'Asia centrale verso oriente (Cina, Giappone) e verso occidente, cioè verso l'Asia Minore, l'Africa (Egitto) e infine l'Europa, nuove specie vegetali e animali vennero condotte in domesticità, e si sviluppò man mano la formazione di razze più specializzate.
Il processo fondamentale che sta alla base della specializzazione razziale è la selezione. L'uomo ha utilizzato, a questo scopo, sia la selezione naturale, che ha agito nei vari ambienti in cui sono state portate le specie originarie, sia la selezione artificiale. L'uomo ha cioè scelto man mano i riproduttori le cui caratteristiche corrispondevano meglio alle prestazioni desiderate. Il processo è stato assai lento, ma nell'insieme piuttosto efficiente: all'inizio dell'età moderna esisteva un buon numero di razze di animali e di piante derivate da quelle dell'antichità, a cui, dopo la scoperta dell'America, si aggiunsero quelle delle specie vegetali indigene di quel continente (granoturco, pomodoro, patata, ecc.). L'allevamento e la selezione erano condotti senza una precisa base scientifica. Nel Settecento, soprattutto in Inghilterra, si cominciò a procedere in modo più rigoroso, specialmente per quanto riguarda gli animali, e si cominciò a tenere conto della genealogia (pedigree) dei riproduttori, in particolare dei cavalli, ma poi anche del bestiame bovino, dei maiali, ecc. Durante il sec. XIX si istituirono, dapprima in Inghilterra, poi in vari altri paesi, i registri dei riproduttori (studbooks), in cui erano annotate, per ciascun capo, le caratteristiche più salienti e più importanti. In campo agrario si fondarono stabilimenti per la produzione dei semi, specialmente di cereali, di cui il più famoso è quello di Svalòf in Svezia, costituito nel 1886. In Italia lo sviluppo della cerealicoltura si deve soprattutto all'attività di Nazareno Strampelli, titolare della cattedra ambulante di granicoltura a Rieti (1903), poi fondatore della R. Stazione Sperimentale di Granicoltura, sempre a Rieti (1907), e infine dell'Istituto Nazionale di Genetica per la Cerealicoltura a Roma (1919).
Dopo la riscoperta delle leggi di Mendel (1900) e la nascita della genetica, ci si rese conto che questa scienza poteva fornire le basi per elaborare procedimenti più razionali, più efficienti e rapidi per il miglioramento genetico delle piante e degli animali. Tuttavia dovettero trascorrere parecchi anni prima che tali speranze si realizzassero; all'inizio furono espressi dubbi da parte degli agronomi e degli zootecnici sull'importanza dei principi della genetica per le applicazioni pratiche. In particolare dovette essere elaborata, nel decennio 1910-1920, per opera soprattutto di H. Nilsson-Ehle (a Svalöf) e di E. M. East (negli Stati Uniti), in campo vegetale, e di A. Lang (in Svizzera), in campo animale, la teoria della polimeria, o dei poligeni, che si applica a quei casi (e sono la maggioranza) in cui un carattere è controllato da numerose coppie di geni, anziché da una sola, come negli esempi paradigmatici che si trovavano nei trattati di genetica. La teoria della polimeria è soprattutto importante per l'eredità dei caratteri quantitativi che sono fra i più interessanti dal punto di vista pratico.
Una chiarificazione essenziale introdotta dalla genetica teorica, inizialmente per merito di W. Johannsen, al quale si deve la terminologia, è la distinzione fra genotipo e fenotipo. Il fenotipo non è uno specchio fedele del genotipo, e poiché i caratteri apparenti sono il risultato della interazione della costituzione genetica (genotipo) con i fattori ambientali, i quali producono modificazioni non ereditarie, è necessario cercare di sceverare le due componenti, genetica e ambientale, e possibilmente misurarne gli effetti relativi. Ciò fu fatto soprattutto da J. L. Lush, che nel 1937 pubblicò un'opera, Animal breeding plans, in cui sono posti i fondamenti teorici e pratici del miglioramento genetico degli animali. Al Lush si deve, fra l'altro, una formula per la misura del coefficiente di ereditabilità (v. eredità biologica).
Attualmente il miglioramento genetico delle piante e degli animali è basato su metodi razionali, elaborati in base ai principi della genetica. In particolare i metodi di selezione (selezione di massa, selezione familiare) e il modo di saggiarne gli effetti (progeny test o saggio della discendenza) sono attuati su basi scientifiche, anziché puramente empiriche. Per lo più si deve tener conto non di un solo carattere, ma di molti, per cui conviene farne una sorta di media ponderata, che si può esprimere con un indice. Per esempio, per i polli, I. M. Lerner enumera non meno di sedici caratteri che l'allevatore deve considerare insieme: fecondità, schiudibilità delle uova, frequenza dei pulcini deformi, mortalità dei pulcini e dei pollastri, istinto di chioccia, dimensioni del corpo, velocità di raggiungimento della maturità sessuale, frequenza di ovideposizione, dimensione dell'uovo, forma dell'uovo, struttura, spessore, colore del guscio, qualità del contenuto, frequenza di macchie sanguigne nell'uovo. Analogamente per le piante molti sono i caratteri che concorrono alla valutazione del ‛merito' di una varietà, o di un cultivar.
Pertanto, mentre da un lato l'allevatore tende a ottenere razze pure (con l'autofecondazione nelle piante, con incroci consanguinei negli animali), in cui siano esaltati alcuni caratteri, dall'altro, considerando gli svantaggi di un elevato grado di omozigosi per geni sfavorevoli o letali (sterilità, alta frequenza di difetti genetici), può convenire invece operare incroci fra razze diverse, al fine di produrre combinazioni nuove e di introdurre in un dato stipite caratteri utili che si trovano in un altro.
Una delle realizzazioni più spettacolari del fenomeno noto ai genetisti con il nome di eterosi, o lussureggiamento degli ibridi, è stata ottenuta negli Stati Uniti d'America con il granoturco ibrido. Questo deriva dall'incrocio di linee selezionate e dà un rendimento eccezionale, come è dimostrato dalle figg. 1, 2, 3.
Nelle piante la produzione di genotipi nuovi è più facile che non negli animali, sia perché le ibridazioni, anche interspecifiche, si realizzano più agevolmente, sia perché si può indurre la poliploidia con adeguati trattamenti chimici, o in seguito a taluni tipi di incrocio. Fra le realizzazioni più importanti, dal punto di vista pratico, ricorderemo soltanto una nuova specie di cereale ottenuta ibridando frumento (Triticum vulgare, con numero di cromosomi 2n = 42) e segale (Secale cereale, con 2n = 14). L'ibrido ha n = 21 + 7 = 28 cromosomi; ma si possono ottenere individui tetraploidi, con 2n = 58 cromosomi, che sono stabili e capaci di riprodursi regolarmente per via sessuale: questi sono indicati con il nome Triticale, specie che presenta alcuni vantaggi rispetto alle specie parentali. Molti altri casi di ibridi-poliploidi sono stati ottenuti, specialmente nelle piante orticole.
Anche la semplice poliploidia, non preceduta da ibridazione, e ottenuta mediante il trattamento con colchicina, ha date luogo a varietà pregevoli, sia nelle piante orticole sia nelle ornamentali.
Un'altra applicazione dei dati della genetica consiste nell'induzione sperimentale di mutazioni con i raggi X o con sostanze mutagene. In tal modo si aumenta di molto la disponibilità dei mutanti così che la selezione può essere fatta molto più rapidamente, a partire da un'ampia gamma di mutazioni.
Negli animali le possibilità di intervento del genetista sono più limitate, sia perché i sistemi riproduttivi sono alquanto diversi (l'impossibilità della autofecondazione, la limitata possibilità di ibridazioni interspecifiche, l'impossibilità della poliploidia, sono condizioni restrittive caratteristiche dei Vertebrati, che sono gli animali economicamente più importanti), sia perché il tempo di generazione degli animali utili è generalmente più lungo che non quello di molte piante. Tuttavia anche nel campo della zootecnia molte pratiche empiriche sono state razionalizzate: così la definizione e la valutazione del ‛merito', i sistemi d'incrocio (la fecondazione artificiale ha facilitato le pratiche relative ai sistemi di selezione). Per alcune specie di animali d'allevamento si è raggiunto lo stadio della produzione su base industriale, anziché su base familiare di piccole installazioni locali. Particolarmente notevoli sono i risultati ottenuti, oltreché su animali da cortile, anche su animali di mole cospicua, come cavalli, bovini, suini, ovini e specialmente su animali da pelliccia, in cui sono state selezionate, su base genetica, moltissime razze i cui requisiti rispondono alle più svariate richieste commerciali.
In conclusione, si può affermare che i progressi della genetica applicata alle esigenze dell'agricoltura e della zootecnia hanno dato contributi di grande importanza all'economia della collettività.
2. Ingegneria genetica
a) Ricombinazione del DNA.
La variabilità genetica, cioè la possibilità di ottenere molteplici combinazioni di geni, è basata - oltreché sul processo di mutazione, che ne fornisce il primo elemento - sulla ricombinazione. Questa, nelle piante e negli animali uni- o pluricellulari, cioè negli eucarioti, è legata ai fenomeni sessuali in virtù dei quali in ogni individuo confluiscono patrimoni genetici provenienti da linee diverse. Lo scambio (crossing-over) che avviene durante la meiosi concede un certo grado di libertà ai geni che risiedono in uno stesso cromosoma, e consente quindi un rimescolamento in seno ai gruppi di associazione aumentando le possibilità di ricombinazione. È difficile, anzi tecnicamente impossibile, allo stato attuale delle conoscenze, intervenire direttamente ad alterare la costituzione genetica di un individuo, inserendovi artificialmente determinati geni, ossia segmenti di DNA. Il miglioramento genetico delle piante coltivate e degli animali d'allevamento, praticato dall'uomo fin da tempi antichi con metodi empirici e ulteriormente sviluppato su base scientifica nei tempi recenti, è basato esclusivamente sugli incroci e sulla successiva selezione, come esposto nel capitolo precedente.
La genetica dei microrganismi e gli sviluppi della biologia molecolare hanno consentito, invece, di operare alcuni interventi diretti: dal 1970 in poi si sono andate perfezionando alcune tecniche, alle quali è stato dato il nome di ingegneria genetica, o manipolazioni genetiche. Queste hanno destato vivo interesse fra i biologi e nel pubblico, nonché molta apprensione per i rischi che possono derivare dalla diffusione incontrollata di tali operazioni.
Le ricerche di genetica dei microrganismi hanno dimostrato che il corredo genetico dei Batteri non risiede tutto nell'unico cromosoma caratteristico della cellula batterica (procariote): esistono segmenti di DNA, i ‛plasmidi' (v. fig. 4), dotati della capacità di riprodursi, i quali possono trasmettersi indipendentemente dal cromosoma, oppure integrarsi con questo e rimanere ad esso uniti, per poi, eventualmente, individualizzarsi nuovamente in successive generazioni. Essi portano alcuni geni, i quali manifestano la propria attività nella cellula che li riceve. Inoltre i plasmidi, quando si integrano con il cromosoma batterico, possono appropriarsi di un pezzo del DNA del cromosoma della cellula che li ha ospitati e trasmettere i geni in esso localizzati ad altre cellule batteriche in cui penetrino (fenomeno cui si dà il nome di trasduzione). Si possono così trasmettere, per via extracromosomica, geni o gruppi di geni fra cellule di diversi ceppi della stessa specie, per esempio di Escherichia coli, o di specie diversa, per esempio di Staphylococcus aureus in E. coli.
Si ignora quale sia in natura la frequenza di questi fenomeni e l'importanza ch'essi hanno nell'evoluzione dei Batteri; ma la loro scoperta e la realizzazione sperimentale hanno consentito di spingere in profondità l'analisi di molti fenomeni genetici elementari. Pioniere delle ricerche in questo campo è stato soprattutto Stanley S. Cohen, della Stanford University School of Medicine (California), con vari collaboratori. Egli è riuscito anche a introdurre in E. coli geni provenienti da cellule di organismi eucarioti, per esempio dell'anfibio Xenopus laevis, i quali hanno funzionato inducendo il citoplasma della cellula batterica a costruire le proteine di cui essi portano l'informazione. A ciò si è arrivati confezionando plasmidi complessi, in cui siano incorporati geni estranei alla loro composizione naturale, grazie a elaborate tecniche biochimiche e genetiche, rese possibili dalla scoperta e dall'isolamento di diversi enzimi che intervengono nella rottura e nella ricomposizione della catena di DNA.
Un primo esempio di queste operazioni è dato dal trasferimento di plasmidi che portano un gene per la resistenza a un antibiotico (per es. la tetraciclina) in una cellula che ne sia priva e perciò sensibile (v. fig. 5). Il DNA del ceppo di E. coli naturalmente resistente viene estratto, riducendo le cellule in poltiglia, con rottura delle membrane cellulari. Alla poltiglia si aggiunge bromuro di etidio che, essendo assunto in misura differente dai cromosomi e dai plasmidi, aumenta la densità di questi ultimi e ne rende possibile la separazione per centrifugazione in cloruro di cesio. I plasmidi, così ottenuti allo stato puro, vengono portati a contatto di cellule di un ceppo sensibile previamente trattate con cloruro di calcio, che facilita la penetrazione dei plasmidi. Le cellule che incorporano il plasmidio diventano resistenti alla tetraciclina. Sono pochissime, dell'ordine di una su un milione; ma, immettendo nel brodo di coltura una soluzione di tetraciclina, si uccidono tutte le cellule batteriche che non hanno incorporato il plasmidio e non hanno acquisito la resistenza. Sopravvivono soltanto quelle resistenti, che si riproducono, e con esse si riproduce il plasmidio portatore del gene per la resistenza: si forma così un ceppo resistente a quell'antibiotico. Le cellule che hanno acquisito questa proprietà si dicono ‛cellule trasformate'. Con questo metodo si possono introdurre nelle cellule geni esistenti in natura in ceppi diversi della stessa specie (E. coli) o, come si è detto, in specie batteriche affini.
Operazioni più complicate consentono di manipolare i plasmidi introducendovi segmenti di DNA provenienti da plasmidi diversi, oppure da cellule di eucarioti.
I plasmidi (come il cromosoma batterico) sono formati da catene di DNA, che hanno struttura anulare, la quale è essenziale perché essi possano manifestare le loro proprietà biologiche. Per formare dei plasmidi compositi, occorre innanzitutto romperli e renderli lineari. La rottura si ottiene trattando le molecole anulari con particolari enzimi chiamati endonucleasi di restrizione, che sono prodotti da vari ceppi di E. coli, nonché da altri microrganismi e da virus. Le prime endonucleasi di restrizione ad essere isolate furono quelle prodotte dal batteriofago lambda, uno dei fagi parassiti di E. coli. Questi enzimi hanno la capacità di rompere la catena di DNA in determinati punti (diversi e ben definiti per ogni enzima). A questo stadio delle operazioni si possono mescolare segmenti lineari di DNA di provenienza diversa, che recano geni differenti (v. fig. 6). Si tratta ora di collegare fra loro questi segmenti e di ridare alla molecola complessa risultante la struttura anulare necessaria alla sua attività biologica. Questi risultati si ottengono trattando prima con un enzima, la transferasi terminale, che lega alle estremità delle catene i nucleotidi che vengano messi a disposizione nell'ambiente di coltura. Si possono così formare catene terminali di nucleotidi complementari, per esempio con la base adenina (A) a una estremità e rispettivamente timidina alla estremità opposta (T). In passi successivi, sempre per azione di enzimi specifici (exonucleasi, DNA-polimerasi, DNA-ligasi), si ottiene la saldatura delle due estremità della catena e così si forma una molecola di DNA composita, di forma anulare, che può essere immessa nelle cellule batteriche e funzionare come un plasmidio, cioè trasformarle, conferendo loro le proprietà dei geni che risiedono sulla molecola di DNA.
La produzione di molecole circolari di DNA può avvenire più semplicemente in un solo passo trattando sia il plasmidio originario, sia la catena di DNA che a esso si vuol legare con una endonucleasi di restrizione particolare, isolata da E. coli (EcoRI), la quale rompe la molecola di DNA in determinati punti, in modo che le estremità sono già dotate dei nucleotidi complementari, sono già rese ‛adesive', così che la riunione e l'‛anellamento' avvengono spontaneamente.
Le operazioni che abbiamo molto sommariamente descritto (per i particolari si rimanda alle opere indicate in bibliografia) possono essere ricapitolate in una sequenza logica. Il primo stadio è l'isolamento dei geni (provenienti da plasmidi, o da molecole di DNA di cellule di eucarioti, ridotte in segmenti), che si può fare utilizzando gli enzimi di restrizione, oppure con altri metodi. Fra questi potrà assumere notevole importanza,la sintesi di geni in vitro, come quella ottenuta da O. Khorana per i geni degli RNA di trasferimento (transfer RNA) della alanina, nel lievito, e della tirosina in E. coli. Il secondo stadio consiste nella formazione delle terminazioni delle catene di DNA in modo tale che possano congiungersi, e ciò può ottenersi con la transferasi terminale, oppure direttamente con alcuni enzimi di restrizione.
Terzo stadio è la combinazione del segmento di DNA che si vuole introdurre con un vettore, cioè un DNA capace di penetrare nella cellula batterica e di riprodursi. Occorre poi ottenere la ricongiunzione delle estremità della molecola composita, in modo da farle assumere la forma anulare, e ciò si ottiene o direttamente quando i due segmenti sono ottenuti usando uno stesso enzima di restrizione, oppure incubando le due molecole insieme con l'enzima DNA polimerasi e infine, per ottenere la saldatura completa dell'anello, con l'enzima DNA-ligasi (v. fig. 7). Il quarto e ultimo stadio consiste nel far penetrare il DNA composito nella cellula. Questo può farsi direttamente, in E. coli, se la molecola di DNA è stata legata a un vettore adeguato, cioè a un plasmidio, che è capace di riprodursi e di fare riprodurre anche la catena molecolare a lui legata, ove questa, come spesso accade, sia sprovvista della facoltà di autoriproduzione. Oppure si può legare il DNA ricombinato a un virus lisogeno, il quale penetra nel batterio e si integra con il cromosoma batterico.
Con questi procedimenti si possono dunque introdurre in cellule di E. coli geni provenienti da altre cellule batteriche. Per assicurarsi che ciò sia avvenuto, è necessario, ovviamente, potere riconoscere l'attività dei geni estranei (cioè non presenti nella cellula ospite), come, per esempio, nel caso che abbiamo illustrato, la resistenza a uno specifico antibiotico: occorre cioè che il segmento di DNA introdotto porti dei geni marcatori facilmente individuabili.
Come si è detto, Cohen e collaboratori sono riusciti anche a legare a piasmidi e a introdurre in E. coli segmenti di DNA di cellule di animali, in particolare dell'anfibio Xenopus laevis. Ciò si ottiene frammentando artificialmente il DNA che si ricava da queste cellule, legandone i segmenti con un plasmidio portatore del gene per la resistenza alla tetraciclina (gene marcatore), ottenendo una discendenza (un cione) da questi batteri, ed esaminando poi, con metodi opportuni, se determinati geni dello Xenopus sono penetrati nel batterio e sono entrati in attività, producendo (tramite l'RNA messaggero) le proteine di cui recano l'informazione. Con questo metodo, Cohen e collaboratori sono riusciti a ottenere cloni di E. coli in cui sono presenti, e attivi, geni che controllano la formazione dei ribosomi in Xenopus. I risultati di queste manipolazioni genetiche sono di grande importanza teorica e pratica. L'importanza teorica è data, oltreché dalla possibilità di studiare metodi di ricombinazione che possono aver luogo naturalmente nei procarioti e di valutare la loro importanza per l'evoluzione, soprattutto dalla possibilità di utilizzare questi sistemi per analizzare l'attività dei geni provenienti da cellule di animali o di piante. I doni batterici in cui i geni artificialmente introdotti si riproducono forniscono grandi quantità dei geni in questione, che possono essere sottoposti ad analisi strutturali e funzionali. Con questo mezzo di amplificazione sono già stati studiati i geni ribosomici di Xenopus, di Drosophila, i geni degli istoni di riccio di mare, i geni delle globine di alcuni mammiferi. E prevedibile che nel prossimo futuro si possano indagare molti problemi d'interesse genetico ed embriologico, quali le condizioni che determinano l'entrata in attività di dati geni e l'inattività di altri durante i processi del differenziamento.
Dal punto di vista delle applicazioni pratiche queste tecniche di ingegneria genetica possono riuscire assai utili nella produzione in notevole quantità di ormoni, di enzimi e in genere di proteine di interesse biologico o medico che sia difficile o dispendioso produrre per via sintetica, o isolare dagli anirriali o dalle piante. Si sono già ottenuti notevoli risultati in alcuni casi e molte sono le speranze che si possono formulare in base alle ricerche sperimentali attualmente in corso in diversi laboratori. Le notizie che si hanno negli ultimi mesi del 1977 si riferiscono all'ottenimento di una pro-insulina umana (che fa sperare che si possa arrivare alla produzione di insulina) e di un altro ormone, la somatostatina. Quest'ultimo ottenuto costruendo artificialmente il gene relativo e inserendolo, con un piasmidio, in E. coli.
Tuttavia, se le possibilità teoriche di costruire linee cellulari capaci di operare sintesi biologiche economicamente importanti (come per es. fissazione dell'azoto atmosferico, costruzione in massa di proteine adatte all'alimentazione, ecc.) sono infinite, occorre tener conto di due fattori limitanti: 1) le difficoltà inerenti allo sviluppo di queste manipolazioni; 2) i rischi a cui si può andare incontro.
Per quanto riguarda il primo argomento, è da rilevare che, se è relativamente facile introdurre, con un vettore adatto, geni estranei nella cellula batterica, non è detto che questi geni manifestino sempre nel nuovo ambiente un'attività tale da dar luogo a una produzione quantitativamente soddisfacente. Inoltre, se è facile introdurre segmenti di DNA nei Batteri, non lo è altrettanto nelle cellule di eucarioti in coltura. Nelle cellule animali si possono utilizzare i virus oncogeni, che si integrano con il genoma dell'ospite. Nelle cellule vegetali praticamente l'operazione non è ancora riuscita. E probabile che queste difficoltà possano, in futuro, venire superate in vari modi; ma sarà necessario ancora molto lavoro.
Il secondo fattore di limitazione ha dato molto da pensare ai biologi e ha avuto notevoli ripercussioni nei pubblico. I rischi prevedibili sono: la creazione di ceppi di batteri patogeni resistenti agli agenti normalmente usati per combatterli; la formazione di batteri portatori di virus oncogeni; la formazione di nuove entità batteriche capaci di determinare nell'uomo e/o negli animali e nelle piante di interesse economico malattie finora sconosciute, di fronte alle quali l'uomo si troverebbe disarmato. Naturalmente questi pericoli sussistono quando i nuovi ceppi di batteri escano dai laboratori e si diffondano nell'ambiente; ma questa possibilità non è tanto remota, trattandosi di organismi la cui diffusione è difficilmente controllabile. Inoltre, poiché la massima parte degli esperimenti sono fatti su E. coli, batterio ubiquitario, ospite normalmente innocuo dell'intestino umano, la possibilità di infezione da parte di ceppi trasformati in senso patogeno non è certo da sottovalutare. Consci di questi pericoli, i microbiologi dediti a questi studi sono stati fin dall'inizio molto attenti a evitare fughe di batteri comunque trasformati e molto prudenti nel distribuirli ad altri laboratori che ne facessero richiesta. Allorché le ricerche si intensificarono e si estesero a diversi centri di studio, il pericolo apparve più imminente, e la Nationai Academy of Sciences degli Stati Uniti incaricò il microbiologo Paul Berg di costituire una commissione per esaminare la questione e formulare proposte per impedire una sperimentazione indiscriminata. Berg riunì nel 1974 numerosi biologi che avevano in corso ricerche di ingegneria genetica, i quali espressero in un comunicato alla stampa le loro apprensioni per le possibili conseguenze sfavorevoli che potevano risultare da un'incauta sperimentazione e chiesero ai ricercatori di astenersi per il momento dal fare due tipi di esperimenti, e cioè: 1) la costruzione di nuovi organismi aventi la capacità di produrre tossine o resistenza agli antibiotici e 2) l'introduzione di DNA proveniente da virus oncogeni nei Batteri, perché in tal modo le molecole ricombinate potevano essere largamente diffuse e potevano aumentare l'incidenza del cancro o di altre malattie nell'uomo. Questa sospensione, o moratorium, sarebbe durata il tempo necessario per poter valutare con maggiore precisione i rischi cui si andava incontro e predisporre le cautele necessarie a evitarli.
La proposta sollevò molto malumore e accese numerose polemiche, che trovarono ampia eco nella stampa, anche perché si temeva che volesse condurre a dare licenza per gli esperimenti pericolosi soltanto ad alcuni laboratori, il che poteva configurarsi come un monopolio concesso ad alcune scuole e non ad altre. Nel febbraio 1975 si tenne una conferenza ad Asilomar (California) alla quale parteciparono 83 biologi americani e 53 provenienti da altre 16 nazioni, nonché studiosi di scienze giuridiche e morali e i rappresentanti delle organizzazioni che finanziano le ricerche scientifiche. Il risultato della conferenza fu innanzitutto il riconoscimento dell'importanza delle ricerche dal punto di vista delle applicazioni pratiche nel campo biomedico ed economico. I rischi potenziali possono essere di vario grado, quindi le cautele da usare devono esser proporzionate ai possibili pericoli e, ove questi non siano conosciuti con sicurezza, i mezzi protettivi per impedire la diffusione dei Batteri devono essere di livello molto elevato. In sostanza, ad Asilomar, come anche a una conferenza tenuta l'anno successivo a Parigi, prevalse il criterio di non porre limiti alla sperimentazione e alla fantasia dei singoli ricercatori, purché questi operino con la massima cautela per impedire fughe degli organismi costruiti artificialmente in laboratorio. Fu proposta una classificazione dei vari tipi di esperimenti secondo la gravità del rischio potenziale che ne deriva e per ciascuna categoria furono indicate le cautele che devono essere messe in opera dai ricercatori.
Altri pericoli sono stati figurati dalla fantasia dei profani: si è ritenuto che si realizzasse la possibilità di alterare la costituzione genetica degli individui umani, cosa che non mancherebbe di sollevare gravissimi problemi d'ordine morale e giuridico. A questo proposito bisogna distinguere due tipi di interventi teoricamente possibili: 1) sostituire nelle cellule somatiche geni difettosi, che producono determinate malattie, con geni ‛sani'. In tal modo si agirebbe in sede terapeutica, curando alcune malattie genetiche, per le quali non si dispone di terapia adeguata. Si opererebbe cioè in modo analogo a quanto abitualmente si fa per curare le malattie in cui basta somministrare al paziente i prodotti che il gene ‛malato' è incapace di formare (per es. insulina ai diabetici), ma, invece di somministrare la sostanza, si fornisce il gene che ne controlla la produzione; 2) sostituire geni nelle cellule germinali, cioè alterare la costituzione genetica di un individuo e dei suoi discendenti. Ambedue queste operazioni sono per il momento irrealizzabili e, soprattutto per la seconda, non si vede neanche quale potrebbe essere una via per renderla tecnicamente possibile.
Si conclude pertanto che i pericoli dell'ingegneria genetica sono molto meno gravi di quanto è stato prospettato in certi ambienti e non vi è alcuna seria ragione per impedire la prosecuzione degli esperimenti, purché siano rigorosamente osservate le cautele che sono raccomandate dalle periodiche riunioni internazionali dei biologi addetti a questi lavori.
b) Ibridazione di cellule somatiche
Un altro mezzo per manipolare il corredo genetico è l'ibridazione di cellule somatiche coltivate in vitro. La coltivazione dei tessuti è una tecnica che si è sviluppata agli inizi di questo secolo e ha fornito un mezzo di indagine molto efficace per lo studio di vari problemi. Con lo sviluppo della genetica, e soprattutto della genetica biochimica e molecolare, si è ravvisata la necessità di coltivare, anziché dei tessuti, delle cellule: costituire cioè linee cellulari in cui i singoli elementi non si uniscono a formare masse più o meno analoghe ai tessuti, ma rimangono separati e sono analizzabili singolarmente. Con questa tecnica si sono ottenuti importanti risultati, sia per quanto riguarda la cariologia, cioè lo studio dell'assetto cromosomico, sia per l'analisi delle capacità biochimiche delle singole cellule.
Nel 1960 G. Barski, S. Sorieul e F. Cornefert osservarono che le cellule di due linee tumorali diverse di topo possono fondersi a due a due, dando origine a ibridi cellulari. Negli anni successivi (1962 e seguenti) B. Ephrussi e collaboratori diederono grande impulso a questi studi, perché compresero che la tecnica dell'ibridazione delle cellule somatiche poteva riuscire assai utile nelle indagini relative al differenziamento cellulare. Nel 1964 J. Littlefield dimostrò che è possibile applicare alle cellule coltivate in vitro i procedimenti selettivi usati dai microbiologi per l'isolamento di rari prodotti di ricombinazione e complementazione genica. Se si tratta una coltura con sostanze capaci di uccidere le cellule che non posseggano una determinata combinazione genica, è chiaro che sopravvivono soltanto quelle che ditale combinazione sono provviste. In tal modo, variando le sostanze (per lo più coinvolte nella sintesi di enzimi importanti per la vita della cellula), si possono studiare gli effetti della complementazione genica e si può riconoscere quali cellule posseggono i geni che controllano determinate funzioni enzimatiche.
L'esempio classico del sistema di Littlefield è dato da due ceppi cellulari deficienti rispettivamente per due enzimi coinvolti nella sintesi degli acidi nucleici: la ipoxantinguaninfosforibosiltransferasi (HGPRT) e la timidinacinasi (TK). Le cellule deficienti per l'uno o l'altro dei due enzimi, se coltivate in presenza di amminopterina, che inibisce la sintesi ex novo degli acidi nucleici, sono incapaci di riprodursi e muoiono. Con la fusione di una cellula deficiente per HGPRT, e normale per TK, e di una deficiente per TK e normale per HGPRT, si ottiene un ibrido che contiene ambedue gli alleli attivi. Queste cellule sono le sole capaci di sopravvivere e di riprodursi in presenza del mezzo selettivo contenente amminopterina, laddove le cellule parentali soccombono.
Con la fusione di due cellule si forma una cellula binucleata: se i due nuclei sono eguali si parla di homocaryon, se sono diversi in quanto provenienti da ceppi diversi della spessa specie, o da specie diverse, di heterocaryon. Per lo più avviene anche la fusione dei nuclei, per cui l'ibrido possiede un genoma complesso, poliploide.
La possibilità di introdurre nelle cellule geni provenienti da ceppi diversi, indipendentemente dai fenomeni sessuali, offre molte opportunità di indagine. Particolarmente interessanti sono gli ibridi di cellule provenienti da specie diverse, per esempio uomo-topo, uomo-ratto, ecc. Si sono ottenute fusioni di cellule appartenenti a specie sistematicamente assai lontane, financo di cellule animali con cellule vegetali. In generale si osserva che il cariotipo, cioè l'assetto cromosomico dell'ibrido, nel corso di poche generazioni cellulari va incontro a rapide modificazioni, per lo più dovute alla eliminazione di cromosomi. Così, per esempio, nell'ibrido cellulare uomo-topo, le cellule perdono man mano la maggior parte dei cromosomi umani; mentre nell'ibrido uomo-ratto i cromosomi che vengono in gran parte eliminati sono quelli del ratto. Le cause di questi fenomeni sono sconosciute.
La fusione cellulare che ha luogo quando si mescolano le colture di due ceppi cellulari diversi è però un fatto che si verifica spontaneamente con frequenza assai bassa, dell'ordine da 1 a 19 su un milione. Ma H. Harris e J. F. Watkins (1965) basandoni su osservazioni di Y. Okada (1958) riuscirono a indurre la fusione con alta frequenza trattando le cellule con un virus parainfluenzale (virus Sendai, indicato convenzionalmente con la sigla HVJ), inattivato con raggi ultravioletti. In seguito si trovarono alcune sostanze (la più efficiente è il glicolpolietilene) che determinano rapidamente la fusione, con alta frequenza. Evidentemente i virus e alcune sostanze chimiche provocano modificazioni della membrana cellulare, che facilitano la coesione e la fusione delle cellule.
Operazioni che danno risultati analoghi a quelli della ibridazione cellulare sono l'introduzione nella cellula di un nucleo intero, ottenuto da T. Ege, N. R. Ringerts e collaboratori (1974), o di cromosomi metafasici (O. W. McBride e H. L. Ozer, 1973).
L'ibridazione cellulare e le operazioni suddette rientrano nell'ambito dell'ingegneria genetica o manipolazione del genoma, in quanto il loro effetto è di modificare a volontà dello sperimentatore il patrimonio genetico senza ricorrere al mezzo naturale, rappresentato dalla fecondazione. Esse costituiscono uno strumento di analisi di grande importanza, che ha già dato risultati notevolissimi in vari campi.
Il risultato più importante, dal punto di vista della genetica formale, è la localizzazione di molti geni sui cromosomi umani. Ciò si ottiene, per i geni che controllano la formazione di determinate proteine indispensabili alla vita della cellula, applicando metodi selettivi che uccidono tutte le cellule, tranne quelle che possiedono quel determinato gene. Se nell'ibrido uomo-topo (in cui le cellule del topo siano prive dell'allele in questione) si vede che sopravvivono soltanto le cellule in cui tutti i cromosomi umani sono stati eliminati, tranne uno, si può concludere che il gene studiato è localizzato in quel cromosoma. Grazie a questi metodi, nel giro di pochi anni sono già stati localizzati più di un centinaio di geni sui 23 cromosomi umani ed è prevedibile che nel prossimo futuro si giunga a costruire una mappa genetica di tali cromosomi analoga a quelle che, con il metodo tradizionale degli incroci (difficilmente applicabile all'uomo, in cui non si possono attuare incroci a volontà dello sperimentatore), sono state stabilite per alcuni animali e piante. Questo risultato è di grande importanza anche ai fini delle genetica medica (v. eredità biologica).
L'ibridazione delle cellule somatiche può servire inoltre allo studio dei meccanismi che intervengono durante la mitosi per eliminare dal cariotipo alcuni cromosomi e mantenerne altri, nonché allo studio del modo con cui i vari geni esprimono le proprie capacità o sono resi silenti da processi di repressione. Lo studio dei processi di regolazione dell'attività dei geni è la base delle indagini sul differenziamento cellulare.
Infine questa tecnica serve anche allo studio della oncogenesi e delle relazioni fra i virus e il cancro. Infatti, con l'introduzione di alcuni virus, si possono trasformare cellule normali in cellule cancerigene; mediante l'ibridazione si può stabilire su quali cromosomi dell'uomo si attaccano i virus oncogeni e si possono così analizzare le variazioni genetiche che si verificano in una cellula trasformata.
L'ibridazione cellulare è duque una tecnica di singolare efficienza per lo studio di molti problemi di genetica, di embriologia e di patologia. Contrariamente a un'opinione che è stata espressa da persone non bene informate, essa non presenta, allo stato attuale, alcun rischio e non dà adito a problemi etici o giuridici.
(Per quanto riguarda le applicazioni all'uomo, che hanno dato origine allo sviluppo di discipline speciali quali la genetica umana, la genetica medica, la immunogenetica, si rimanda alle precedenti partizioni di questo articolo, nonché agli articoli biometria, eredità biologica, immunologia e immunopatologia, sangue, sesso, trapianti).
Bibliografia
AA. VV., Recombinant molecules: impact on science and society, New York 1977.
Bernhard, H. P., The control of gene expression in somatic cell hybrids, in ‟International review of cytology", 1976, XLVII, pp. 289-325.
Cohen, St., S., La manipolazione dei geni, in ‟Le scienze", 1975, XV, n. 87, pp. 28 ss.
Davidson, R. L., Gene expression in somatic cell hybrids, in ‟Annual review of genetics", 1974, VIII, p. 195-218.
Davidson, R. L., de la Cruz, F. F. (a cura di), Somatic cell hybridization, New York 1974.
Duvigneaud, P., La synthèse écologique, Paris 1974.
Freeden, H. T., Animal breeding today: its dimensions and accomplishments, in ‟Canadian journal of genetics and cytology", 1977, XIX, pp. 193-210.
Hayes, H. K., Immert, F. R., Smith, D. C., Methods of plant breeding, New York 19552.
Lederberg, J., DNA research: uncertain peril and certain promise, in ‟Prism", June 15, 1975.
Lerner, I. M., The genetic basis of selection, New York 1958.
Lerner, I. M., Heredity, evolution and society, San Francisco 1968 (tr. it. Eredità, evoluzione, società, Milano 1972).
Lush, J. L., Animal breeding plans, Ames, Iowa, 1937.
Maddox, J., Prospettive della manipolazione genetica, in Annuario della enciclopedia di scienza e tecnica, Milano 1976, pp. 324-328.
Müntzing, A., Genetics: basic and applied, Stockholm 19672.
Murray, K., Biochemical manipulation of genes, in ‟Endeavour", 1976, XXXV, p. 29.
Rogers, M., Biohazard, New York 1977.
Sgaramella, V., Ingegneria genetica, Padova 1975.
Sgaramella, V., Realtà, psicosi e mito nell'ingegneria genetica, in La cultura scientifica nel mondo contemporaneo (a cura della rivista ‟Scientia"), Milano (in stampa).
Singer, M. F., The recombinant DNA debate, in ‟Science", 1977, CLXXXXVI, p. 127.
Siniscalco, M., Ibridi cellulari somatici, in ‟Atti dell'Associazione Genetica Italiana", 1970, XV, pp. 3-71.
Siniscalco, M., Somatic cell hybrids as tools for genetic studies in man, in ‟Symposia of the International Society for Cell Biology", 1970, IX, pp. 205-231.
Terzi, M., Genetics and the animal cells, New York 1974.
Wade, N., Gene splicing: Congress starts framing law for research, in ‟Science", 1977, CLXXXXVI, pp. 39-40.
Wade, N., Rational containment on recombinant DNA, in ‟Nature", 1977, CCLXVIII, p. 91.
Wade, N., Recombinant DNA debate three years on, in ‟Nature", 1977, CCLXVIII, p. 185.
Wade, N., The ultimate experiment: man-made evolution, New York 1977.