RNA. Piccoli RNA
RNA. Piccoli RNA
Fino agli anni Settanta del XX sec. le funzioni biologiche note dell'acido ribonucleico (RNA) erano ristrette alla funzione codificante (RNA messaggero) e alla sintesi proteica (RNA ribosomale e RNA transfer) e l'idea prevalente era che l'RNA svolgesse un ruolo minimo nella regolazione dell'espressione dei geni. Tuttavia, nelle decadi successive, è diventato evidente che l'RNA è coinvolto in tutti gli aspetti che riguardano il mantenimento, il trasferimento, il processamento e la regolazione dell'informazione genetica. È stato dimostrato che nella cellula esistono una varietà incredibile di molecole di RNA e che l'RNA può svolgere funzioni enzimatiche, dirigere la rimozione degli introni dai trascritti nucleari precursori dell'RNA messaggero, dirigere il processamento dell'RNA precursore degli RNA ribosomali, funzionare come guida per gli enzimi che modificano il precursore dell'RNA ribosomale o altri piccoli RNA e controllare l'espressione genica sia a livello trascrizionale sia a livello post-trascrizionale. Tutte queste funzioni dell'RNA sono possibili per le sue caratteristiche: l'RNA è una biomolecola altamente reattiva con proprietà chimiche uniche, può formare strutture secondarie e terziarie estremamente complesse e riconoscere in maniera specifica certe sequenze complementari presenti in altre molecole di RNA.
Negli organismi eucarioti l'RNA comprende una vasta gamma di molecole che in generale sono classificate a seconda della loro funzione, unità di sedimentazione o dimensioni e localizzazione nella cellula. L'espressione 'piccoli RNA' fu originalmente introdotta per indicare molecole di RNA non codificanti di lunghezza compresa fra 50 e 300 nucleotidi. Piccoli RNA di questa categoria sono presenti sia nel nucleo (snRNA, Small nuclear RNA) sia nel citoplasma (scRNA, Small cytoplasmic RNA) e in associazione con proteine specifiche formano le cosiddette 'piccole ribonucleoproteine' (RNP) nucleari (snRNP) o citoplasmatiche (scRNP). A loro volta, le RNP nucleari sono divise in diverse classi a seconda del subcompartimento nucleare in cui sono localizzate. Quelle che si trovano nel nucleoplasma, le snRNP propriamente dette, funzionano per la maggior parte nel processamento dell'RNA messaggero. Un gruppo diverso di snRNP, chiamate 'piccole ribonucleoproteine nucleolari' (snoRNP, Small nucleolar RNP), risiedono nel nucleolo, il compartimento dove avvengono la sintesi, la maturazione e l'assemblaggio delle sottounità del ribosoma. Qui sono coinvolte nei tagli endonucleolitici e nell'introduzione di modificazioni covalenti durante il processo di maturazione del precursore degli RNA ribosomali. Una terza classe di RNP, le piccole RNP dei corpi di Cajal (scaRNP, Small Cajal RNP), risiedono nei corpi di Cajal, regioni specializzate del nucleo caratterizzate dalla presenza della proteina coilin e che funzionano da stazioni di maturazione per le snRNP e le snoRNP. Nel nucleo delle cellule eucariotiche di organismi superiori ci sono milioni di RNP contenenti piccoli RNA, l'abbondanza delle diverse specie varia da circa 103 (scaRNP) a più di 106 (snRNP dello spliceosoma) per nucleo.
Alla fine degli anni Novanta del XX sec., la scoperta dell'interferenza genetica mediata da RNA a doppio filamento (dsRNA) ha portato all'identificazione di nuove classi di piccoli RNA; essi hanno una lunghezza compresa fra i 20 e i 30 nucleotidi e costituiscono quindi i più corti RNA conosciuti negli organismi eucarioti. Tali piccoli RNA sono denominati 'piccoli RNA interferenti' (siRNA, Small interfering RNA) e 'piccoli RNA microscopici' (miRNA, MicroRNA). In generale questi piccoli RNA riconoscono per complementarità sequenze omologhe presenti in RNA messaggeri (o in altri RNA) e servono come guida per indurre la degradazione dell'RNA bersaglio o per bloccare il processo di sintesi proteica.
La scoperta della interferenza genica mediata da questi piccoli RNA ha generato una completa rivoluzione della concezione di come l'espressione dei geni sia regolata negli organismi eucarioti. Oggi si calcola che nell'uomo più di un terzo dei geni siano regolati attraverso i miRNA e, probabilmente, il numero dei geni regolati è ancora più alto. È chiaro che, anche se sono piccoli, questi RNA hanno sufficiente specificità per funzionare con accuratezza. Non è esagerato affermare che non c'è campo della biologia che non sia stato profondamente cambiato dalla scoperta dei siRNA e dei miRNA.
Piccoli RNA nucleari e citoplasmatici
Piccoli RNA nucleari
Funzionano, insieme a miriadi di proteine, come fattori essenziali dei processi di maturazione degli RNA precursori del messaggero (pre-mRNA) o trascritti primari. I pre-mRNA devono essere efficientemente e accuratamente processati da meccanismi specifici che producono le molecole di RNA messaggero maturo; fra questi meccanismi vi sono la rimozione degli introni e il legame degli esoni, o splicing, e il processamento al terminale 3′ OH mediante poliadenilazione. Un notevole numero di snRNA partecipa al processo di splicing degli introni convenzionali che cominciano con GU e terminano con AG, specificamente U1, U2, U4, U5 e U6 snRNA (uridine-rich small nuclear RNP). U11, U12, U4atac e U6atac snRNA, invece, sono la controparte di U1, U2, U4 e U6, e funzionano nella rimozione di una seconda e rara classe di introni che hanno siti di splicing non canonici. A eccezione di U6 e U6atac, tutti gli snRNA sopraddetti sono associati con un gruppo di proteine, chiamate Sm, che formano un anello composto da sette sottounità. Durante il processo di splicing gli U snRNA, in forma di RNP, prendono parte alle interazioni RNA-RNA che sono altamente dinamiche e complesse e che sono alla base della funzione dello spliceosoma (il complesso ribonucleoproteico dove avviene lo splicing). L'evidenza sperimentale attuale suggerisce che questi piccoli RNA nucleari formino il centro catalitico dello spliceosoma.
Nella maggior parte degli organismi il processo di splicing dei pre-mRNA normalmente avviene in cis, in altre parole gli introni e gli esoni sono trascritti in un'unica molecola. Invece, nel processo di splicing in trans, che è caratteristico dei tripanosomi (Protozoi), di Euglena (alga), dei Nematodi e Trematodi e di alcuni Cordati primitivi, l'esone al 5′ terminale del messaggero è derivato da un piccolo RNA chiamato 'spliced leader RNA' (RNA SL) che è trascritto indipendentemente dal pre-mRNA. L'RNA SL è un snRNA specializzato e si trova in forma di ribonucleoproteina con le proteine Sm, caratteristiche delle snRNP. Quindi il processo di trans-splicing lega esoni derivati da due diverse molecole di RNA. La conseguenza del trans-splicing è che, in organismi come per esempio i tripanosomi, tutti i messaggeri contengono la stessa sequenza SL, lunga 39 nucleotidi, al terminale 5′. I pre-mRNA che sono processati mediante trans-splicing sono policistronici e la principale funzione del trans-splicing è di formare il 5′ terminale dell'RNA messaggero monocistronico maturo.
Un altro piccolo RNA nucleare degno di nota è U7 snRNA. Nelle cellule animali U7 è un cofattore essenziale per la formazione del terminale 3′ degli RNA messaggeri degli istoni che sono espressi solo durante la fase S del ciclo cellulare. Questi messaggeri non sono poliadenilati e il terminale 3′ viene formato tagliando con una endonucleasi il pre-mRNA. Una porzione al 5′ di U7 si lega a sequenze complementari localizzate a distanza fissa a valle del sito di taglio e questa interazione specifica la posizione del legame fosfodiesterico da scindere. In aggiunta agli snRNA che funzionano nel processamento del pre-mRNA, ce ne sono alcuni che funzionano durante il processo di trascrizione. Fra questi è l'snRNA 7SK, un componente di un fattore di trascrizione (P-TEBb) che regola in maniera positiva la fase di elongazione di RNA trascritti dalla polimerasi II.
Piccoli RNA nucleolari
Sono coinvolti: (a) nei tagli endonucleolitici che avvengono durante il processo di maturazione del precursore degli RNA ribosomali o pre-rRNA (U3, U14 e U8 sono esempi di snoRNA delle cellule animali che partecipano al taglio del pre-rRNA); (b) nella introduzione di modificazioni covalenti in posizioni specifiche degli RNA ribosomali o anche di alcuni snRNA, come per esempio U6 snRNA. Le più comuni e abbondanti modificazioni covalenti dell'RNA sono la metilazione della posizione 2′-OH del ribosio e la conversione di uridina a pseudouridina. Nell'RNA ribosomale di mammifero più di duecento nucleotidi sono modificati, circa la metà sono 2′-O-metilazioni e il resto sono pseudouridine. In tutti gli eucarioti e anche negli archeobatteri queste modificazioni sono guidate da snoRNA, i quali identificano il nucleotide specifico da modificare mediante la formazione di una regione a doppio filamento con sequenze complementari presenti nell'RNA bersaglio. Sia la 2′-O-metilazione che la conversione a pseudouridina sono catalizzate da enzimi specifici che, insieme ad altre proteine, formano complessi stabili con gli snoRNA. È interessante notare che la grande maggioranza degli snoRNA sono codificati e processati da introni.
Piccoli RNA associati a corpi di Cajal
Come gli RNA ribosomali, anche gli U snRNA vanno incontro a numerose modificazioni post-trascrizionali che includono 2′-O-metilazioni e conversione di uridine a pseudouridine. A eccezione di U6 snRNA che viene modificato nel nucleolo, U1, U2, U4, U5 e U12 snRNA sono modificati in regioni specializzate del nucleo chiamate 'corpi di Cajal'. Questi sono caratterizzati dalla presenza della proteina coilin e funzionano nella maturazione degli snRNA. Gli scaRNA sono simili agli snoRNA e, come questi, sono per la maggior parte derivati dalla trascrizione e dal processamento di introni.
Piccoli RNA citoplasmatici
I maggiormente conosciuti sono l'RNA 5S, che è parte della sottounità 60S del ribosoma, l'RNA transfer, che funziona come adattatore fra gli amminoacidi e i codoni dell'RNA messaggero durante la sintesi proteica, e l'RNA 7SL, l'RNA componente della particella ribonucleoproteica chiamata SRP (Signal recognition particle), che è essenziale nel processo di importazione cotraduzionale di alcune proteine destinate a far parte delle membrane o a essere secrete.
La scoperta della interferenza genica o silenziamento genico a opera di piccoli RNA
Come succede spesso per le scoperte scientifiche rivoluzionarie, il concetto e la dimostrazione dell'interferenza genica mediata da piccoli RNA non sono risultati da un unico esperimento, ma si sono sviluppati attraverso molteplici osservazioni riportate dagli anni Novanta del XX sec. in poi da parecchi laboratori che utilizzavano organismi completamente diversi. Nel 1990 due gruppi avevano cercato di produrre piante di petunia con fiori di colore più viola del normale e, come strategia, ambedue i gruppi avevano scelto di introdurre copie transgeniche di un enzima che determina la formazione del pigmento. L'idea era molto semplice: più geni per l'enzima avrebbero prodotto più enzima ed era logico pensare che la quantità di pigmento sarebbe aumentata dando luogo a fiori viola scuro. Al contrario, molti dei fiori che si svilupparono dalle piante transgeniche avevano petali bianchi o screziati, e quindi senza o con poco pigmento. Questa osservazione e altri esperimenti dimostrarono che nella petunia, in presenza di copie transgeniche, la sintesi dell'enzima responsabile per la formazione del pigmento era stata seriamente repressa invece di essere aumentata. Questo fenomeno fu chiamato cosuppression, per indicare che l'espressione sia del gene endogeno sia delle copie transgeniche era stata simultaneamente repressa. Risultati simili furono descritti nel fungo Neurospora e il fenomeno fu battezzato quelling.
Durante gli anni che seguirono la maggior parte dei tentativi di esprimere geni sovrannumerari nelle piante fallì per il fenomeno di cosuppression. Inoltre, esperimenti chiave dimostrarono che esso si verificava solo quando sia la sequenza codificante (sense) che la sequenza non-codificante (antisense) di un gene venivano espresse contemporaneamente. A questo punto cominciò a prendere forma l'idea che la cosuppression dipendesse dalla formazione di dsRNA, il funzionamento dei quali non era stato ancora chiarito. Mentre i primi esperimenti nelle piante procedevano, molti ricercatori cercavano di reprimere l'espressione genica utilizzando la cosiddetta 'strategia antisense'. Anche questa era un'idea semplice: si pensava che l'espressione di sequenze antisense avrebbe ridotto l'espressione di geni specifici attraverso la complementarità con le sequenze sense dell'RNA messaggero e la conseguente formazione di RNA a doppio filamento che, probabilmente, venivano degradati o non potevano essere utilizzati per la traduzione. Gli esperimenti antisense a volte funzionavano, ma per la maggior parte fallivano. A tali osservazioni si aggiunse, nel 1995, la pubblicazione di un lavoro che dimostrava come l'espressione genica in Caenorhabditis elegans poteva essere repressa mediante iniezione di molecole sintetiche di RNA o di tipo sense o antisense.
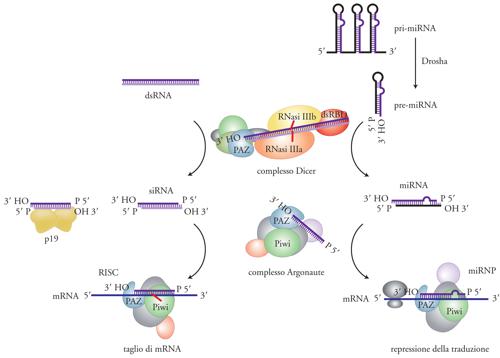
Dobbiamo ai laboratori di Andrew Fire e Craig Mello la comprensione del fatto che tutte le preparazioni di molecole di RNA a singolo filamento (che si usano comunemente in laboratorio) sono contaminate da basse concentrazioni di dsRNA e la fondamentale scoperta che sono le molecole di dsRNA a iniziare il processo di silenziamento genico. Il fenomeno fu battezzato 'RNA interference' (RNAi) ed è il meccanismo attraverso il quale molecole di dsRNA omologhe a RNA messaggeri specifici inducono la degradazione dell'RNA messaggero bersaglio (fig. 1). In seguito al lavoro di Fire e Mello, il silenziamento dell'espressione genica mediata da dsRNA fu scoperto e utilizzato sperimentalmente in un ampio spettro di organismi eucariotici, dai Protozoi all'uomo. L'evidenza che piccoli RNA interferenti, i siRNA, erano correlati con il fenomeno di cosuppression fu documentata per la prima volta nel 1999 da David Baulcombe e collaboratori nelle piante e, poco tempo dopo, il laboratorio di Thomas Tuschl dimostrò che i siRNA sintetici erano capaci di indurre RNAi in cellule di mammifero. Questa scoperta è stata fondamentale perché nei Mammiferi l'espressione o la trasfezione di dsRNA di lunghezza superiore a 30 nucleotidi provoca una generale distruzione di tutti gli RNA messaggeri e le cellule vanno poi incontro ad apoptosi. L'uso di siRNA sintetici ha permesso di applicare l'RNAi allo studio della funzione dei geni di mammifero in maniera rapida, specifica e su scala genomica. Tutti gli organismi in cui, come nei Mammiferi, i metodi di genetica classica sono di difficile applicazione, hanno tratto benefici enormi dalla scoperta dell'RNAi e dall'inizio del 2000 lo studio della funzione di geni, noti o sconosciuti, si è sviluppato in maniera esponenziale.
La scoperta degli RNA microscopici (miRNA) avvenne indipendentemente da quella dei siRNA e iniziò con l'identificazione da parte di Victor Ambros e collaboratori nel 1993 di un piccolo RNA di 22 nucleotidi, lin-4, che era necessario per il corretto sviluppo temporale delle larve del verme modello C. elegans. Fu proposto che lin-4 bloccasse, attraverso parziale complementarità con sequenze non codificanti al 3′ terminale, la traduzione dell'RNA messaggero per lin-14, una proteina la cui continua sintesi abrogava il corretto sviluppo temporale delle larve di C. elegans. Il successivo clonaggio di RNA piccoli derivati da una serie di animali e piante modello ha poi portato alla scoperta di centinaia di miRNA che sono espressi in vari organismi. Nel genoma umano si calcola che possano essere codificati circa 1000 miRNA, ma il loro numero è molto probabilmente destinato ad aumentare, visto che nella pianta modello Arabidopsis thaliana sembra siano espresse decine di migliaia di piccoli RNA.
siRNA e miRNA
I siRNA e i miRNA vengono distinti sulla base della loro origine e non della loro funzione. È importante notare che i miRNAs sono stati identificati solo in piante e animali e nei loro virus, ma non in organismi unicellulari. I siRNA sono derivati da dsRNA che possono essere lunghi centinaia o forse migliaia di basi. Nelle cellule di piante, di Nematodi e anche in organismi unicellulari queste lunghe molecole di dsRNA si possono formare come conseguenza della trascrizione di ambedue le eliche del DNA di famiglie di sequenze ripetute, come per esempio i trasposoni, i retroposoni e sequenze ripetute localizzate ai centromeri di cromosomi di certi funghi. Una volta sintetizzati questi RNA formerebbero dsRNA attraverso l'appaiamento di regioni di estensiva complementarità. Alternativamente, nei Nematodi, nelle piante e nei funghi il dsRNA può essere formato mediante copiatura di RNA a singolo filamento da parte di una specifica RNA-polimerasi chiamata RdRp (RNA-dependent RNA-polymerase).
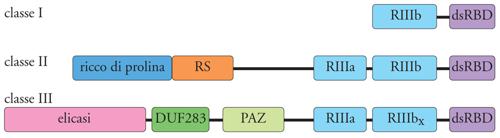
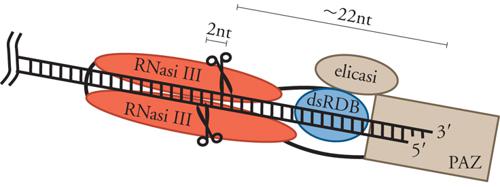
Lunghe molecole di dsRNA vengono poi tagliate progressivamente, a cominciare da una delle due estremità, da una ribonucleasi, chiamata Dicer, che appartiene alla famiglia delle ribonucleasi di tipo III (fig. 2). Dicer è una proteina con parecchi domini strutturali, fra i quali sono di particolare rilevanza due domini RNasi III presenti al terminale carbossilico. A differenza della ribonucleasi III batterica, che è un dimero di due sottounità identiche, Dicer funziona come monomero e i due domini RNasi III della molecola dimerizzano per formare il sito attivo dell'enzima. A seconda dell'organismo, Dicer produce frammenti a doppio filamento di lunghezza variabile fra i 21 e i 30 nucleotidi. Molto probabilmente la variabilità della lunghezza dei siRNA è dovuta a differenze strutturali fra gli enzimi Dicer dei vari organismi. Si pensa che Dicer funzioni come un ruler molecolare e misuri la distanza fra il sito di legame di una delle due estremità del dsRNA e il sito attivo dell'enzima. Ad ambedue le estremità dei siRNA sono presenti due nucleotidi non appaiati, una caratteristica comune a tutti i prodotti di digestione delle RNasi III (fig. 3). Nell'accezione odierna i siRNA sono o perfettamente o estensivamente complementari a sequenze dei trascritti bersaglio e ne guidano la specifica degradazione.
I siRNA possono inibire l'espressione genica a livello post-trascrizionale e anche a livello della trascrizione. Infatti, siRNA corrispondenti a trasposoni e sequenze ripetute sono coinvolti in certi processi di formazione di eterocromatina, e quindi nella formazione di regioni di DNA con una struttura non compatibile con la trascrizione. Il silenziamento del DNA attraverso la formazione di eterocromatina ha conseguenze fisiologiche molto importanti. Per esempio, i trasposoni e i retroposoni, che sono frequenti nel genoma degli eucarioti, se fossero trascritti in modo indiscriminato potrebbero saltare da un sito genomico a un altro provocando mutazioni e instabilità su scala genomica. Una delle importanti funzioni dell'RNAi è quella di prevenire la trascrizione di questi elementi mobili mediante formazione di eterocromatina o di indurre la degradazione dei corrispondenti trascritti attraverso i siRNA. Nelle piante, l'RNAi funziona anche come sistema di protezione contro infezioni virali.
I miRNA derivano da lunghi transcritti primari (Primary-miRNA, pri-miRNA), sintetizzati dall'RNA-polimerasi II, contenenti numerose regioni con strutture secondarie a forcella (hairpin) di circa 70 nucleotidi. Negli animali questi hairpin sono liberati dal pri-miRNA da una endonucleasi nucleare di tipo RNAsi III, chiamata Drosha, e i prodotti di questa digestione sono chiamati 'pre-miRNA'. I pre-miRNA vengono esportati dal nucleo nel citoplasma dove vanno incontro a un secondo step di maturazione da parte dell'enzima Dicer. Dicer taglia da ambedue le parti della doppia elica dell'hairpin producendo il miRNA maturo. Dal punto di vista funzionale i miRNA possono essere parzialmente o completamente complementari a sequenze presenti negli RNA messaggeri bersaglio. L'assoluta complementarità fra il messaggero e i miRNA attiva il processo di degradazione del messaggero bersaglio. Questo è il caso delle piante in cui tutti i miRNA conosciuti sono perfettamente complementari dei loro rispettivi RNA messaggeri. Invece negli animali i miRNA sono solo parzialmente complementari dei loro rispettivi RNA bersaglio e reprimono l'espressione genica a livello della traduzione, probabilmente bloccando il processo di inizio. È interessante notare che i messaggeri bersaglio, una volta riconosciuti dall'apparato di silenziamento, vengono associati a un complesso di proteine citoplasmatiche (Processing bodies, p-bodies) la cui funzione è la degradazione degli RNA messaggeri. Tuttavia, al momento non è completamente chiaro se l'inibizione della traduzione da parte dell'apparato di silenziamento genico associato ai miRNA sia la causa o l'effetto del trasferimento dei messaggeri bersaglio ai p-bodies.
La maggior parte dei messaggeri che codificano per proteine strutturali o coinvolte in processi metabolici di base non ha potenziali siti di legame per i miRNA. Al contrario, siti di legame per miRNA sono presenti in RNA messaggeri che codificano per proteine di regolazione, come per esempio fattori di trascrizione. È importante notare che alcuni RNA messaggeri hanno la potenzialità di legare più di un tipo di miRNA. Questa possibilità ha stimolato l'idea che la regolazione mediata dai miRNA possa avvenire in maniera combinatoria, quindi aumentando in maniera molto significativa la potenzialità della regolazione dell'espressione genica mediata da questi piccoli RNA. Tuttavia, la classe prevalente di RNA messaggeri che sono potenzialmente regolati dai miRNA sembra possedere un solo sito di riconoscimento.
La funzione dei miRNA
Si stima che i geni codificanti per i miRNA rappresentino dall'1 al 5% circa dei geni degli animali e, quindi, molto probabilmente costituiscano la classe più abbondante di geni dedicati alla regolazione dell'espressione di altri geni. A paragone nel genoma umano ci sono circa 1700 geni che codificano per fattori di regolazione della trascrizione. L'importanza della regolazione genica mediata dai miRNA è sottolineata dal fatto che la sequenza di alcuni miRNA è stata conservata attraverso l'evoluzione dai Nematodi all'uomo e dal ruolo essenziale che questi piccoli RNA svolgono in processi biologici di base, come lo sviluppo embrionale, la proliferazione cellulare, l'apoptosi, il metabolismo dell'insulina, la differenziazione cellulare e la morfogenesi. Quando i miRNA furono scoperti e il loro ruolo di regolatori genici fu riconosciuto in diversi sistemi modello, l'idea più accettata era che questi piccoli RNA fossero principalmente coinvolti nella regolazione dello sviluppo degli animali e funzionassero come 'interruttori' centrali dello sviluppo embrionale. Tuttavia, questa iniziale generalizzazione della funzione dei miRNA molto probabilmente non è del tutto corretta. Prima di tutto, per funzionare come repressori è sufficiente che i miRNA riconoscano solo sette nucleotidi nel messaggero bersaglio. Questa incredibile flessibilità di riconoscimento ha come conseguenza il fatto che un singolo miRNA può regolare l'espressione di molti messaggeri contemporaneamente, cosa che è stata verificata sperimentalmente. In aggiunta, embrioni del modello pesce zebra, in cui l'enzima Dicer e quindi tutti i miRNA sono assenti, mostrano difetti di sviluppo a cominciare dal processo di gastrulazione. Quindi, la funzione dei miRNA non sembrerebbe essere necessaria all'inizio dello sviluppo embrionale. Una possibilità alternativa è che i miRNA conferiscano precisione ai programmi di espressione genica durante il differenziamento cellulare e la formazione dei tessuti.
La biochimica del silenziamento genico mediato da piccoli RNA
Il meccanismo più chiaro dal punto di vista biochimico è il processo di degradazione degli RNA messaggeri mediato dai siRNA (o dai miRNA perfettamente complementari dell'RNA bersaglio). Questo processo è stato ricostruito in vitro usando estratti di cellule in coltura o di embrioni di drosofila. Una volta che i siRNA sono stati processati da Dicer, i siRNA vengono trasferiti alla proteina chiamata Argonaute2 (Ago2). È chiaro che sia i siRNA sia i miRNA cominciano la loro esistenza come molecole a doppio filamento, ma esercitano la loro funzione come molecole a singolo filamento. La trasformazione da doppio a singolo filamento avviene durante o probabilmente dopo il trasferimento di queste molecole ad Ago2, con l'aiuto di altre proteine che contengono domini di riconoscimento per dsRNA. Le regole che determinano quale delle due eliche dei siRNA sia incorporata nella proteina Ago2 sono di natura termodinamica e dipendono in gran parte dalla stabilità delle due estremità di queste molecole. L'elica scelta, che nel caso dei siRNA è chiamata 'elica guida', è normalmente quella in cui l'estremità 5′ del doppio filamento è meno stabile dal punto di vista energetico.
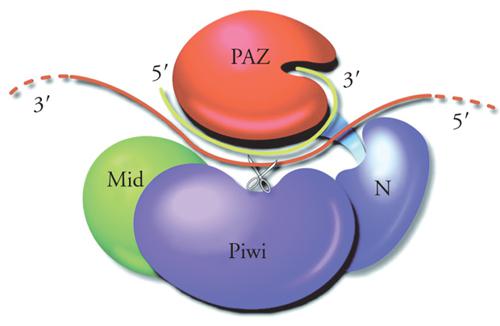
I membri della famiglia delle proteine Argonaute sono proteine basiche di circa 1000 amminoacidi e contengono due domini caratteristici: il dominio PAZ e il domino Piwi. PAZ, lungo circa 150 amminoacidi, si trova più o meno al centro della molecola e riconosce il 3′ OH del siRNA guida. Il dominio Piwi comprende circa 300 amminoacidi al terminale carbossilico e riconosce il terminale 5′ del siRNA guida. La proteina Ago2 dell'uomo e della drosofila (o Ago1 in A. thaliana) insieme ad altre proteine (il cui numero varia a seconda dei metodi di purificazione) forma un complesso multiproteico chiamato RISC (RNA-induced silencing complex) che possiede attività endonucleasica (anche chiamata slicer) ed è capace di degradare in maniera specifica un RNA bersaglio contenente sequenze complementari della sequenza guida del siRNA (fig. 4). È la proteina Ago2 che fornisce l'attività endonucleasica. L'attività endonucleolitica risiede nel dominio Piwi e, in particolare, in una regione di Piwi che ha omologia strutturale con gli enzimi della famiglia delle RNasi H, endonucleasi che classicamente digeriscono il filamento di RNA in un ibrido DNA/RNA.
Negli organismi superiori ci sono molteplici proteine Ago. Per esempio, nell'uomo sono stati identificati 8 membri della famiglia Ago, in A. thaliana 10 e in C. elegans più di 20. Tutti i membri della famiglia Ago analizzati fino a ora formano complessi ribonucleoproteici con un piccolo RNA e sono essenziali per vari processi di silenziamento genico. Tuttavia, mentre è stato verificato sperimentalmente che la proteina umana Ago2 (e le proteine omologhe ad Ago2 in altri organismi) ha attività endonucleasica, al momento non si conosce la precisa funzione enzimatica delle altre proteine della famiglia Ago, sia umane che di altri organismi. Per esempio, le proteine umane Ago1, Ago3 e Ago4 non sono in grado di digerire un RNA messaggero bersaglio, nonostante possano formare complessi con i miRNA o i siRNA complemetari al bersaglio. L'evidenza sperimentale accumulata fino a ora suggerisce che le diverse proteine Ago abbiano funzioni specializzate a seconda del tipo di cellula in cui sono espresse e del periodo di sviluppo dell'organismo.
Origine ed evoluzione della famiglia di proteine Argonaute
Mentre l'enzima Dicer è specifico per gli organismi eucariotici, le proteine della famiglia Ago sono presenti anche in una minoranza di organismi procariotici, sia archeobatteri che eubatteri, come per esempio l'archeobatterio Methanococcus jannaschii e l'eubatterio Aquifex aeolicus. Al contrario di quanto avviene per le proteine Ago2 degli eucarioti, endonucleasi che tagliano eliche RNA/RNA, la specificità delle proteine Ago batteriche non è al momento chiara. La presenza di proteine Ago soltanto in alcuni organismi procariotici rappresenta forse un altro esempio di trasmissione orizzontale genica fra eucarioti e procarioti. Alternativamente, è possibile che le proteine Ago abbiano origine batterica e si siano diversificate funzionalmente durante l'evoluzione degli organismi eucariotici.
Anche se al momento non è possibile determinare con certezza l'origine evolutiva della famiglia Argonaute, è chiaro che questa famiglia di proteine ha origini evolutive molto antiche essendo presente in Giardia intestinalis e Trypanosoma brucei, Protozoi che sono considerati discendenti di cellule eucariotiche primitive. I siRNA espressi in questi Protozoi sono tutti derivati da famiglie di retroposoni che sono localizzati per la maggior parte in regioni eterocromatiche del genoma, come per esempio i telomeri. Sulla base di queste osservazioni è molto probabile che il silenziamento di elementi genomici mobili rappresenti la funzione 'originale' del meccanismo di repressione genica mediato dai siRNA. La prevenzione della mobilizzazione di retroposoni, e anche di trasposoni, è essenziale per il mantenimento dell'integrità del genoma.
Nonostante le proteine Ago, e quindi i meccanismi di silenziamento mediati da piccoli RNA, siano presenti in tutti i rami dell'albero evolutivo eucariote, in alcuni organismi unicellulari non c'è traccia di Ago. Per esempio, nel genoma del fungo modello Saccharomyces cerevisiae il gene Ago non è stato trovato, mentre proteine Ago sono presenti nella maggior parte dei funghi. Osservazioni simili sono state fatte nei Protozoi della famiglia Trypanosomatidae (il gene Ago è presente solo nei tripanosomi africani, ma non nella Leishmania o nel Trypanosoma cruzi) e del phylum Apicomplexa (il gene Ago è presente nel parassita Toxoplasma gondii, ma non nelle varie specie di Plasmodium). La spiegazione più semplice è che il meccanismo di RNAi sia stato perso più volte durante l'evoluzione degli organismi eucariotici. Infatti, in condizioni di crescita in laboratorio, è stato possibile rimuovere il singolo gene Ago presente nel genoma dei funghi e dei tripanosomi o anche rimuovere certi membri della famiglia Ago del C. elegans o della drosofila. Tuttavia, gli organismi che sopravvivono hanno difetti nella divisione dei cromosomi e nel ciclo cellulare e, sicuramente, non sarebbero capaci di competere in natura con organismi wild-type. È probabile che in certi organismi la delezione dell'RNAi sia stata favorita dall'esistenza di meccanismi di compensazione alternativi all'RNAi o anche dall'esistenza di selezione negativa del meccanismo di RNAi.
Potenziali applicazioni terapeutiche dell'RNAi
L'osservazione che l'RNAi può portare alla repressione dell'espressione di prodotti genici specifici non poteva passare inosservata come possibile terapia per curare o alleviare certe malattie dell'uomo. Oncogeni, geni codificanti per proteine che sopprimono la crescita dei tumori o recettori di membrana che stimolano la crescita delle cellule, e anche il genoma di alcuni virus come l'HIV-1, sono stati fra i primi geni testati in cellule in coltura e in topi transgenici modello come possibili bersagli dell'RNAi. I primi risultati sono stati incoraggianti. Tuttavia è chiaro che l'applicazione terapeutica dell'RNAi a malattie umane con base genetica necessita ancora di sperimentazione. Come nel caso di ogni nuova tecnica, la metodologia dell'RNAi ha bisogno di essere analizzata criticamente in tutti i suoi aspetti. Tecnicamente parlando, l'introduzione e il mantenimento di molecole di RNA in vivo è difficile, ma non insormontabile. Fondamentale per l'uso dell'RNAi nell'uomo sarà determinare la specificità dei siRNA o dei miRNA terapeutici nel reprimere l'espressione di un gene specifico e non di altri. La sperimentazione in vitro o in topi è stata decisiva e sicuramente continuerà ad avere un ruolo essenziale per definire le regole per la costruzione di molecole di piccoli RNA che possano avere valore terapeutico. Un altro aspetto importante che deve essere preso in considerazione è lo studio della funzione e della regolazione dell'RNAi nell'uomo. Come accennato sopra, ci sono otto membri della famiglia Ago nell'uomo e soltanto la funzione della proteina Ago2 è nota. Ci sono ancora molte domande che devono trovare risposta prima che l'applicazione terapeutica dell'RNAi diventi realtà. Qual è la funzione delle varie proteine Ago umane? La funzione di queste varie proteine è ridondante o specifica? Può l'introduzione di quantità significative di piccoli RNA sintetici alterare o danneggiare altri processi regolati dall'RNAi nell'uomo?
Bibliografia
Elbashir 2001: Elbashir, Sayda M. e altri, Duplexes of 21-nucleotide RNAs mediate RNA interference in cultured mammalian cells, "Nature", 411, 2001, pp. 494-498.
Fire 1998: Fire, Andrew e altri, Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans, "Nature", 391, 1998, pp. 806-811.
Guo, Kemphues 1995: Guo, Su - Kemphues, Kenneth J., Par-1, a gene required for establishing polarity in C. elegans embryos, encodes a putative Ser/Thr kinase that is asymmetrically distributed, "Cell", 81, 1995, pp. 611-620.
Hamilton, Baulcombe 1999: Hamilton, Andrew J. - Baulcombe, David C., A species of small antisense RNA in posttranscriptional gene silencing in plants, "Science", 286, 1999, pp. 950-952.
Hammond 2000: Hammond, Scott M. e altri, An RNA-directed nuclease mediates post-transcriptional gene silencing in Drosophila cells, "Nature" 404, 2000, pp. 293-296.
Lee 1993: Lee, Rosalind C. - Feinbaum, Rhonda L. - Ambros, Victor, The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14, "Cell", 75, 1993, pp. 843-854.
Liu 2004: Liu, Jidong e altri, Argonaute2 is the catalytic engine of mammalian RNAi, "Science", 305, 2004, pp. 1437-1441.
Napoli 1990: Napoli, Claudio - Lemieux, Claude - Jorgensen, Richard A., Introduction of a chimeric chalcone synthase gene into petunia results in reversible co-suppression of homologous genes in trans, "The plant cell", 2, 1990, pp. 279-289.
Van der Krol 1990: van der Krol, Alexander R. e altri, Flavonoid genes in petunia: addition of a limited number of gene copies may lead to a suppression of gene expression, "The plant cell", 2, 291, 1990.