Microbiologia
Microbiologia
Sommario: 1. Introduzione: a) premessa; b) come definire i microrganismi? 2. Sistematica dei Procarioti. 3. I metodi della microbiologia: a) microscopia; b) crescita microbica e metodi di coltura; c) metodi quantitativi di misura della crescita; d) terreni di coltura e fattori che influenzano la crescita; e) colture pure e conservazione. 4. Microbi in condizioni estreme: a) termofili; b) acidofili. 5. Ciclo vitale: a) la cellula e la sua struttura; b) moltiplicazione e sporulazione; c) fattori di resistenza. 6. Genetica molecolare dei microrganismi: a) controllo dei geni nei Procarioti; b) mutazioni; c) reversioni; d) elementi genetici trasponibili; e) mappa fisica del genoma batterico; f) introni ed esoni; g) inteine. 7. Virus e batteriofagi. 8. Archaea. 9. Biotecnologia: a) introduzione; b) microrganismi produttori di sostanze chimiche di interesse industriale; c) lisciviazione batterica e bioestrazione; d) bioeliminazione dell'inquinamento ambientale; e) metodo microbiologico per il controllo degli Insetti; f) individuazione rapida di batteri patogeni. 10. Interazione di piante e microrganismi: a) premessa; b) resistenza delle piante all'attacco di microrganismi. 11. Azione patogena dei Batteri: a) premessa; b) sistematica dei batteri patogeni; c) relazione ospite-batterio; d) fattori di virulenza. □ Bibliografia.
1. Introduzione
a) Premessa
I microrganismi sono numerosissimi, vari, invasivi e invadenti e, al pari delle religioni, influenzano nel bene e nel male l'esistenza degli uomini. Alcuni di essi sono di grande utilità: per esempio, i batteri presenti nella massa escreta giornalmente da Homo sapiens sono utili perché producono nell'intestino alcune delle vitamine essenziali; Saccharomyces cerevisiae è impiegato nella fermentazione del maltosio (lievito di birra) e del glucosio (lievito di panificazione), rispettivamente per produrre la birra e per far lievitare il pane. Altri producono sostanze caratteristiche, come il pigmento elaborato in opportune condizioni da Serratia marcescens, presente anche nelle cucine, tra le fibre di legno delle assi per tagliare, che impartisce alle colture una singolare colorazione rossa, responsabile di alcuni ‛miracoli del sangue' segnalati in passato nelle ostie, nel pane, nella polenta e talvolta immortalati in opere d'arte. Altri ancora, infine, sono stati causa di gravissime epidemie le quali, oltre a provocare elevatissime mortalità, hanno talvolta scatenato la ‛caccia agli untori' (per lo più Ebrei): Yersinia (Pasteurella) pestis fu l'agente delle pestilenze che tra il XIV e il XVII secolo uccisero il 30% della popolazione mondiale (Boccaccio fece riferimento, nel Decameron, a quella che infierì a Firenze nel 1348, e Manzoni, nei Promessi sposi, descrisse quella di Milano del 1630); il virus dell'influenza, tristemente nota come ‛spagnola', causò nel 1918 un numero maggiore di vittime che non il primo conflitto mondiale.
Si deve alle ricerche dei microbiologi il chiarimento dei vari fenomeni la cui origine risiede nel mondo dei microrganismi. La microbiologia ha subito nel tempo fasi alterne di sviluppo: a un periodo iniziale di grande interesse, nei primi anni del secolo, dovuto alla scoperta dei Batteri e alla messa a punto delle tecniche e degli strumenti di analisi (microscopio ottico e mezzi di coltura), ha fatto seguito, dagli anni cinquanta, una fase di stasi coincidente con la scoperta di sostanze battericide e batteriostatiche (penicillina e sulfamidici). Attualmente si è destato un rinnovato interesse per lo studio dei microrganismi per due ordini di motivi: appartengono al primo la comparsa della resistenza agli antibiotici in alcuni batteri (dovuta ai plasmidi R) e la descrizione quasi giornaliera di nuovi organismi o forme epidemiche dagli effetti drammatici (infezioni da streptococco invadente di gruppo A, in grado, per esempio, di provocare la distruzione di tutti i muscoli in pochi giorni; diffusione epidemica, in Cina e a New York nel 1995, di forme tubercolari da Mycobacterium tubercolosis resistente agli antibiotici; gravi epidemie di febbri emorragiche da virus Ebola, che uccidono in poche ore); appartengono invece al secondo ordine di motivi l'impiego dei microrganismi nelle ricerche di genetica e lo sviluppo della biologia molecolare, che ha consentito acquisizioni di notevole importanza sia per l'ampliamento delle conoscenze scientifiche, sia per il futuro sviluppo di nuovi mezzi di difesa.
b) Come definire i microrganismi?
Il termine ‛microrganismi' si deve all'esistenza di un criterio secondo il quale venivano considerati microscopici gli organismi di dimensioni non superiori ai 10 µm (10 millesimi di millimetro); la terminologia corrente include nella ‛microbiologia' lo studio di organismi genericamente unicellulari o microscopici, che per maggiore precisione scientifica debbono essere trattati in altri capitoli della biologia. Fino al secolo scorso gli organismi viventi erano classificati secondo il ruolo svolto nell'economia della natura: si faceva distinzione tra animali, eterotrofi per il carbonio (che assumono col nutrimento le sostanze organiche complesse di cui hanno bisogno), e piante, autotrofe (che utilizzano l'energia solare per sintetizzare sostanze necessarie alla vita da materiale inorganico); i Funghi erano considerati vegetali, mentre organismi unicellulari come Batteri e muffe mucose apparivano di non facile classificazione. Solo successivamente si è scoperto che i microrganismi seguono diverse vie metaboliche: alcuni convertono il materiale organico in composti inorganici e minerali usando l'energia luminosa, altri hanno la capacità di assimilare composti organici o di ossidare composti inorganici.
Considerando la cellula come unità fondamentale della vita è possibile un'altra definizione di microrganismi. Mentre Ernst Haeckel nel 1866 aveva coniato il termine Protisti (forme arcaiche) per indicare un grande gruppo comprendente Alghe, Funghi e Protozoi, una suddivisione più recente ha distinto due gruppi sistematici, i Procarioti e gli Eucarioti, sulla base della struttura del loro nucleo (karyon) cellulare; nelle cellule dei Procarioti - molti dei quali sono in grado di vivere in condizioni di anaerobiosi e di fissare l'azoto atmosferico - il nucleo è costituito da una molecola circolare di DNA immersa in un citoplasma privo di compartimenti e generalmente è presente una parete cellulare contenente mureina; nelle cellule degli Eucarioti, invece, il nucleo è delimitato da una membrana cellulare.
I Virus costituiscono un gruppo a parte, di difficile collocazione sistematica: la loro caratteristica fondamentale è quella di essere parassiti obbligati. Nella presente trattazione saranno presi in considerazione soltanto i batteriofagi, cioè i virus che infettano i Batteri.
2. Sistematica dei Procarioti
I Procarioti includono il gruppo degli schizomiceti (Batteri nel senso più ampio, o Schizofite) e quello degli schizoficeti (Cianobatteri). La loro descrizione e il loro nome debbono essere in accordo con il codice batteriologico stabilito dal Comitato Internazionale di Batteriologia Sistematica; per ogni nuovo ceppo descritto deve essere depositata una coltura vivente, catalogata in un genere riconosciuto che serva come riferimento.
Il sistema della classificazione linneana degli organismi viventi, basata sulla morfologia, è stato applicato finora anche ai microrganismi, benché riconosciuto inadeguato fin dalla metà di questo secolo. Effettivamente, le differenze morfologiche tra i microrganismi non sono sufficienti per la loro tassonomia; d'altra parte le tecniche di studio non potevano fornire alcuno strumento valido per sostenerne scientificamente le affinità e le derivazioni evolutive.
Attualmente la sistematica dei microrganismi sta subendo trasformazioni fondamentali e i progressi conseguiti nella caratterizzazione molecolare delle cellule hanno portato alla conclusione che le cellule dei Procarioti sono altrettanto complesse quanto quelle degli Eucarioti. Gli studi di biochimica molecolare e di genetica hanno fornito ai sistematici una chiave scientifica non solo per la classificazione dei microrganismi, ma anche per risalire alla loro origine filogenetica.
La conoscenza dell'evoluzione di molecole complesse nell'interno delle cellule, ben riconoscibili e poco soggette a variazione, quali le molecole dell'RNA 16S del ribosoma, si è dimostrata la linea più affidabile per risalire alle affinità e alle derivazioni filogenetiche dei microrganismi. Secondo tale metodo i Procarioti sono stati riuniti in gruppi molto affini tra loro dal punto di vista della biochimica molecolare, tenendo conto anche delle conoscenze circa la derivazione evolutiva sia dei Procarioti che degli Eucarioti. Relativamente agli Eucarioti è a questo punto sorto un dubbio: derivano veramente dall'evoluzione dei Procarioti o, piuttosto, ambedue i gruppi derivano da un progenitore comune?
In base ai dati molecolari, Carl Woese ha proposto un nuovo prospetto sistematico, l'albero filogenetico universale degli organismi viventi: si tratta di un albero senza radici, costruito su una matrice di distanza evolutiva derivata dall'allineamento di piccole sequenze della subunità 16S dell'rRNA. Nel calcolo dell'allineamento sono state usate solo le posizioni giudicate omologhe tra i tre dominî (v. Woese, 1987). Questo albero presenta tre diramazioni: Bacteria, Archaea, ed Eucarya. Woese fa notare che lo schema non si dirama da un progenitore comune; inoltre gli studi biochimici e sul metabolismo dimostrerebbero che i Bacteria sono meno affini agli Archaea che agli Eucarya, e pertanto è bene sostituire il termine Archaebacteria delle vecchie classificazioni con quello di Archaea, e quello di Eubacteria con Bacteria.
Questo metodo ha rivelato l'esistenza di relazioni insospettate, per esempio tra Azotobacter ed Escherichia, ed è in discussione se batteri come Methanococcus jannaschii, Methanobacterium autotrophicum e Sulfolobus solfataricus siano più vicini agli organismi superiori che a Eubacteria come Escherichia coli ed Haemophilus influenzae, o se siano invece tutti un ramo evolutivo unico: a tali domande si potrà rispondere quando sarà completato lo studio del genoma di questi batteri. Attualmente sono note le sequenze dell'rRNA 16S di almeno 10.000 microrganismi e il numero totale di queste sequenze per tutti i tipi di organismi e organelli (per es., mitocondri, cloroplasti) è di circa 20.000. La classificazione pertanto può essere fatta basandosi sulle somiglianze del loro RNA ribosomiale.
Oltre a queste classificazioni scientifiche sono in uso elenchi di microrganismi suddivisi secondo le loro funzioni ambientali (batteri patogeni, di utilità industriale e agricola, ecc.); inoltre, l'edizione più recente del Bergey's manual of determinative bacteriology divide i Procarioti in 35 gruppi distinti in base a tre caratteristiche: la reazione alla colorazione di Gram, la morfologia cellulare e il rapporto con l'ossigeno (v. Holt e altri, 19949).
3. I metodi della microbiologia
a) Microscopia
L'occhio umano non può distinguere particelle più piccole di 0,1 mm, mentre i microrganismi sono almeno 10 volte più piccoli, tranne poche eccezioni (per esempio, Epulopiscium fishelsoni di circa mezzo millimetro, recentemente trovato in un pesce del Mar Rosso).
Le ipotesi più elaborate e ardite formulate dagli scienziati richiedono conferma da tecniche sempre più sofisticate che permettano di collegare concettualmente le funzioni cellulari con le strutture morfologiche e le macromolecole che le costituiscono. Ad esempio, recentemente (v. Holzmann, 1996) è stato possibile confermare, grazie all'analisi con la reazione di polimerizzazione a catena (PCR, Polymerase Chain Reaction), che organismi simili a batteri (BLO, Bacterium-Like Organisms), osservati sin dal 1972 al microscopio elettronico nelle spore del fungo Gigantospora margarita, sono effettivamente batteri. Appartengono al gruppo II di Pseudomonadi del genere Burkpolderia e ogni spora ne contiene centinaia: fanno parte integrante del sistema endosimbiotico di scambio metabolico col fungo ospite, il quale a sua volta è un simbionte delle piante. Questi risultati mettono in evidenza l'importanza della simbiosi, un sistema più largamente distribuito di quanto non si pensasse.
Il microscopio ottico permette ingrandimenti fino a 1.000 volte; il microscopio elettronico, che consente di condurre l'indagine all'interno delle cellule, e quello a scansione, che permette di verificare in dettaglio le strutture delle superfici, rendono possibili ingrandimenti fino a 100.000 volte. A tali ingrandimenti si possono vedere elementi ultrastrutturali: ribosomi, plasmidi, strati di pareti cellulari, addirittura strutture molecolari. Il massimo potere di risoluzione del microscopio ottico composto consente di distinguere tra loro due punti separati da una distanza di soli 200 nm. Le prestazioni di questo microscopio possono essere migliorate con un sistema che permette di accentuare il contrasto nel campione in esame senza bisogno di coloranti, che danneggiano le cellule (microscopia a contrasto di fase). Il microscopio a fluorescenza sfrutta lunghezze d'onda della luce nella fascia del blu o dell'UV, capaci di eccitare la fluorescenza naturale del campione o quella indotta mediante colori fluorescenti. Il microscopio elettronico a trasmissione utilizza un fascio di elettroni sotto vuoto al posto della sorgente di luce e una serie di elettromagneti in luogo delle lenti: si debbono usare preparati molto sottili (50 nm), ottenuti con la tecnica del criodecapaggio e ombreggiati con metalli.
b) Crescita microbica e metodi di coltura
Per studiare i microrganismi occorre poterli isolare, arricchire e coltivare: ‟Lo studio della crescita di colture batteriche è il metodo base della microbiologia" (v. Monod, 1949, p. 372). Il tasso di crescita indica l'attività delle reazioni di sintesi dei Batteri nell'ambiente di coltura in cui sono immersi e costituisce un dato quantitativo che ha permesso di iniziare gli studi di fisiologia batterica. Per quanto riguarda i metodi di coltura, negli ultimi anni non si sono registrate novità sostanziali. Tuttavia, le tecniche per le colture anaerobiche, recentemente migliorate, hanno mostrato che il rapporto tra aerobi facoltativi (ad esempio E. coli) e anaerobi obbligati era largamente sottovalutato: si trovano infatti meno di 107 E. coli per grammo di massa batterica intestinale e 1010 - 1011 anaerobi obbligati. È interesse dei biologi e di chi utilizza colture di Batteri a scopo industriale mantenere una coltura in crescita continua indefinita: questa possibilità, realizzabile in condizioni che restino immutate, come nelle colture continue (chemostat o bactogène), è di fatto limitata da mutazioni e contaminazioni che danneggiano la resa industriale. A questi inconvenienti si è ovviato, almeno in parte, con la selezione di batteri più produttivi, che danno una resa maggiore in tempi più brevi.
c) Metodi quantitativi di misura della crescita
I metodi che permettono di misurare la crescita batterica sono rimasti pressoché immutati. Due sono gli elementi sui quali si basa l'analisi quantitativa: monitorare l'aumento del numero dei Batteri e misurare i cambiamenti della biomassa. Col primo metodo si misura nel tempo la crescita numerica dei Batteri, col secondo si misura la massa, ponderale o biochimica, delle colture. Il conteggio dei Batteri può essere effettuato o mediante la conta diretta delle cellule batteriche, al microscopio su preparati colorati, o indirettamente misurando la torbidità della coltura, oppure mediante la conta delle cellule vitali. Per cellule vitali si intendono non le cellule capaci di metabolizzare, ma quelle capaci di riprodursi e formare una colonia su terreno solido di coltura (CFU, Colony Forming Unit): su terreno solido in piastre di Petri si possono contare fino a 500 colonie isolate. Il metodo più usato è quello della misura della torbidità di colture liquide, mediante un colorimetro o uno spettrofotometro che ne danno la densità ottica standardizzata, misurabile fino a valori di 108/ml. Più complessa è la misura della biomassa, che può essere determinata con la misura del peso umido o del peso secco: data la difficoltà di sottrarre dal peso delle cellule quello del diluente negli spazi interstiziali, è usato più comunemente il peso secco, che si ottiene essiccando un volume determinato in forno a 105 °C o in essiccatore a vuoto o per liofilizzazione.
d) Terreni di coltura e fattori che influenzano la crescita
La possibilità di sopravvivenza e di diffusione dell'enorme varietà dei microrganismi è legata alla loro capacità di vivere in ambienti molto diversi: dal polo all'equatore, dal petrolio alle solfatare, dall'interno di organismi viventi alle profondità degli oceani. Il metodo delle colture in laboratorio ha permesso di condurre indagini sulle condizioni necessarie per la vita di molti microrganismi.
In relazione alle necessità ambientali di vita, i Batteri possono essere così raggruppati: prototrofi, auxotrofi, sintrofici, autotrofi ed eterotrofi. I prototrofi crescono in un terreno di coltura con pochi sali minerali e utilizzano una sola sorgente di energia e carbonio, mentre gli auxotrofi (per esempio, i batteri lattici), incapaci di sintetizzare i componenti metabolici essenziali, richiedono la presenza nel terreno di fattori di crescita (vitamine, amminoacidi, ecc.). Alcuni gruppi batterici (batteri sintrofici) non possono vivere autonomamente, ma solo in presenza di cellule animali, vegetali o batteriche, capaci di fornire nutrimenti non identificati: per esempio, i batteri anaerobi obbligati dipendono dalla presenza di batteri riduttori; Syntrobacter, Syntrophomonas, ecc. vivono in presenza di metanogeni anaerobi che utilizzano idrogeno. I batteri autotrofi (per esempio, Azotobacter, Nitrobacter, ecc.) ricavano energia dall'utilizzazione della luce (cianobatteri e batteri fotosintetici) o dalla trasformazione chimica di sostanze inorganiche quali nitrati, nitriti e azoto atmosferico. I batteri eterotrofi, al contrario dei precedenti, richiedono fonti organiche di energia.
Tutti i microrganismi necessitano per vivere di azoto, zolfo e carbonio, utilizzati da alcuni in forma organica, da altri in forma inorganica. Sono loro indispensabili anche microelementi, richiesti alcuni in piccola quantità (10-9-10-8 molare) - come cofattori di enzimi (Zn, Mg, Mn, Fe, K, ecc.), nel trasporto di membrana (Na, Ca) e nelle molecole a struttura complessa (Ca nelle spore) - altri soltanto in traccia (Cu, Co, Mo, ecc.); ai terreni di coltura vanno aggiunte anche le vitamine, che non tutti i batteri sintetizzano.
Per soddisfare la necessità di mantenere i terreni di coltura ad acidità costante e specifica per i diversi batteri, si impiegano soluzioni tampone naturali o sintetiche, non metabolizzabili: il pH del terreno può variare da estremamente acido a molto basico.
e) Colture pure e conservazione
Qualsiasi studio si intenda intraprendere sui Batteri, prima di tutto è necessario ottenere delle colture pure. A tal fine, sfruttando le proprietà specifiche di ogni ceppo, nella sospensione batterica si cerca di favorirne uno in particolare, mediante opportune variazioni delle condizioni ambientali (temperatura, pressione, composizione del terreno, radiazioni, centrifugazione con gradiente di densità, ecc.) e procedendo poi alla sua separazione spaziale su terreno solido: si usano per questo piastre di Petri con agar (estratto da alghe marine rosse), non degradabile enzimaticamente e stabile a 65 °C, o con gel di silice, altro materiale solidificante, completamente privo di sostanze organiche; per l'isolamento di organismi termostabili si usa ‛gellongum', un polimero eterosaccaride ricavato da Pseudomonas, stabile a 125 °C e 265 atm.
Una volta ottenuta la coltura pura, esistono vari metodi per conservarla, come il rapido congelamento con azoto liquido, previa aggiunta di glicerolo - probabilmente il metodo migliore -, o la liofilizzazione (freeze-drying), molto usata anche nell'industria, che permette la conservazione in piccolo volume. Nonostante l'aggiunta di sostanze protettive (crioprotettori), durante il processo si possono verificare mutazioni e la sopravvivenza, specialmente quella dei mutanti con plasmidi o costruiti con ingegneria genetica, è limitata: per esempio, la sopravvivenza di E. coli riidratato (in presenza di glicole polietilenico e di saccarosio come protettori) è inferiore allo 0,01%.
4. Microbi in condizioni estreme
L'esistenza di tanti tipi differenti di microrganismi - termofili, acidofili, alcalofili, oligotrofi, osmofili, alotolleranti, alofili, metallotolleranti - rappresenta una conferma della loro capacità di adattamento e di colonizzazione di ambienti diversi. Nel caso dei termofili e degli acidofili, la temperatura è un buon parametro per illustrarne la diversità d'adattamento.
a) Termofili
I microrganismi termofili sopravvivono e crescono a temperature più alte di quelle considerate normali per i sistemi biologici. Nella grande maggioranza gli organismi termofili sono batteri in grado di crescere tra 42 e 100 °C o più (i mesofili, come E. coli, vivono tra 10 e 42 °C). Salinità, pH e pressione sono fattori importanti per i termofili: le sorgenti calde (geysers) solubilizzano cloruri, solfati, bicarbonati, ecc., e quelle che hanno origine dalle profondità marine sono soggette anche a pressioni elevate, così che i microrganismi in grado di vivervi debbono essere resistenti a più fattori. Con l'aumento della temperatura aumentano anche le specie anaerobiche, probabilmente perché diminuisce la solubilità dell'ossigeno. I termofili obbligati crescono tra 40 e 80 °C, ma, se formano spore, sopravvivono a temperature più alte; a temperature superiori agli 80 °C predominano gli Archaea. Lo studio dei termofili presenta difficoltà tecniche, quali ad esempio la necessità di mantenere l'acqua in forma liquida a temperature superiori ai 100 °C e la disponibilità di terreni di coltura che, a temperature così elevate, conservino inalterate le loro caratteristiche.
Come possono i termofili sopravvivere alla temperatura di ebollizione dell'acqua e anche più alta? La risposta a questa domanda non è semplice. Si è constatato che molte proteine dei termofili sono termostabili e sono stati identificati fattori che ne stabilizzano le molecole nei confronti della temperatura. I fosfolipidi a doppio strato della membrana citoplasmatica sono importanti nel regolarne l'attività e la struttura, essenziali per mantenere il gradiente ionico e controllare l'assorbimento dei fattori nutritivi; gli acidi grassi controllano la fluidità della membrana.
I microrganismi termofili più studiati sono: Bacillus coagulans (termofilo facoltativo 30-60 °C), Bacillus stearothermophilus (termofilo obbligato 40-80 °C) e Thermus aquaticus (45-79 °C), dal quale viene estratta la polimerasi TAQ1 usata nell'amplificazione dei geni.
b) Acidofili
I gas vulcanici sono spesso ricchi in solfuro d'idrogeno (idrogeno solforato) che, arrivato in superficie, è ossidato dall'ossigeno dell'aria a zolfo e poi ad acido solforico: classici ambienti di questo tipo sono la solfatara di Pozzuoli (Forum Vulcani), un piccolo cratere vulcanico fumante nei Campi Flegrei, e altri in varie parti del mondo, per esempio nel parco di Yellowstone nel Wyoming. Sembra impossibile concepire l'esistenza di forme di vita in ambienti estremi come le aree geotermiche, nelle quali l'acido solforico prodotto distrugge addirittura le rocce tranne il quarzo; eppure in questi terreni a basso pH vivono e prosperano alcuni organismi, come i generi Thermoplasma, Sulfolobus, Cyanidium. È interessante notare il rapporto esistente tra temperatura e pH, tant'è che le sorgenti di acqua bollente a pH neutro contengono batteri, ma non quelle a basso pH e alta temperatura: Sulfolobus acidocalcarius, che nel parco di Yellowstone cresce abbondante quando la temperatura diminuisce a 90 °C (da tener presente che all'altitudine del parco il punto di ebollizione dell'acqua è a 92,5 °C), non può crescere a temperature inferiori a 55 °C, alle quali invece cresce Thiobacillus. Sorgenti industriali di calore, come i bollitori delle lavanderie, le raffinerie dello zucchero, le stazioni generatrici delle industrie atomiche, le macine della carta e dei tessuti, sono ricche in Thermus aquaticus (v. Brock, 1995).
5. Ciclo vitale
Osservando i processi vitali di un organismo pur apparentemente semplice come un batterio, si resta sorpresi della straordinaria organizzazione delle sue funzioni, talvolta molto complesse, come una crescita regolare e coordinata. Gli elementi chiave di questa serie ordinata di eventi (ciclo cellulare), ognuno dei quali avviene in un momento preciso della vita della cellula, sono individuabili nella replicazione del DNA e nella segregazione dei nucleoidi che iniziano la divisione cellulare. A tutt'oggi, tuttavia, non ci è noto quale sia il segnale che dà inizio a questi eventi del ciclo cellulare, ma alcuni biologi (tra i quali J. Shapiro) ipotizzano che possa dipendere dalla massa cellulare, dalla densità di sopravvolgimento del cromosoma e dalla concentrazione di due nucleotidi regolatori, i cosiddetti allarmoni: AMP ciclico e ppGpp (pirofosforilguanosindifosfato). Altri allarmoni sono stati indicati quali iniziatori della transizione da anaerobiosi ad aerobiosi, per segnalare lo shock termico, ecc.
In una colonia batterica i singoli individui comunicano tra di loro con interazioni e coordinazioni multicellulari: questo è il meccanismo che permette alle colonie di alcuni batteri di assumere forme caratteristiche (autoaggregazione chemiotattica) usando segnalazioni chimiche a distanza e mobilità di gruppo.
a) La cellula e la sua struttura
La forma dei Batteri (sferica, a bastoncino, a spirale, ecc.) è dovuta all'involucro rigido, costituito da vari strati, che li racchiude; le cellule dei Procarioti non hanno citoscheletro né mitocondri; tuttavia, la loro struttura a livello molecolare e biochimico è estremamente complessa e paragonabile a quella degli organismi superiori. Un esempio di tale complessità è riscontrabile nella parete cellulare che, oltre a dare la forma specifica alla cellula, la isola dall'ambiente esterno, col quale tuttavia deve permettere scambi continui: è chiaro che, per consentire lo svolgimento di queste due funzioni antagoniste, la sua struttura debba essere molto raffinata. Gli studi più approfonditi sono stati condotti sulla membrana dei batteri gram-negativi come E. coli. Recentemente si è cominciato ad analizzare anche la membrana dei gram-positivi, nella quale è stata individuata la presenza di proteine con lipidi legati all'azoto terminale di una molecola di cisteina, analogamente a quanto osservato nei gram-negativi; nei gram-positivi lo strato esterno costituito dai peptidoglicani (mureina), per quanto abbia spessore molto maggiore che nei gram-negativi, è tuttavia molto poroso; mentre tutti i gram-negativi producono peptidoglicani di struttura identica, i gram-positivi ne elaborano molti di tipo differente per resistere agli enzimi lisanti dell'ambiente. La parete cellulare dei Batteri ha una triplice funzione fisiologica: è il limite esterno del periplasma (regione compresa tra membrana esterna e membrana cellulare); presenta numerose cariche negative, fondamentali per evitare la fagocitosi e le reazioni immunologiche; è una barriera di permeabilità contro agenti tossici dell'ospite (lisozimi, proteine leucocitarie, ecc.) e protegge il batterio dall'azione degli antibiotici.
La struttura della parete degli Archaea è diversa da quella dei Batteri. Gli Archaea, infatti, non solo contengono un particolare tipo di peptidoglicani, chiamato endomureina, ma, a differenza dei Batteri, la loro membrana contiene esteri del glicerolo e manca dell'acido muramico; questo è uno dei fattori che differenziano gli Archaea da tutti gli altri sistemi biologici.
La presenza della parete cellulare non deve impedire il passaggio di sostanze nutritizie e di prodotti del catabolismo, il cui flusso è garantito da speciali strutture che formano dei canali attraverso lo spessore della membrana. Questi canali sono: porine; canali specifici con siti di legame per le sostanze trasportate; recettori ad alta affinità che trasportano grandi molecole contro il gradiente osmotico, utilizzando energia (vitamina B12, complessi chelanti Fe3+). Attraverso le porine ha luogo il trasporto spontaneo di soluti di piccole dimensioni; per esempio la porina per il trasporto del maltosio e delle maltodestrine si lega alle molecole di questi zuccheri mediante un sito specifico di legame ed è ripiegata 18 volte nell'interno della membrana.
Le cellule dei Procarioti mancano di membrane interne e di organelli intracellulari, ad eccezione dei tilacoidi (apparato fotosintetico dei Cianobatteri).
b) Moltiplicazione e sporulazione
In condizioni ambientali favorevoli, i Batteri si moltiplicano vegetativamente per scissione; se le condizioni diventano sfavorevoli, sia in natura che in coltura, inizia la formazione di spore, che rappresentano per i microrganismi una fase di quiescenza (criptobiosi) e un modo di sopravvivenza. La formazione di endospore è tipica dei bacilli gram-positivi del terreno; esse si formano dentro la cellula madre ma hanno proprietà strutturali, chimiche e fisiologiche diverse. I biologi sono tuttora alla ricerca di quali siano i segnali molecolari che provocano una serie così complessa di modifiche nella biochimica della cellula. Lo stato di quiescenza delle spore può durare anche milioni di anni: sono state isolate, dall'intestino di un'ape domenicana inclusa nell'ambra, spore di Bacillus sphaericus datate 25-40 milioni di anni fa. Spore trovate in antichi depositi fossili di migliaia e forse milioni di anni fa, messe in condizioni favorevoli, hanno potuto germinare (risultato però ancora discusso). Le spore possono tollerare variazioni enormi delle condizioni ambientali, per esempio possono resistere a un'ora di ebollizione; a che cosa è dovuta una capacità così notevole di sopravvivenza?
La descrizione della formazione delle spore fornisce una risposta a questo interrogativo. La cellula che percepisce il segnale per la sporulazione interrompe la normale divisione cellulare e, anziché separare i due nucleoidi con una membrana, li fonde in un filamento assiale di cromatina. Una metà di questo filamento si concentra a un polo della cellula dove, per invaginazione della membrana della cellula madre, si forma un setto traverso che porta all'individuazione di una prespora. La cellula madre ingloba la prespora e inizia a costruirvi attorno la corteccia, composta in prevalenza da un caratteristico peptidoglicano, uguale in tutti i gruppi di Batteri e costituito da tre subunità: alanina, tetrapeptide e lattame muramico. All'esterno della corteccia si forma la tunica sporale, costituita da proteine ad alto contenuto di cisteina e amminoacidi idrofobi, resistentissimi alla solubilizzazione: la comparsa della resistenza al calore è dovuta a un massiccio assorbimento di Ca2+ correlato alla sintesi di grandi quantità (5-10% del peso secco) di acido dipicolinico, specifico della spora.
Poiché il DNA nell'interno (core) della spora rimane vivente per periodi indeterminati (da decenni a milioni di anni), ci si domanda come non sia modificato da mutazioni (talvolta letali). È chiaro che allo stato dormiente tutti gli enzimi sono inattivi, ma la sopravvivenza del DNA rimane ancora inspiegata. All'inizio della sporulazione il DNA è condensato e saturato da un gruppo di piccole proteine acido-solubili (SASP, Small Acid-Soluble Proteins) recentemente descritte e considerate responsabili della sua resistenza (v. Setlow, 1992).
È interessante notare che durante la formazione della spora si svolge la sintesi di peptidi a basso peso molecolare con attività antibiotica, comprendenti le edeine, che inibiscono la sintesi del DNA, le bacitracine, che inibiscono la sintesi della parete cellulare, e altri peptidi che modificano strutture o funzioni della membrana, quali polimixina, tirocidina e gramicidina: alcuni di questi antibiotici sono usati anche in farmacologia.
Esistono, oltre agli aerobi, anche sporogeni anaerobi: questi ultimi, altamente patogeni a causa delle tossine che producono, si trovano nel terreno, come il genere Clostridium, comprendente gli agenti eziologici del tetano, del botulismo e della gangrena gassosa.
Una spora rimane allo stato quiescente, anche se si trova in condizioni favorevoli allo sviluppo, finché non interviene un processo di attivazione (calore, frantumazione, ecc.). La germinazione è preceduta dall'eliminazione di dipicolinato di calcio dal citoplasma della spora e di frammenti di peptidoglicani della corteccia, così che rimane la sola parete esterna. Perché abbia inizio la germinazione occorre uno stimolo chimico (alanina, ribosidi, glucosio e altri) in seguito al quale si attivano gli enzimi presenti allo stato inattivo nel citoplasma della spora: la cellula si accresce allora sino a raggiungere le dimensioni idonee per la divisione cellulare e riprende il ciclo vegetativo.
c) Fattori di resistenza
La scoperta di sulfamidici e di antibiotici, sostanze battericide che agiscono formando canali ionici nella membrana o aggredendo gli acidi nucleici della cellula batterica, aveva fatto sperare che i batteri patogeni potessero essere completamente debellati. In realtà, dato che i Batteri sono sopravvissuti per quattro miliardi di anni era illusorio sperare di poterli eliminare in breve tempo. Infatti nel 1956 si constatò che l'efficacia di penicillina, streptomicina, tetraciclina e cloramfenicolo era temporanea e si andavano sviluppando ceppi resistenti anche a tutti e quattro gli antibiotici. Questi dati, apparsi inizialmente paradossali ai biologi, poterono essere confermati da semplici esperimenti: bastava mescolare tra loro colture di ceppi batterici resistenti e sensibili per ottenere batteri che, coltivati su terreno solido selezionante, erano tutti resistenti. La resistenza era acquisita per trasferimento di geni mediante contatto cellulare e coniugazione batterica: il fattore R (un plasmide), infatti, porta geni di resistenza ai vari antibiotici. Si cercò di arginare l'uso indiscriminato di antibiotici: è da notare che nel 1980 solo il 50% degli antibiotici prodotti era per uso umano e ne fu proibito l'uso in massa in agricoltura o per l'allevamento del bestiame. La scoperta del fattore R ebbe grande importanza al fine di individuare la possibilità di scambio di geni in natura e diede nuovo impulso agli studi sul genoma batterico, alle tecnologie del DNA ricombinante e alle sue applicazioni. L'effetto negativo della resistenza ai farmaci ha tuttavia permesso lo sviluppo di studi e di tecnologie che possono, e ancor più potranno in futuro, risolvere patologie umane e alterazioni genetiche finora senza speranza.
E. coli è il batterio più usato in ingegneria genetica, perché capace di incorporare DNA (plasmidi) e costituire un trasposone (v. cap. 6, § d) che dissemina non solo geni per la resistenza agli antibiotici, ma anche geni responsabili della produzione di sostanze utili come l'insulina. La capacità di resistenza è dovuta non solo a mutazioni genetiche, ma anche a sostanze naturali prodotte da batteri sia gram-positivi che gram-negativi. L'elenco di queste sostanze si allunga continuamente e comprende agenti vari: le batteriocine sintetizzate nei ribosomi (per esempio colicine, prodotte da enterobatteri) rappresentano un pot-pourri di molecole proteiche che interferiscono con la crescita; sostanze colicino-simili sono prodotte anche da gram-positivi, come i batteri acidolattici. Inoltre la maggioranza dei batteri, durante la crescita, produce una serie di sostanze inibitrici verso loro stessi o verso altri batteri o cellule eucariote: ad esempio tossine, prodotte da germi patogeni, che agiscono contro gli Eucarioti provocando malattie quali tetano, colera e difterite; tossine antibatteriche; enzimi batteriolitici; prodotti del metabolismo (H2O2, NH3, acidi organici, ecc.); antibiotici (gramicidina, valinomicina, ecc.).
6. Genetica molecolare dei microrganismi
Il nostro secolo sarà sicuramente ricordato per la realizzazione di enormi progressi scientifici: nel giro di poco più di cento anni si è passati dall'illuminazione a petrolio alla fissione dell'atomo, dalle navi a carbone agli aerei ultrasonici, dal trasporto a cavallo allo sbarco sulla Luna. La biologia non è stata da meno: dalle intuizioni di Mendel si è arrivati all'ingegneria genetica di elementi trasponibili.
Nel 1953 James Watson e Francis Crick misero a punto la struttura del DNA e nel 1960 fu decifrato il codice genetico a esso legato. Questi studi fondamentali aprirono le porte a tecniche rivoluzionarie come quelle del DNA ricombinante (1970), dell'ingegneria molecolare e dell'amplificazione dei geni mediante reazione polimerasica a catena (1994). Grazie a queste tecniche hanno trovato soluzione problemi microbiologici precedentemente considerati insolubili e geneticamente di difficile approccio. Si è visto come l'analisi genetica di Archaea abbia contribuito alla conoscenza dell'evoluzione dei microrganismi (il più vasto gruppo di organismi viventi) e allo studio della diversità della vita.
L'ingegneria genetica è stata utilizzata nell'industria farmaceutica, nella medicina, nell'agricoltura e nell'allevamento del bestiame (per es., produzione di vegetali e animali di peso superiore alla norma, ecc.), e la lista delle sue applicazioni si allunga ogni giorno (v. biotecnologie, vol. X; v. botanica, vol. X; v. genetica, vol. X).
a) Controllo dei geni nei Procarioti
I microrganismi, per poter sopravvivere in ambienti soggetti a rapidi mutamenti, debbono essere dotati di sistemi di regolazione capaci di adeguarsi altrettanto rapidamente alle mutate condizioni di vita.
Se si considerano le complesse e simultanee reazioni biochimiche che stanno alla base della conservazione dell'omeostasi (costanza dell'ambiente interno) della cellula vivente, si può intuire facilmente che debba esistere all'interno della cellula stessa un sistema di regolazione. Ora sappiamo che questo sistema di regolazione è imperniato sul meccanismo di lettura del codice del DNA mediante la trascrizione a RNA messaggero (mRNA) e la traduzione in proteine tramite l'RNA transfer (tRNA) e l'RNA ribosomiale (rRNA). Nei Batteri la mancanza della membrana nucleare fa sì che i processi di trascrizione e di traduzione per la sintesi delle proteine siano accoppiati, dato che l'mRNA non deve essere modificato chimicamente, come negli Eucarioti, prima di essere tradotto. Ugualmente importanti nella regolazione sono sia l'inizio che la terminazione della trascrizione, così da evitare produzione di mRNA non necessario. Nei Batteri l'mRNA ha vita molto breve, cioè ha un rapido turnover, e di conseguenza anche la sintesi di ogni proteina specifica si arresta. Sequenze specifiche di DNA (terminator stop codons) segnalano la fine della trascrizione e la dissociazione dell'RNA-polimerasi dallo stampo. La trascrizione è infatti operata dall'enzima RNA-polimerasi, costituito da cinque subunità, una delle quali, il fattore σ, si lega a una regione del DNA chiamata ‛promotore' (P): questo legame provoca l'apertura e lo srotolamento della doppia elica del DNA, che segna l'inizio della trascrizione, quindi la catena peptidica si allunga finché il fattore σ si libera. Esistono vari tipi di fattori σ, destinati a riconoscere gruppi di promotori diversi: per esempio, σ32 riconosce i promotori per le proteine associate allo shock da calore, σ43 quelli per le proteine vegetative di Bacillus subtilis sporogeno; σ54 opera in condizioni in cui l'azoto costituisce un fattore limitante per E. coli, ecc.
Un promotore spesso serve una serie di geni legati tra loro, che vengono così trascritti da un unico messaggero policistronico: questa sequenza di geni è chiamata ‛operone'. I geni di un operone sono tradotti indipendentemente l'uno dall'altro, cioè ogni segmento ha il suo sito di legame coi ribosomi. Per la regolazione dell'operone lac per il metabolismo del lattosio, la trascrizione inizia al sito P (promotore) e può continuare se il sito O (operatore) dell'operone ne dà il permesso; il sito operatore, infatti, blocca la trascrizione quando è legato alla molecola ‛repressore'; tuttavia la sua liberazione, resa possibile da un segnale esterno - come l'aggiunta nel terreno di un induttore (per es., lattosio o un analogo) - capace di modificare il repressore, avvia la trascrizione che procede poi lungo l'operone. I geni di una cellula non sono sempre espressi tutti, perché l'espressione è regolata da fattori specifici, per esempio dal nutrimento presente nel terreno. Numerose proteine, essenziali per la struttura e per il metabolismo della cellula, sono codificate da geni strutturali trascritti costitutivamente a velocità più o meno costante, mentre la trascrizione di un gruppo minore di proteine che serve alla cellula per adeguarsi ai cambiamenti ambientali è regolata positivamente (induzione) o negativamente (repressione). Questo meccanismo, unico per la sua originalità, fu descritto da Jacques Monod e dai suoi collaboratori dell'Institut Pasteur (1958-1975) i quali lo avevano individuato per la prima volta nella regolazione della sintesi della β-galattossidasi di E. coli indotta dallo zucchero lattosio (e da altri β-galattosidi analoghi) e poi usato come modello di regolazione negativa; a questo primo modello si sono ormai aggiunti migliaia di sistemi diversi di E. coli e di altri organismi.
L'effetto d'induzione e repressione è immediato, come è stato dimostrato misurando direttamente la cinetica di comparsa e scomparsa dell'mRNA specifico. Non tutti i regolatori sono associati in operoni: per esempio, i geni responsabili della sintesi dell'arginina in E. coli sono sparsi nel cromosoma (regulone), come avviene negli Eucarioti, ma sono tutti repressi se è presente arginina. Questo si spiega col fatto che gli operatori hanno tutti una sequenza simile (consensus sequence).
b) Mutazioni
Le mutazioni costituiscono una risorsa insostituibile per lo studio di strutture e funzioni della cellula. Le ricerche genetiche generalmente iniziano con l'analisi di mutazioni relativamente a una funzione specifica, cioè con la selezione di mutanti: si definisce mutante un organismo il cui genotipo, e più precisamente la sequenza di basi del suo DNA, è diverso da quello del tipo selvaggio. Consideriamo tre fenomeni che avvengono nei Batteri, ben noti, ma tuttora oggetto di studio: chemiotassi, sporulazione e sopravvivenza della cellula.
Chemiotassi. - Le cellule di Escherichia coli e di Salmonella typhimurium rispondono alla presenza di sostanze chimiche nel terreno con movimenti di attrazione e repulsione (chemiotassi). Questa capacità di riconoscere le sostanze e di muoversi nel mezzo è il prodotto di ben 50 geni: 10 per riconoscere il segnale, tutti gli altri per costruire e rendere operativi i flagelli. È chiara la funzione di ogni componente del meccanismo che riceve il segnale chimico: esso richiede comunque la fosforilazione di un certo numero di proteine citoplasmiche, ma rimangono oscuri l'interazione e i cambiamenti di struttura. Tutti questi geni sono stati scoperti grazie a mutazioni della funzione chemiotattica e ulteriori mutanti dell'apparato di mobilità hanno reso possibile estendere le conoscenze sulla struttura dei flagelli e delle proteine che partecipano alla torsione (v. Blair, 1995).
Sporulazione. - Mutanti che bloccano lo sviluppo della spora servono allo studio del meccanismo di sporulazione. Uno dei problemi ancora soltanto parzialmente risolti è come il DNA nell'interno della spora resista a danni che potrebbero provocare mutazioni letali o irreparabili, cioè come il DNA rimanga intatto nei secoli. La scoperta delle nuove piccole proteine acido-solubili α/β SASP e il comportamento di mutanti α-β- hanno dimostrato che tali proteine svolgono un ruolo chiave nel proteggere il DNA a cui si legano (v. Setlow, 1995).
Sopravvivenza della cellula. - La diminuzione del tasso di sopravvivenza di batteri in fase stazionaria di crescita segnala l'esistenza di geni essenziali alla sopravvivenza (geni sur, da survival; v. Torno e altri, 1990).
Questi esempi dimostrano che attraverso l'isolamento di mutanti si arriva alla scoperta di nuovi geni con conseguente progresso dell'analisi biochimica.
Le mutazioni sono rare, con una probabilità di 10-10 ÷ 10- per ogni gene specifico, ma la probabilità può salire sino a 10-3 con l'uso di agenti mutageni, quali radiazioni (UV, γ), agenti alchilanti, analoghi di una base (2-amminopurina) e derivati dell'acridina; da notare che tutti i mutageni sono cancerogeni. È chiaro che per isolare i mutanti occorre un metodo di selezione, il più diretto dei quali consiste nell'eliminare gli individui non mutati (selvaggi): per esempio, su un terreno contenente antibiotici, sopravvivono solo i mutanti resistenti. Se la selezione non è possibile, si possono distinguere i mutanti usando coloranti non tossici, o substrati cromofori di un enzima, e separarli fenotipicamente.
Le mutazioni (transizioni, trasversioni, mutazioni di senso, mutazioni nonsenso, inserzioni, delezioni) alterano la sequenza di una o più basi di un gene, modificano uno o più codoni e quindi la proteina prodotta. Inoltre, si possono produrre mutanti mediante riarrangiamenti della sequenza del DNA, usando segmenti che hanno la capacità d'inserirsi in nuovi siti del genoma (in situ mutagenesis) con trasposoni (v. sotto, § d). Queste mutazioni non sono reversibili.
c) Reversioni
La reversione delle mutazioni, con restauro del fenotipo selvaggio, avviene sia per rimpiazzo dell'amminoacido originario (o di un altro con proprietà analoghe) nella struttura della proteina, sia con una seconda mutazione in un sito differente dello stesso gene che compensa l'errore provocato dalla prima: si tratta in questi casi di soppressori intragenici. Il fenotipo può essere, almeno parzialmente, ripristinato anche con mutazioni extrageniche (soppressione informazionale): per esempio, una mutazione dell'RNA di trasporto acilato (tRNA-AA) può essere ottenuta utilizzando un anticodone mutato in grado di riconoscere il codone ‛stop', e completare la proteina mediante aggiunta di un amminoacido nel sito mutato.
In passato, per determinare i geni e i loro prodotti che contribuiscono a un fenotipo specifico venivano isolati centinaia di mutanti esaminandone, mediante ricombinazione genetica, il fenotipo specifico (complementazione); fu creata così la mappa dei geni mutati in un sistema (v. Yanofsky e altri, 1966).
Sono già stati descritti i tre metodi correnti di trasferimento dell'informazione genetica dei Batteri (trasformazione, trasduzione e coniugazione). Una volta poste le basi delle funzioni biochimiche e chiariti i principî di struttura, funzione e regolazione del gene, le mutazioni e questi metodi di trasferimento furono considerati all'origine delle diversità genetiche e dell'evoluzione di nuove specie batteriche. Tuttavia, la bassa probabilità di mutazioni difficilmente può spiegare la velocità dell'evoluzione. Spiegazioni più plausibili possono forse scaturire dalle nuove conoscenze sulla genetica molecolare e sulla mobilità dei geni, cioè la possibilità di trasferire molecole ricombinanti nei gram-negativi, quali E. coli, S. typhimurium, Acinetobacter calcoaceticus, Haemophilus influenzae, Neisseria, Pseudomonas, ecc.
Nei batteri gram-positivi, in cui non esiste un meccanismo di coniugazione come nei gram-negativi, fu usata la trasduzione generalizzata per costruire una mappa grossolana del cromosoma. D'altra parte i gram-positivi (Bacillus subtilis e Streptococcus) hanno una ‛competenza' naturale (che E. coli non ha) a ricevere plasmidi, che è stata utilizzata per costruire una mappa genetica.
L'induzione artificiale della competenza a ricevere il DNA trasformante nei gram-negativi può essere ottenuta con cationi bivalenti (Ca2+) e shock termico. Un metodo recente è l'elettroporesi, efficace sia nei gram-positivi che nei gram-negativi con resa di 109 ÷ 1010 trasformanti per µg di DNA; questa tecnica consiste nell'applicazione di un campo elettrico ad alta potenza che produce pori nella cellula.
Con il termine ‛plasmide' Joshua Lederberg, nel 1952, ha definito un frammento di DNA extracromosomale che si replica autonomamente, di dimensioni variabili da 1 a 200 kdalton. I plasmidi dipendono dall'ospite per la loro replicazione, non sono essenziali alla sopravvivenza della cellula e hanno enorme importanza nella tecnologia del DNA ricombinante per il clonaggio di geni estranei.
d) Elementi genetici trasponibili
Alcuni segmenti di DNA della cellula batterica hanno la capacità di trasporsi in siti diversi del genoma con un sistema indipendente dalla ricombinazione classica, poiché fu osservato anche in mutanti non ricombinanti (rec-). Gli elementi genetici trasponibili (v. genetica, vol. X) possono dividersi in tre classi (v. Berg e Howe, 1989): 1) elementi semplici, come le sequenze d'inserzione (IS elements) di 800-1.500 coppie di basi, costituite da un gene per la trasposasi (l'enzima necessario alla trasposizione) fiancheggiato da sequenze di DNA terminali ripetute che servono come substrato alla trasposasi; 2) elementi trasponibili composti, chiamati trasposoni o elementi Tn, costituiti da elementi d'inserzione IS e da altri geni indipendenti dalla trasposizione (per es., geni per la resistenza ad antibiotici): questi elementi esistono in natura come parte di plasmidi; 3) batteriofagi come Mu (mutator), un virus batterico unito a un trasposone, scoperto nel 1963 come un nuovo fago che può integrarsi in molti siti del cromosoma dell'ospite.
I trasposoni si sono dimostrati estremamente utili negli studi di genetica batterica, perché quando se ne inserisce uno in un gene, questo diviene inattivo: tale inserzione è stabile, dato che l'estrusione è molto rara. Inoltre, l'inserzione esercita un forte effetto polare sui geni seguenti e può essere utilizzata per determinare la struttura dell'operone. Il trasposone inserisce nel sito prescelto geni marcatori identificabili, come quelli per la resistenza ad antibiotici.
In ceppi patogeni di Streptococcus sono stati isolati per la prima volta, nel 1981, elementi di DNA che conferivano al batterio ospite gram-positivo la capacità insolita di comportarsi come un donatore nella coniugazione batterica. Questi mutageni d'inserzione di nuovo tipo, dotati di specificità differenti dagli altri trasposoni e denominati ‛trasposoni coniugativi', possono inserirsi in un gran numero di siti diversi del cromosoma batterico e hanno proprietà in comune con gli altri trasposoni, coi plasmidi e coi fagi: infatti sono in grado d'integrarsi nel DNA, hanno enzimi integrasi ed escissionasi come i batteriofagi e i trasposoni, e possono trasferire, oltre al proprio, anche altri elementi di DNA. Dopo essere stati individuati nei batteri gram-positivi, furono riscontrati anche nei gram-negativi (Neisseria gonorrhoeae) e nei batteroidi (anaerobi gram-negativi e patogeni presenti nel colon dell'uomo): in questi ultimi mobilizzano plasmidi coresidenti e provocano escissione, circolazione e mobilizzazione di NBU (Non-replicating Bacteroides Units), ossia segmenti di DNA non legati.
I trasposoni coniugativi dei batteroidi sono particolarmente importanti, perché responsabili dell'espandersi della resistenza agli antibiotici tetraciclina, eritromicina, clamidomicina.
e) Mappa fisica del genoma batterico
Il metodo classico, tradizionale, di mappatura era basato sull'elaborata determinazione della frequenza di ricombinazione, che fornisce una misura piuttosto approssimata della distanza tra i geni (l'1% della mappa di associazione di E. coli varia tra 33 e 60 coppie di basi, bp, da base pairs). Metodi più recenti ricorrono a tecniche diverse, più precise. Per ottenere la mappa di un genoma bisogna innanzi tutto estrarre intatto il cromosoma, tagliarlo in un numero limitato di frammenti (shotgun) e quindi separare questi frammenti mediante elettroforesi su gel in campo pulsante (Pulse Field Gel Electrophoresis, PFGE; v. genoma, vol. X); questo metodo ha subito miglioramenti tecnici grazie alla disponibilità di apparecchiature sempre più sofisticate e all'evoluzione concettuale nel campo della bioinformatica, con l'uso di ‛algoritmi dinamici'.
La prima sequenza di genoma a essere completata è stata quella di Haemophilus influenzae, un batterio innocuo in laboratorio ma capace di causare infezioni all'orecchio e meningite.
Il metodo usato da Craig Venter, dell'Institute of Genomic Research, e da Hamilton Smith, della Johns Hopkins University, si è rivelato utile, in generale, anche per la ricerca su geni e su agenti eziologici di malattie. Per prima cosa questi ricercatori hanno analizzato le basi costitutive dei frammenti di geni, quindi hanno sequenziato tali frammenti e li hanno riassociati. Operando un confronto con mappe di altri organismi hanno potuto osservare, per esempio, che in H. influenzae 736 geni non avevano corrispondenti e mancavano tre dei geni del ciclo dell'acido tricarbossilico: questo spiega la necessità di fornire elevate concentrazioni di glutammato per la crescita in coltura di H. influenzae e la tendenza di tale microrganismo a incorporare DNA liberato da cellule morte della stessa specie, di cui contiene ben 1.465 copie. Queste connessioni ricavate dal mappaggio dimostrano l'utilità di tale metodo. La rapidità con cui è stata determinata la sequenza di Mycoplasma genitalium fa prevedere che sarà possibile completarne dieci ogni anno e garantisce un promettente sviluppo di nuovi trattamenti e di nuovi vaccini contro le malattie infettive, e questo è particolarmente importante dato che sono sempre più numerosi i batteri che sviluppano resistenza agli antibiotici.
f) Introni ed esoni
Gli introni sono sequenze non codificanti inserite tra quelle codificanti, gli esoni. Normalmente negli Eucarioti gli introni, trascritti nell'RNA messaggero, vengono eliminati prima della traduzione. Solo nel 1984 si scoprì che anche i Procarioti contengono introni: fu individuato un introne nel gene td del fago T4 di E. coli e, nel 1995, un altro nel gene thy nel fago β22 di B. subtilis. Il fago β22 appartiene a una vasta famiglia di fagi morfologicamente identici (ospiti di gram-positivi), molti dei quali contengono introni: uno di questi, il fago dello stafilococco, originariamente descritto da Frederick W. Twort nel 1915, contiene il più grande numero di introni sinora trovati nel genoma dei Procarioti.
Benché presenti in varie specie di batteri e di fagi, gli introni non sono tuttavia comuni in questi organismi. Il primo introne fu scoperto per caso osservando che non vi era colinearità tra sequenza del DNA e sequenza delle proteine e che le due sequenze potevano essere riconciliate se si ammetteva la presenza di una sequenza intermedia di 1.016 nucleotidi. La natura infettiva degli introni e il fatto che la loro presenza non porta alcun vantaggio selettivo, fa supporre che essi siano elementi parassiti; tuttavia la loro persistenza sia nel genoma dei Procarioti, sia nel sistema regolatore dei fagi suggerisce che possano essere elementi benefici per l'ospite del fago. Gli introni aumentano le possibilità di riarrangiamenti genetici nei fagi T pari.
g) Inteine
Il dogma centrale di Crick per la sintesi delle proteine stabilisce una colinearità tra sequenza di nucleotidi del DNA e sequenza degli amminoacidi nella proteina prodotta. Lo sviluppo delle conoscenze in questo campo permette ora di acquisire e interpretare alcuni risultati inattesi e precedentemente scartati come stranezze. Recentemente sono stati osservati geni microbici il cui prodotto è tagliato non al livello dell'RNA messaggero (come avviene nel caso degli introni), ma delle proteine. Il precursore della proteina si taglia autocataliticamente subito dopo la sintesi e forma immediatamente una proteina attiva: queste proteine, chiamate ‛inteine', agiscono come endonucleasi. Inteine sono state trovate nel gene recA di Mycobacterium tuberculosis e di Mycobacterium leprae e nel gene per la DNA-polimerasi di un Archaea termofilo sottomarino (Thermococcus litoralis).
7. Virus e batteriofagi
Il termine ‛virus', usato inizialmente come sinonimo di sostanza infettiva, si riferisce ora a organismi più piccoli dei Batteri incapaci di moltiplicarsi al di fuori di una cellula ospite vivente, cioè a organismi parassiti obbligati, definiti attualmente in base alle loro caratteristiche ultrastrutturali e alle loro modalità di replicazione. Il primo virus isolato (filtrabile) fu quello del mosaico del tabacco, a opera di Dmitrij Ivanovskij (1892) in Russia e di Martinus Beijerinck (1899) in Olanda. Si riuscì a dimostrare che virus filtrabili isolati da animali erano gli agenti eziologici di malattie del bestiame (Friedrich Löffler e Paul Frosch, 1898) e della febbre gialla studiata nell'esercito americano (1900). Questi parassiti per lo più uccidono l'ospite rapidamente, come i virus della febbre emorragica a cui l'uomo è particolarmente sensibile (Ebola, Dengue, Hantaan, ecc., rispettivamente della famiglia Filiviridae e dei generi Flavivirus e Bunyavirus), così che non potrebbero sopravvivere e propagarsi se non nel caso di epidemie infettive o di un ospite resistente (Roditori e zanzare). I virus della febbre emorragica, conosciuti con tanti nomi quante sono le località dove si è sviluppata un'epidemia, sono tutti virus a RNA trascritto dall'RNA-polimerasi: durante la trascrizione avvengono numerosi errori che provocano rapide variazioni dei virus prodotti; il loro genoma, inoltre, è segmentato e per ricombinazione nelle cellule ospiti produce ulteriori variazioni da riassortimento (v. virus, vol. VII). La causa delle epidemie va ricercata nella povertà di mezzi degli ospedali (per es., mancanza di siringhe monouso, impossibilità di procedere alla sterilizzazione) e nell'inadeguatezza dei metodi di difesa (quali, per es., camere a bassa pressione, laboratori con sicurezza biologica) per cui medici e infermieri diventano vittime del loro stesso lavoro (spesso non remunerato).
Lo spettro delle cellule ospiti che i Virus possono infettare comprende tre classi: animali, vegetali e batteriche; ogni virus è capace d'infettare una sola classe specifica di cellule. Naturalmente molto è stato scritto nell'ultimo ventennio sul virus pandemico HIV (Human Immunodeficiency Virus) che provoca l'AIDS (Acquired Immuno-Deficiency Syndrome) e sui virus oncogeni che provocano il cancro nell'uomo.
Di tutta la vasta area di studi relativi ai Virus, caratterizzata in questi anni da una crescita esponenziale e ampiamente trattata in capitoli specializzati, ci riferiamo qui solo a quanto riguarda il rapporto Virus-Batteri, cioè ai batteriofagi e ai ceppi di batteri lisogeni. Virus che infettano i Batteri erano stati scoperti indipendentemente da F. W. Twort nel 1915 in Inghilterra e da Félix d'Hérelle nel 1917 in Francia. L'importanza di questo argomento è stata sottolineata nel convegno, tenutosi a Cold Spring Harbor nel 1995, per il cinquantesimo anniversario del primo corso sui fagi.
Lo studio quantitativo della genetica e della biologia dei fagi iniziò quando fu possibile ottenerli come placche di lisi in uno strato di batteri coltivati su terreno solido, e in particolare con l'esperimento di Max Delbrück (v. Ellis e Delbrück, 1939) sulla moltiplicazione del fago T4 in E. coli. Nella vita del fago si possono distinguere tre fasi: adsorbimento, moltiplicazione e liberazione; i fagi virulenti infettano la cellula e uccidono l'ospite per lisi; i fagi temperati possono seguire la via della lisogenia e cioè inserire il loro DNA nel cromosoma dell'ospite.
In questi ultimi anni, grazie alle conoscenze sulla fisiologia e sulla genetica dei fagi e agli sviluppi della biologia molecolare, ci si è resi conto delle possibilità che offre l'utilizzazione di tali virus. I fagi rappresentano attivi vettori per l'ingegneria genetica e permettono la traduzione di caratteri genetici in cellule non omologhe. È stata costruita una serie di plasmidi vettori chimerici di micobatteriofagi (L5 e D29) che portano inseriti cosmidi di E. coli: questi vettori si replicano nei micobatteri come fagi e in E. coli come cosmidi. Un cosmide tipico è un plasmide circolare (ColE, resistenza alla colicina) nel quale sono stati inseriti, per esempio, un gene per la resistenza alla rifampicina e un sito (cos) del fago λ: queste proprietà combinate rendono i cosmidi particolarmente adatti per l'utilizzazione in ingegneria genetica. Con questi vettori è stato possibile introdurre nei micobatteri un gene sensibile alla temperatura che provoca la morte della cellula.
8. Archaea
Archaea rappresenta un gruppo tassonomico unico di organismi che deve essere studiato indipendentemente dai Procarioti, in quanto costituisce un'insospettata terza forma di vita: più vicini agli Eucarioti, Archaea non sono Batteri e il loro studio è utile per comprendere la natura e l'evoluzione delle cellule eucariote. Archaea sono anaerobi termofili che presentano, a livello molecolare, proprietà analoghe a quelle degli Eucarioti, con segnali e fattori di trascrizione, chaperons e istoni, ma come i Procarioti mancano di membrana nucleare; per la predilezione che mostrano verso ambienti estremi (alta temperatura, alta concentrazione di sali, anaerobiosi totale e uso di metano come sorgente di energia) il loro studio richiede sistemi e apparecchiature speciali. Questo argomento è ampiamente trattato nei tre volumi recentemente pubblicati da Frank T. Robb (v., 1995).
Esistono resti fossili di cellule procariote risalenti a più di 3 miliardi di anni fa; poiché la Terra si è raffreddata a temperatura compatibile con la vita solo 4 miliardi di anni fa, si è indotti a ritenere che a quell'epoca si siano formate cellule indistinguibili da Eubatteri e da Archaea e che da questo progenitore comune, chiamato Progenote, Archaea si siano evolutivamente diramati in epoca molto antica.
Archaea thermoplasma contiene una singola specie di piccole proteine basiche Hta simili agli istoni, in grado di formare complessi simili a nucleosomi contenenti una struttura centrale (core) fatta di 4 molecole di proteine Hta attorno a cui si avvolgono 40 coppie di basi di DNA.
9. Biotecnologia
a) Introduzione
La biotecnologia ha avuto origine con la rapida applicazione di risultati sperimentali a tecnologie industriali o biologiche. Già migliaia di anni fa il procedimento dell'ibridazione aveva permesso di selezionare prodotti agricoli o animali più redditizi, ma è stato con l'introduzione di metodi scientifici radicalmente nuovi che si è avuta recentemente una vera rivoluzione in questo campo (v. biotecnologie, voll. VIII e X). Alla base di tale progresso scientifico sono le conoscenze acquisite sulla struttura del DNA e sul rapporto DNA-proteine, la realizzazione della mappatura genica e la scoperta degli enzimi di restrizione, che hanno reso possibile tagliare e ricostituire il DNA in modo deliberato e sostituire geni (per ora solo animali e vegetali, ma, in un futuro forse lontano e certamente non desiderabile, anche nell'uomo). Queste applicazioni prospettano problemi etici e impongono la necessità d'introdurre delle regole (v. bioetica, vol. X).
b) Microrganismi produttori di sostanze chimiche di interesse industriale
Procedimenti naturali di fermentazione e sostanze chimiche di base sono stati usati per secoli, o addirittura millenni, da un'industria ancora rudimentale senza che si fosse in grado di darne una spiegazione scientifica. La scoperta che tanti processi sono dovuti a microrganismi si deve a Pasteur: da allora la fermentazione è diventata una scienza e gli agenti specifici della fermentazione alcolica, butirrica, lattica, ecc. sono stati individuati e utilizzati in campo industriale.
La conoscenza approfondita delle diverse fasi della fermentazione ha permesso anche l'utilizzazione di prodotti intermedi (per esempio amminoacidi, vitamine, acido citrico, acetico, lattico, ecc.) e di sostanze chimicamente modificate (come steroidi, sorbitolo, ecc.). Le fermentazioni microbiologiche portano alla produzione industriale di sostanze non solo organiche, ma anche inorganiche: ad esempio, Clostridium acetobutylicum, un anaerobio obbligato, a partire da amido, melassa o cellulosa produce per fermentazione etanolo, butanolo, acetone, acido butirrico, acido acetico, isopropanolo, e inoltre CO2 e H2 come prodotti intermedi. Alcuni ceppi batterici fermentanti (come Brevibacterium flavium, Corynebacterium glutamicum, Microbacterium ammoniaphylum, Brevibacterium lactofermentum) sono produttori di amminoacidi. La fermentazione naturale ha sostituito in molti casi la sintesi chimica, in quanto la resa è molta elevata (quasi del 100%).
A uno dei problemi inerenti ai procedimenti ora citati, quello della contaminazione, si è recentemente ovviato, sulla scorta dei risultati delle ricerche scientifiche, abbreviando il processo di fermentazione e diminuendo così la probabilità d'inquinamento delle colture. Selezionando organismi appropriati e inducendo o attivando proteine specifiche si può ottenere un incremento della produttività: si riesce, per esempio, a ottenere una grande quantità di prodotto da cellule al massimo della loro curva di crescita, praticamente alla fine del loro ciclo riproduttivo.
L'industria farmaceutica ha iniziato, con la scoperta della penicillina a opera di Fleming nel 1928, la produzione di una vasta gamma di antibiotici microbici commerciali, il cui numero è attualmente di oltre 2.000. Questa grande varietà aiuta a prevenire l'insorgere della resistenza da parte dei germi. Sempre per via microbica (Streptomyces spp., Streptomyces olivoreticuli, Pseudomonas spp., Bacillus cereus, ecc.) si possono produrre industrialmente agenti immunomodulatori, sostanze antidepressive e antinfiammatori; mediante le tecnologie del DNA ricombinante è ora possibile introdurre in E. coli i geni produttori dell'insulina e ottenerne la produzione industriale. Si ritiene che in breve tempo sarà possibile ottenere per questa via anche la produzione di polipeptidi, come l'ormone umano della crescita, e di proteine di interesse medico, come il fattore VIII della coagulazione per la terapia dell'emofilia e come l'interferone.
Molto importante è la produzione di ormoni steroidi da parte di microrganismi: con l'impiego di Corynebacterium si è arrivati, a partire da steroli delle piante, alla sintesi di un derivato del cortisone, il prednisolone, usato come antinfiammatorio; Arthrobacter simplex trasforma l'idrocortisone in prednisolone. L'impiego di microrganismi rende possibili reazioni che sarebbero difficili o impossibili con mezzi chimici: mediante l'impiego di Acinetobacter HOI-N è possibile ottenere la polimerizzazione di esadecani in modo più economico che per estrazione da fonti naturali (jojoba e altre piante, balene). Se il materiale di partenza per queste sintesi è l'acido acetico, si ottengono catene a numero pari di atomi di carbonio; se è invece l'acido propionico, si ottengono catene a numero dispari di atomi di carbonio.
Alcuni microbi producono pigmenti, agenti aromatici, enzimi inibitori, erbicidi, insetticidi, ormoni delle piante e agenti emulsionanti. Inoltre, è possibile ottenere dai Batteri, che hanno un alto contenuto di proteine (72%), alimenti utilizzabili per la nutrizione degli animali, specie nelle zone del mondo nelle quali è impossibile, o di costo troppo elevato, la produzione di soia e di polvere di pesce.
c) Lisciviazione batterica e bioestrazione
Fin dal 1000 a.C. è diffusa nel bacino del Mediterraneo la tecnica di ricavare i minerali setacciando le acque di drenaggio delle miniere e dei fiumi, ma solo nel 1920 si è scoperto che si deve ai Batteri la possibilità di lisciviare il solfuro di zinco e il solfuro di ferro. Furono A. Colmer e M. E. Hinkle a individuare, nel 1947, Thiobacillus ferrooxidans, responsabile della lisciviazione di solfuri metallici. La lisciviazione batterica è ora largamente usata in tutto il mondo per ottenere rame, uranio, cobalto, zinco, piombo e oro. Si prevede che la tecnica della bioestrazione verrà sempre più largamente utilizzata per gli enormi vantaggi che presenta: non richiede l'impiego di alcun tipo di energia, elimina la necessità di scavare miniere profonde e di rimuovere enormi quantità di rocce, rende possibile utilizzare anche giacimenti piccoli o poco accessibili, infine non provoca alcun inquinamento o alterazione dell'ambiente. Gli organismi più importanti per questo scopo sono Archaea: Thiobacillus ferrooxidans, T. thiooxidans, Leptospirillum ferrooxidans, e specie del genere Sulfolobus. È interessante notare come questi organismi ricavino energia dalle reazioni: ione ferroso → ferrico e zolfo ridotto → acido solforico; inoltre T. ferrooxidans è azotofissatore, cioè trasforma l'azoto atmosferico in ammoniaca. Questi organismi in colture miste incrementano la loro attività lisciviante e si pensa che le ricerche d'ingegneria genetica recentemente iniziate e attualmente in corso possano ottenere ulteriori miglioramenti.
d) Bioeliminazione dell'inquinamento ambientale
Ancora una volta è l'ingegneria genetica che viene in soccorso all'uomo per ovviare a uno dei più gravi problemi connessi con lo sviluppo industriale e con l'aumento della popolazione: l'inquinamento ambientale. Alla soluzione di questo problema è legata in parte la possibilità di sopravvivenza delle specie viventi. La scienza ha messo a punto nuovi microrganismi, programmati per eliminare anche in condizioni sfavorevoli sostanze inquinanti o non degradabili: si sono ottenuti buoni risultati introducendo stabilmente nei cromosomi batterici, mediante mini-trasposoni, geni stabili capaci di trasformare sostanze tossiche in carbonio innocuo, risolvendo così, almeno in parte, il problema dell'inquinamento conseguente al funzionamento di inceneritori e discariche.
e) Metodo microbiologico per il controllo degli Insetti
La lotta ai parassiti di animali e piante è stata condotta, fino a pochi anni fa, prevalentemente con l'utilizzazione di varie sostanze chimiche (v. pesticidi, voll. V e XI). Questo metodo, però, è in grado di provocare danni agli animali e all'uomo, e può causare la selezione di specie resistenti contro le quali non è facile trovare metodi di lotta efficaci; inoltre, la degradazione di pesticidi quali il DDT può portare alla formazione di prodotti ancora più dannosi (DDD) e alla distruzione di specie biologiche utili, come per esempio le api. L'inquinamento ambientale ha raggiunto valori preoccupanti: si sono trovate tracce di DDT persino nelle nevi dei ghiacciai ad alta quota.
L'introduzione della lotta biologica è stata seguita da risultati molto promettenti (v. lotta biologica, vol. III) e già da diversi anni sono impiegati a questo scopo alcuni batteri, come Bacillus thuringiensis, il quale durante la sporulazione produce un cristallo di una tossina che uccide le larve di oltre cento specie di Lepidotteri e Ditteri; Pseudomonas syringae produce una proteina che induce sulle foglie la formazione di cristalli di ghiaccio a una temperatura fra 0 e 2 °C con conseguente danno alle colture; un suo mutante, P. syringae ice-minus, costruito geneticamente, fa abbassare la temperatura di congelamento a - 8 ÷ - 6 °C. Il rilascio nell'ambiente di microrganismi modificati geneticamente non è però privo di pericoli: alterazioni cromosomiche, mutazioni o eliminazione di specie esistenti possono alterare il normale equilibrio biologico e favorire lo sviluppo di germi pericolosi e non controllabili, con conseguenze preoccupanti.
f) Individuazione rapida di batteri patogeni
Le tecniche diagnostiche comunemente usate in laboratorio per identificare i microrganismi, anche se molto precise e affidabili, sono laboriose e richiedono tempi non brevi; esse pertanto non risultano adatte nei casi in cui è indispensabile individuare l'agente patogeno in tempi molto rapidi, onde mettere in atto tempestivamente una terapia appropriata dalla quale dipende la vita o la morte di una o più persone. In questo campo si sono registrati progressi nella diagnosi d'infezioni da alimenti, utilizzando metodi basati sull'analisi dell'acido nucleico e sull'uso di anticorpi, sensibili a quantità minime dei prodotti batterici; si tratta di tecniche analitiche che possono essere impiegate su campioni di dimensioni molto ridotte, utilizzando attrezzature che permettono di effettuare esami multipli in tempi assai brevi, così che l'individuazione dei microrganismi patogeni risulta rapida e precisa.
La bioluminescenza, che tra i metodi di più largo impiego è uno dei più sensibili (103 ÷ 104 batteri per pozzetto di conta) e dei più rapidi (richiede solo pochi minuti), consiste nel misurare i fotoni liberati dall'ossiluciferina per azione dell'enzima luciferasi, che è proporzionale alla concentrazione di ATP nella reazione (v. bioluminescenza, vol. I). Un altro metodo si basa sulla specificità della reazione antigene-anticorpo e sulla sensibilità delle tecniche impiegate per metterla in evidenza, e cioè l'immunofluorescenza e l'ELISA (Enzyme-Linked Immunosorbent Assay): l'immunofluorescenza consiste nel rendere intensamente fluorescenti, mediante coniugazione chimica con un colorante fluorescente, gli anticorpi serici di un animale infetto, che in tal modo, combinandosi con l'antigene specifico, formano un complesso fluorescente che rende possibile riconoscere un batterio anche in una popolazione batterica mista; l'ELISA misura le molecole ibride anticorpo-antigene con saggi enzimatici quantitativi di immunoadsorbimento, utilizzando anticorpi legati covalentemente a un enzima (perossidasi del rafano, fosfatasi alcalina, o β-galattossidasi; v. immunologia clinica e immunopatologia, vol. VIII). Usando anticorpi monoclonali ad alta specificità è possibile rilevare la presenza di tossine batteriche negli alimenti (per esempio, enterotossina di Staphylococcus aureus). In casi particolari, per i quali il saggio immunologico non risulta sufficientemente sensibile (come per Clostridium botulinum, che produce sette potenti neurotossine immunologicamente differenti), si preferisce ricorrere a una prova biologica molto sensibile, come l'inoculazione nel topo.
Quantità infinitesime di DNA di organismi patogeni possono essere rilevate usando la reazione di polimerizzazione a catena (PCR) di sequenze specifiche di DNA: i prodotti della PCR sono misurabili con metodi colorimetrici e di chemioluminescenza. Col metodo IMS (Immunomagnetic Separation) si possono separare e identificare microbi in campioni di cibo: tale metodo, che utilizza particelle supermagnetiche ricoperte da anticorpi che si legano agli antigeni della superficie batterica, non è però utilizzabile nel caso in cui nel campione di cibo siano presenti tossine preesistenti alla contaminazione batterica.
10. Interazione di piante e microrganismi
a) Premessa
Le piante vivono in un ambiente estremamente ricco di microrganismi coi quali interagiscono: alcuni sono parassiti e ricavano dalle piante energia e nutrimento, altri scambiano con esse segnali biochimici e genetici che inducono meccanismi di difesa da parte delle piante stesse.
Gli agrobatteri sono molto diffusi nel suolo e provocano alterazioni delle radici o del fusto di molte famiglie di piante dicotiledoni. Agrobacterium tumefaciens causa la formazione di galle e tumori (v. botanica, vol. X), mentre A. rhizogenes provoca una crescita abnorme dell'apparato radicale. Studi recenti hanno permesso di stabilire i meccanismi genetici di questi due ceppi batterici. Il meccanismo genetico che consente ad A. rhizogenes di trarre vantaggio dall'aumentata crescita delle radici, dalle quali ricava energia, consiste nell'inserire stabilmente il T-DNA del plasmide Ti (Tumor inducing) nel genoma della pianta: questa trasformazione avviene in natura, e infatti sono stati trovati plasmidi portatori del gene Ri (Root inducing) in piante normali. Agrobacterium tumefaciens introduce nella pianta un frammento di DNA oncogeno portato dal plasmide Ti che induce la proliferazione delle cellule vegetali; l'infezione avviene attraverso una ferita della pianta, e la crescita batterica è la conseguenza della sintesi di auxine, di citochine e di composti specifici detti opine (amminoacidi guanidinici) da parte del tumore.
b) Resistenza delle piante all'attacco di microrganismi
Le piante, pur mancando di un sistema immunitario, hanno messo a punto un meccanismo di difesa espresso da geni di resistenza (R genes): questi geni, la cui esistenza era stata ipotizzata sulla base dei processi d'ibridazione realizzati dagli agricoltori, sono stati isolati e clonati solo dal 1992.
Il gene, o fattore, di resistenza R riscontrato nelle piante produce proteine con proprietà comuni in varie specie e attive per la resistenza a una vasta gamma di patogeni. Il meccanismo di difesa della pianta, molto complesso, inizia con un segnale molecolare prodotto dal patogeno e riconosciuto dalla pianta: è stato identificato finora un solo segnale molecolare di virulenza di un patogeno, il locus avrD (avirulence D) di Pseudomonas syringae, che produce enzimi per la sintesi di sostanze chimiche in grado di indurre ipersensibilità (HR, Hypersensitivity Reaction) e quindi difesa di tipo immunitario nei semi di soia portatori del gene R. La selezione di piante portatrici di una miscela di geni R permetterebbe di ottenere colture più resistenti, limitando l'uso dei pesticidi.
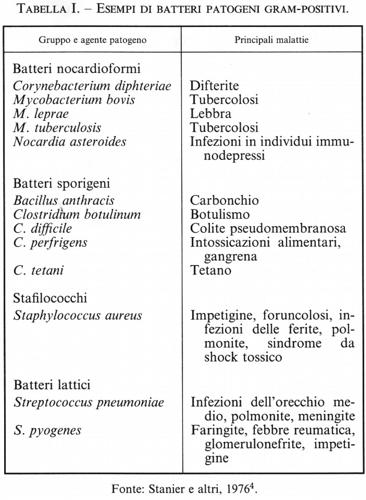
11. Azione patogena dei Batteri
a) Premessa
L'esistenza, dimostrata dalle acute osservazioni di scienziati quali Pasteur e Koch, di un rapporto diretto tra agenti patogeni e malattie ha rappresentato la pietra miliare che ha dato inizio a due branche fondamentali della scienza: la microbiologia e la medicina moderna. Senza le conoscenze microbiologiche non sarebbe mai stato possibile sviluppare metodi di prevenzione (vaccinazioni) e di difesa nei confronti dei germi patogeni. La farmacologia, passata dall'empirismo allo studio di farmaci capaci di uccidere i germi (molti antibiotici) o di bloccarne la crescita (sulfamidici e alcuni antibiotici), si avvale ora anche degli studi di biologia molecolare per intervenire specificamente nel ciclo biologico della loro azione patogena.
Sembrerebbe dunque logico ritenere che la percentuale di malattie e di morti sia molto diminuita rispetto a un secolo fa. Ci si accorge invece con stupore che è rimasta pressappoco uguale; malattie che sembravano debellate, come la tubercolosi, ricompaiono con aumentata aggressività (v. malattie emergenti, vol. XI). La capacità dei Batteri di mutare, la sopravvivenza di ceppi resistenti, la comparsa di nuove forme patogene rendono necessaria la messa a punto di sempre nuove strategie difensive. Responsabili di molte patologie sono i Virus, dei quali non si sospettava neppure l'esistenza fino agli ultimi anni del XIX secolo.
Una maggiore conoscenza delle interazioni ospite-batterio può favorire lo sviluppo di metodi di difesa più efficaci e forse più generalizzati, o di migliori vaccini. Studi recenti hanno dimostrato, per esempio, che tra le difese dell'organismo umano va annoverata quella messa in atto dai villi intestinali che producono peptidi, detti criptidine e difensine, tossici per clostridi e altri microbi.
b) Sistematica dei batteri patogeni
I batteri patogeni possono essere classificati in diversi modi a seconda della forma, della presenza di capsule, della formazione di spore, ecc. Finora la classificazione più usata è stata quella basata sulla loro reazione alla colorazione di Gram. La tassonomia moderna usa lo schema filogenetico, e anche per i batteri patogeni è stata recentemente proposta una classificazione di questo tipo, per la quale si è presa in considerazione la sequenza dell'rRNA 16S; risultati simili sono però conseguibili anche servendosi di altri geni costantemente conservati, quali quelli dell'rRNA 23S. Nella figura la lunghezza delle linee orizzontali indica la distanza genetica tra geni differenti e le ramificazioni indicano la parentela, più o meno stretta, tra generi. Se in passato si fosse seguito questo criterio di classificazione, i generi Escherichia, Shigella e Salmonella avrebbero costituito tre specie di un unico genere. È interessante notare che, nonostante le diversità genetiche, gli stessi tipi di fattori di virulenza sono presenti in più gruppi filogenetici, per esempio le capsule antifagocitiche compaiono tanto nel gruppo gram-negativo (Escherichia) che nel gruppo gram-positivo.
Le adesine, piline e non piline, si ritrovano praticamente in tutti i gruppi. Nelle tabb. I e II sono indicati alcuni dei batteri patogeni più comuni: a questi vanno aggiunti nuovi batteri prima sconosciuti, come gli agenti eziologici della malattia dei legionari (Legionella pneumophila, che vive nel terreno e nell'acqua), della malattia di Lyme (Borrelia burgdorferi), delle sindromi da shock tossico. Nessuno, fino a pochi anni fa, sapeva che le ulcere dello stomaco sono provocate da Helicobacter pilori (v. malattie emergenti, vol. XI). Una maggiore conoscenza dell'interazione molecolare batterio-ospite può certamente favorire lo sviluppo di metodi di difesa più efficaci.
c) Relazione ospite-batterio
Qual è la strategia messa in atto da un batterio per esplicare la propria virulenza? Il primo passo è rappresentato dalla sua adesione alla superficie della cellula ospite, realizzata da ogni patogeno in un suo modo specifico. Il meccanismo più conosciuto è quello mediante fimbrie dette ‛piline'; contro la formazione di queste strutture, la cellula ospite può produrre anticorpi antipili. Alcuni batteri si attaccano all'ospite direttamente, mediante proteine della loro membrana denominate ‛adesine'. Questi meccanismi d'adesione, ben conosciuti, sono specifici per i vari tessuti e caratteristici della maggior parte dei gram-negativi; quello operante nei gram-positivi, invece, è ancora ignoto. In alcuni casi il contatto ospite-batterio è assicurato da un collante di polisaccaridi, detto ‛biofilm'. Il contatto provoca un cambiamento nel citoscheletro della cellula ospite e il conseguente inglobamento del batterio in una vescicola interna. Se le difese cellulari non sono adeguate, il batterio ha la possibilità di rompere la vescicola che l'ingloba e di moltiplicarsi nel citoplasma. A questo punto, poiché la sua sopravvivenza è legata alla capacità d'ingannare le difese dell'ospite, il patogeno può disporre di molti meccanismi di resistenza, tra i quali: la presenza di una capsula esterna, che in alcuni casi mima la composizione del tessuto dell'ospite; la secrezione di proteine che interferiscono con le funzioni cellulari dell'ospite; un processo di mimetismo antigenico, innescato dall'espressione in superficie di molecole antigenicamente simili a macromolecole dell'ospite; l'inibizione della produzione di anticorpi specifici da parte dell'ospite; la capacità di mimetizzarsi alterando la propria superficie o ricoprendosi di proteine dell'ospite, come per esempio, la fibronectina.
Da quanto sinora detto sembrerebbe che la lotta batterio-ospite sia assolutamente persa per quest'ultimo. Occorre dunque considerare anche le difficoltà che il batterio è costretto ad affrontare per svolgere la sua azione patogena: infatti deve non solo combattere contro le difese immunitarie, ma sopravvivere alla competizione con la microflora simbionte e superare le barriere opposte alla sua penetrazione (mucose) nonché altri ostacoli come il pH dell'ambiente, la pressione osmotica, la temperatura, la concentrazione di ossigeno. Il patogeno, inoltre, deve disporre dei nutrienti indispensabili, come ad esempio il ferro, la cui concentrazione nei tessuti umani è di 10-18 M, mentre i batteri ne richiedono 10-8 M: alcuni batteri secernono molecole, dette siderofori, capaci di legarsi al ferro e ai recettori specifici della membrana esterna; altri invece posseggono proteine di superficie capaci di rimuovere il ferro direttamente dalle proteine dell'ospite che lo contengono (transferrina, lattoferrina, ferritina ed emina).
d) Fattori di virulenza
La virulenza di molti batteri è dovuta alla produzione di tossine (proteine o lipopolisaccaridi) che possono essere escrete nell'ambiente (esotossine) o contenute nel batterio stesso (endotossine) e rilasciate solo alla sua lisi. Alcune tossine agiscono su molti tipi di cellule, altre su tessuti specifici (neurotossine, epatotossine; v. tossine, vol. VII).
Alcune malattie letali sono provocate dalle tossine liberate dai batteri nel terreno di crescita, che può essere rappresentato anche da alimenti: per esempio, E. coli virulento O157::H7 ha determinato la morte di persone che avevano mangiato carne tritata contaminata. L'avvelenamento da cibi può essere causato anche da altri enterobatteri (Salmonella typhimurium, Shigella spp.), da stafilococchi e da clostridi (Clostridium perfringens e C. botulinum): Staphylococcus aureus produce un'enterotossina; C. perfringens produce la tossina CPE che altera la permeabilità della membrana cellulare, la tossina teta che induce shock con azione indiretta e la tossina alfa che sopprime direttamente la contrattilità del cuore. Oltre che con le tossine, il patogeno attacca l'ospite anche con altri mezzi, quali enzimi idrolitici che lisano i tessuti. Recentemente è stata individuata una regolazione trascrizionale e post-trascrizionale dei geni stessi della virulenza che fa ritenere che tale caratteristica sia legata a meccanismi genici di controllo: amplificazione del gene, riarrangiamento della sua struttura e velocità di trascrizione.
BIBLIOGRAFIA
Alonso De Velasco, E., Verheul, A. F. M., Verhoef, J., Snippe, H., Streptococcus pneumoniae: virulence factors, pathogenesis, and vaccines, in ‟Microbiological reviews", 1995, LIX, pp. 591-603.
Belfort, M., Phage T4 introns: self-splicing and mobility, in ‟Annual review of genetics", 1990, XXIV, pp. 363-385.
Berg, D. E., Howe, M. M. (a cura di), Mobile DNA, Washington 1989.
Blair, D. F., How bacteria sense and swim, in ‟Annual review of microbiology", 1995, IL, pp. 489-522.
Brock, T., The road to Yellowstone Park and beyond, in ‟Annual review of microbiology", 1995, IL, pp. 21-29.
Caldwell, D. R., Microbial physiology and metabolism, Dubuque, Ia., 1995.
Clyman, J., Some microbes have splicing proteins, in ‟American Society for Microbiology news", 1995, LXI, pp. 344-346.
Darnell, J., Lodish, H., Baltimore, D., Molecular cell biology, New York 19902.
Davies, J., Vicious circles: looking back on resistance plasmids, in ‟Genetics", 1995, CXXXIX, pp. 1465-1468.
Davis, B., Dulbecco, R., Eisen, H. N., Ginsberg, H. S., Microbiology, Philadelphia, Pa., 19904.
Dawes, I. W., Sutherland, I. W., Microbial physiology, Oxford 19922.
Edwards, C. (a cura di), Microbiology of extreme environments, New York 1990.
Ellis, E. L., Delbrück, M., The growth of bacteriophage, in ‟Journal of general physiology", 1939, XXII, pp. 365-384.
Fleischmann, R. e altri, Whole-genome random sequencing and assembly of Haemophilus infuenzae Rd, in ‟Science", 1995, CCLXIX, pp. 496-512.
Freifelder, D., Molecular biology, Boston 19872.
Gerhardt, P. (a cura di), Methods for general and molecular bacteriology, Washington 1994.
Hobot, J., Carlemalm, E., Villiger, W., Kellenberger, E., Periplasmic gel: new concept resulting from the reinvestigation of bacterial cell envelope ultrastructure by new methods, in ‟Journal of bacteriology", 1984, CLX, pp. 143-152.
Holt, J. G., Krieg, N. R., Sneath, P. H. A., Staley, J. T., Williams, S. T. (a cura di), Bergey's manual of determinative bacteriology, Baltimore, Md., 19949.
Holzmann, D., Bacteria in fungi may be organelle precursors, in ‟American Society for Microbiology news", 1996, LXII, pp. 621-622.
Howe, C., Gene cloning and manipulation, Cambridge 1995.
Hunter, P. A., Darby, G. K., Russell, A. D. (a cura di), Fifty years of antimicrobials: past perspectives and future trends, Cambridge 1995.
Lewin, B., Genes V, Oxford 1995.
Maloy, S. R., Cronan, J. E. Jr., Freifelder, D., Microbial genetics, Boston 19942.
Monod, J., The growth of bacterial cultures, in ‟Annual review of microbiology", 1949, III, pp. 371-394.
Nash, C. (a cura di), Microbial metabolites, Dubuque, Ia., 1995.
Neidhardt, F. C., Ingraham, J. L., Schaechter, A. M., Physiology of the bacterial cell: a molecular approach, Sunderland, Mass., 1990.
Nester, E. W., Roberts, C. E., Pearsall, N. N., McCarthy, B. J., Microbiology, Orlando, Flo., 19782.
Pace, N. R., New perspective on the microbial world: molecular microbial ecology, in ‟American Society for Microbiology news", 1996, LXII, pp. 463-470.
Piggot, P. J., Bylund, J. E., Higgins, M. L., Morphogenesis and gene expression, in Regulation of bacterial differentiation (a cura di P. J. Piggot, C. P. Moran Jr. e P. Youngman), Washington 1994, pp. 113-137.
Potrykus, I., Spangenberg, G. (a cura di), Gene transfer to plants, Berlin-New York 1995.
Robb, F. T. (a cura di), Archaea: a laboratory manual, Plainview, N. Y., 1995.
Salyers, A. A., Shoemaker, N., Stevens, A. M., Li, L.-Y., Conjugative transposons: an unusual and diverse set of integrated gene transfer elements, in ‟Microbiological reviews", 1995, LIX, pp. 579-590.
Salyers, A. A., Whitt, D. D., Bacterial pathogenesis: a molecular approach, Herndon, Va., 1994.
Schlegel, H. G., Schmidt, K., Allgemeine Mikrobiologie, Stuttgart 19927.
Schleif, R., Genetics and molecular biology, Baltimore, Md., 19932.
Scott, J. R., Conjugative transposons, in B. subtilis and other Gram-positive bacteria: biochemistry, physiology and molecular genetics (a cura di A. L. Sonenshein), Washington 1993, pp. 597-614.
Setlow, P., I will survive: protecting and repairing spores DNA, in ‟Journal of bacteriology", 1992, CLXXIV, pp. 2737-2741.
Setlow, P., Mechanisms for the prevention of damage to DNA in spores of Bacillus species, in ‟Annual review of microbiology", 1995, IL, pp. 29-54.
Shapiro, J., The significances of bacterial colony patterns, in ‟BioEssays", 1995, XVII, pp. 597-607.
Shub, D., Goodrich-Blair, A., Protein introns: a new home for endonucleases, in ‟Cell", 1992, LXXI, pp. 183-186.
Stanier, R. Y., Adelberg, E. A., Ingraham, J., The microbial world, Englewood Cliffs, N. J., 19764 (tr. it.: Il mondo dei microorganismi, Bologna 1985).
Staskawicz, B., Ausubel, F. M., Baker, B. J., Ellis, J. G., Jones, J. G., Molecular genetics of plant disease resistance, in ‟Science", 1995, CCLXVIII, pp. 661-667.
Swaminathan, B., Feng, P., Rapid detection of food-borne pathogenic bacteria, in ‟Annual review of microbiology", 1994, XLVIII, pp. 401-426.
Torno, A., Almiron, M., Kolter, R., SurA, an E. coli gene essential for survival in stationary phase, in ‟Journal of bacteriology", 1990, CLXXII, pp. 4339-4347.
Tortora, G. J., Funke, B. R., Case, C. L., Microbiology: an introduction, Reading, Mass., 19924.
Watson, J. D., Tooze, J., Kurtz, D. T., Recombinant DNA: a short course, New York 19832.
Woese, C., Bacterial evolution, in ‟Microbiological reviews", 1987, LI, pp. 221-271.
Woese, C., Prokaryote systematics: the evolution of a science, in The Prokaryotes: a handbook on the biology of Bacteria. Ecophysiology, isolation, identification, applications (a cura di A. Balows e altri), Berlin-New York 1992, vol. I, pp. 3-18.
Woods, D., Rawling, D. E., Bacterial leaching and biomining, in A revolution in biotechnology (a cura di J. L. Marx), Cambridge 1989, pp. 82-91.
Yanofsky, C., Ito, J., Horn, V., Amino acid replacements and the genetic code, in ‟Cold Spring Harbor symposia on quantitative biology", 1966, XXXI, pp. 151-162.