Cellula. Movimento cellulare
Cellula. Movimento cellulare
Il movimento e la capacità di riprodursi sono le due caratteristiche che si associano più comunemente alla vita. A differenza degli oggetti inanimati, gran parte degli organismi viventi si muove intenzionalmente in cerca di ambienti ospitali, di cibo, di partner e si allontana da ambienti e situazioni che ne minacciano l'esistenza. È vero che taluni organismi potrebbero spostarsi attraverso processi di diffusione o convezione, come fanno gli oggetti inanimati e, in effetti, alcuni lo fanno. Tuttavia, una volta apparse le prime rudimentali forme di motilità, gli organismi che ne erano carenti si sono trovati in una situazione di forte svantaggio nel mondo competitivo della selezione naturale. Come si genera il movimento negli organismi viventi? I processi evolutivi naturali hanno prodotto una classe di proteine altamente specializzate per la conversione di energia chimica in lavoro meccanico. Le proprietà delle proteine motrici implicate sono decisamente molteplici ed eterogenee. Le proteine che costituiscono i motori molecolari sono state identificate, le loro sequenze amminoacidiche determinate e, in alcuni casi, si è giunti a una risoluzione dettagliata della loro struttura tridimensionale.
Oggi, con tecniche avanzate di microscopia ad alta risoluzione, è possibile studiare il lavoro compiuto (forza per spostamento) da una singola proteina motrice. Alcune di queste proteine funzionano in maniera cooperativa, altre da sole, ma tutte vanno incontro a un ciclo termodinamico che si ripete, in maniera analoga a quella di un motore a combustione interna. I requisiti per il funzionamento di un motore molecolare sono comunque assai differenti da quelli richiesti in un contesto macroscopico. Nel mondo microscopico fenomeni di surriscaldamento sono del tutto trascurabili, dato che la dispersione del calore è praticamente istantanea. Inoltre, le masse coinvolte sono minuscole e rendono irrilevante l'azione dell'inerzia e della gravità. D'altra parte, per un motore deputato a far muovere un batterio o un organulo intracellulare, l'acqua è così viscosa da essere quasi impenetrabile e l'agitazione termica (moti browniani) costituisce un impedimento non trascurabile. Nonostante ciò, i motori molecolari naturali hanno raggiunto un grado di efficienza e perfezione di gran lunga superiore alle più sofisticate macchine costruite dall'uomo. Per esempio, la chinesina, un tipico motore molecolare, ha un'efficienza del 50% e percorre in un secondo una distanza che è cento volte la propria lunghezza (paragonabile a un jet militare), e produce più potenza per unità di massa del motore di un'automobile di Formula 1. Da sempre la ricerca sui meccanismi alla base del movimento dipende fortemente dai progressi tecnologici della strumentazione utilizzata. Dato che la velocità con cui vengono introdotte innovazioni tecnologiche in questo campo è andata costantemente aumentando negli ultimi decenni, si può essere ottimisti sulla prospettiva di svelare i segreti più nascosti delle sorprendenti macchine molecolari. Vi è la fondata speranza che comprendere pienamente tali fenomeni possa rivoluzionare il modo con cui i motori costruiti dall'uomo sono concepiti e progettati.
Il motore flagellare dei batteri
Molti batteri si muovono verso specifici composti chimici, che fungono da sostanze attraenti, e si allontanano da altri, che fungono da sostanze repellenti, tramite un sistema di propulsione che ricorda la turbina di un sommergibile. Il motore è costituito dall'assemblaggio di una serie di diverse molecole proteiche, situate nella parete cellulare, che interagiscono in modo cooperativo per generare una torsione su un flagello rigido, a forma di cavatappi, che funge da propulsore. Il motore dei flagelli batterici funziona come un motore elettrico, sfruttando un flusso di ioni idrogeno (protoni) che vengono convogliati da un gradiente elettrochimico. Il potenziale elettrochimico viene mantenuto da enzimi specifici localizzati altrove nella membrana cellulare, che idrolizzano l'adenosintrifosfato (ATP) e pompano protoni all'esterno della cellula batterica. In genere il flagello ruota in senso antiorario, se osservato dal suo apice, e la velocità di rotazione è proporzionale al potenziale elettrochimico poiché il motore trasporta un numero fisso di protoni (ca. 1000) per rotazione. Tuttavia improvvise inversioni del senso di rotazione avvengono frequentemente senza modificazione della velocità.
Tali episodi alterano il normale movimento in avanti del batterio nuotatore, che a quel punto compie una serie di capriole senza meta. Questo movimento casuale può essere seguito nuovamente da un movimento direzionato ma in un altro verso. Inversioni del senso di rotazione possono anche essere ottenute sperimentalmente cambiando il senso del gradiente di concentrazione protonico o il potenziale elettrico. In assenza di un gradiente protonico non si osserva rotazione spontanea, ma in questa circostanza il motore può funzionare come una pompa protonica se una torsione esterna viene applicata al flagello. Ciò indica che il processo termodinamico coinvolto è reversibile. Questa caratteristica è peculiare del motore flagellare dei batteri e lo distingue da tutti gli altri.
Nella struttura complessiva del motore si distinguono due componenti principali: più esternamente, uno statore simile a un anello, rigidamente ancorato alla parete cellulare, e all'interno un rotore che ruota con il flagello. La torsione deriva da un'interazione tra lo statore e il rotore. La base del flagello, detta 'asta', è collegata a un filamento a forma di cavatappi con un giunto universale, denominato 'uncino'. È sorprendente che i batteri, i più semplici organismi viventi, abbiano sviluppato il motore biologico con la struttura più complessa e sofisticata. Oltre quaranta prodotti genici sono coinvolti nell'assemblaggio del motore e dell'elica, ma solo cinque di essi sono direttamente responsabili della rotazione: MotA e MotB si appaiano a formare dodici eterodimeri distribuiti simmetricamente all'interno dello statore come perle di una collana; FliG, FliM e FliN formano il cosiddetto 'complesso interruttore' nel rotore e all'interfaccia col citoplasma, e si pensa che essi determinino il senso di rotazione. Un'intricata via di trasduzione del segnale, che inizia sulla superficie cellulare tramite recettori per le sostanze chemio-attraenti o chemio-repellenti, agisce sul complesso interruttore per determinare il senso di rotazione. Si ritiene che la torsione derivi da interazioni specifiche tra il rotore e le unità generatrici di forza presenti nello statore: un'unità generatrice di forza si legherebbe consecutivamente a due o più siti (o a un semicanale continuo) sul rotore per incanalare un protone attraverso la membrana. Se la linea che congiunge questi siti di legame è obliqua rispetto all'asse del rotore, sarebbe necessaria una rotazione di quest'ultimo per completare il trasporto del protone attraverso la membrana. L'inversione del senso di rotazione richiederebbe una variazione conformazionale sulla superficie del rotore tale da ribaltare le posizioni dei siti di legame.
In alternativa, l'unità generatrice di forza potrebbe funzionare come una 'porta protonica', associandosi contemporaneamente a due adiacenti siti di legame per i protoni distribuiti come perle su un anello che circonda il rotore. Un protone potrebbe giungere a un sito di legame sul rotore entrando dalla parte extracellulare del canale, rimanendovi finché un movimento stocastico laterale di questo sito, in rapporto con l'unità generatrice di forza, lo porti a contatto con la parte citoplasmatica del canale, permettendo una fuga del protone all'interno della cellula. Un cambio direzionale del senso di rotazione richiederebbe un cambiamento conformazionale a livello dei componenti dello statore e si tradurrebbe in una configurazione invertita della porta protonica. Importanti progressi si sono dunque ottenuti per quanto concerne la conoscenza dei componenti del motore flagellare batterico, ma saranno necessarie ulteriori ricerche prima di giungere a una completa comprensione dei meccanismi di funzionamento di questo notevole sistema. La risoluzione della struttura tridimensionale delle proteine dello statore e del rotore, tramite la tecnica di diffrazione dei raggi X, potrebbe fornire preziose informazioni riguardo alle modalità di trasferimento protonico e alle interazioni fra rotore e statore. Tuttavia, per conseguire tale obiettivo sarà necessaria la cristallizzazione di MotA e MotB, compito assai complesso data la nota difficoltà di ottenere cristalli di proteine di membrana.
I motori molecolari basati sui filamenti
Tutte le cellule eucariotiche possiedono internamente un'impalcatura dinamica, il citoscheletro, costituita da vari filamenti proteici. Una delle funzioni di questi filamenti è servire da percorsi lungo i quali le proteine motrici trasportano componenti subcellulari come le vescicole o i cromosomi. Inoltre, le proteine motrici utilizzano i filamenti come cavi per trasmettere la tensione necessaria a modulare rapidamente la forma della cellula. Tutti i movimenti del corpo umano dipendono dalla presenza di oltre 10 miliardi di km di filamenti sui quali si svolge l'azione sinergica di numerose proteine motrici. Le nostre conoscenze sui motori molecolari eucariotici sono fondate su decenni di ricerche sulle basi molecolari della contrazione muscolare, che funge da paradigma generale per il funzionamento di questa classe di motori.
Miosina
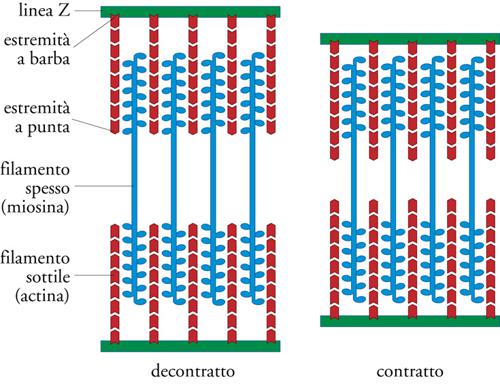
Il muscolo è un tessuto formato da grosse cellule multinucleate (sincizi) dette 'fibre', che possono svilupparsi fino a diversi centimetri in lunghezza e 100 μm in diametro. All'interno della fibra sono stipate parallelamente strutture longitudinali filamentose dette 'miofibrille'. Al microscopio ottico ciascuna miofibrilla appare internamente costituita da una sequenza, ripetuta e altamente organizzata, di bande scure e chiare. Queste ultime si accorciano durante la contrazione. Ciascun insieme di bande costituisce un'unità base contrattile, il sarcomero, e gli accorciamenti simultanei di più sarcomeri portano a una modificazione, su scala macroscopica, della lunghezza della fibra. Il sarcomero (fig. 1) è composto da un fascio quasi cristallino di filamenti molto sottili, disposti parallelamente all'asse longitudinale della miofibrilla: i filamenti sottili, ancorati ai margini del sarcomero ed estesi per circa la metà di quest'ultimo verso il centro, e i filamenti spessi, a cavallo del centro del sarcomero; entrambe le estremità dei filamenti spessi si inseriscono negli spazi tra i filamenti sottili, in una zona di sovrapposizione larga circa 0,5 μm.
Durante la contrazione i filamenti mantengono inalterata la loro lunghezza, mentre la zona di sovrapposizione tra filamenti sottili e filamenti spessi aumenta significativamente, osservazione che ha originato la 'teoria dello scorrimento dei filamenti' per la contrazione muscolare. I filamenti risultano disposti secondo uno schema esagonale, in cui ciascun filamento spesso è circondato da sei filamenti sottili, a cui sono connessi, mediante ponti trasversali nella regione di giustapposizione. Tra le molte proteine trovate nel tessuto muscolare, due sono particolarmente abbondanti: una proteina con massa molecolare 460 kDa, la miosina, e una proteina con massa molecolare 42 kDa, l'actina. Filamenti purificati di miosina si legano spontaneamente a filamenti purificati di actina; i filamenti scorrono l'uno rispetto all'altro in presenza del nucleotide adenosintrifosfato (ATP). Oggi sappiamo che la miosina è un enzima che converte l'energia chimica del legame fosforico dell'ATP in lavoro meccanico, esercitando una forza parallela su un filamento di actina.
La polarità dei filamenti è importante: la miosina (che costituisce i filamenti spessi) forma un polimero bipolare, con due metà simmetriche, composto da circa 300 molecole proteiche; al contrario, ciascun filamento di actina è unipolare, presenta siti asimmetrici di legame con la miosina e possiede estremità differenti dette 'a barba' (barbed), 'a V rovesciata', e 'a punta'. Ciascun filamento sottile nel sarcomero contiene circa 380 molecole di actina allineate in disposizione testa-coda. I filamenti di actina in regioni opposte del sarcomero sono orientati in direzioni opposte, con le loro estremità a barba ancorate al margine del sarcomero. Ciascuna metà del filamento bipolare di miosina si muove verso l'estremità a barba dell'adiacente filamento di actina, così da trascinare i filamenti di actina antiparalleli l'uno verso l'altro col risultato di un accorciamento del sarcomero. La singola molecola di miosina presenta due teste globulari ‒ responsabili del legame con l'actina e dell'attività meccano-enzimatica ‒ e una lunga coda in grado di formare oligomeri affiancandosi strettamente e sfalsandosi in modo regolare con altre molecole di miosina. La molecola di miosina è un dimero composto da due catene polipeptidiche pesanti identiche, i cui lunghi domini ad α-elica si attorcigliano a formare la coda. Le teste, ciascuna associata a una coppia di piccole catene leggere coinvolte nella regolazione enzimatica, costituiscono i ponti trasversali che connettono i filamenti di actina e miosina.
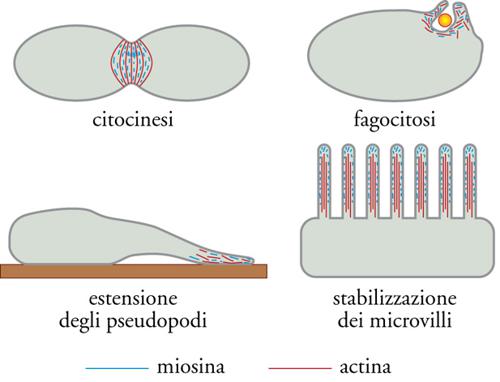
La miosina del muscolo rappresenta solo un membro della superfamiglia delle miosine. Queste sono proteine ubiquitarie nelle cellule eucariotiche. I domini motori (teste) sono altamente conservati in tutta la superfamiglia, mentre la maggior variabilità nella sequenza amminoacidica si trova a livello delle code e, presumibilmente, riflette differenze funzionali specifiche (per es., alcune di queste regioni si associano alle membrane). Alcune miosine non dimerizzano e ciò suggerisce che una sola testa è sufficiente a espletare la funzione meccanica. Tra le possibili funzioni delle miosine (fig. 2) citiamo per esempio la citocinesi, durante la quale la miosina permette la separazione delle due cellule figlie. La miosina è coinvolta anche in processi di fagocitosi; nella formazione degli pseudopodi; nella formazione dei microvilli delle cellule intestinali, o delle stereociglia, che, nell'organo di Corti del meato uditivo, sono necessarie per la risonanza delle vibrazioni acustiche. È stato anche dimostrato che alcune miosine trasportano, lungo i filamenti di actina, le vescicole intracellulari.
Chinesina
Oltre ai filamenti di actina, tutte le cellule eucariotiche contengono un altro tipo di filamenti citoscheletrici, i microtubuli; si tratta di polimeri cilindrici cavi derivanti dall'autoassemblaggio lineare di dimeri di α- e β- tubulina. Questi dimeri sono disposti in tredici file parallele, i protofilamenti, a formare la parete del cilindro; quattro volte più larghi e notevolmente più rigidi dei filamenti di actina, sono in genere disposti radialmente rispetto a un'area al centro della cellula detta 'centrosoma'. Come i filamenti di actina, i microtubuli presentano una polarità positiva e una negativa e siti di attacco per le proteine asimmetriche che vi si associano. I microtubuli hanno un ruolo centrale nell'organizzazione spaziale dell'ambiente intracellulare e fungono da arterie per il trasporto di componenti intracellulari, quali organelli o cromosomi, alle destinazioni e al momento giusti. Questi movimenti dipendono dall'attività di proteine motrici con specificità direzionale.
La chinesina è stata inizialmente identificata come l'enzima responsabile del trasporto assonale rapido in avanti (cioè verso l'estremità + del microtubulo) del corpo cellulare verso la connessione sinaptica. La geometria molecolare della chinesina è simile a quella della miosina, sebbene in una scala dimensionale leggermente inferiore (360 kDa). Si tratta di un dimero formato da due teste globulari ad attività meccano-enzimatica, una lunga coda elicoidale incappucciata da un'altra coppia di piccoli domini globulari e da una coppia di catene leggere. Si pensa che queste abbiano un ruolo nell'interazione con l'organello trasportato. La chinesina è il prototipo della superfamiglia delle chinesine. Alcune di esse si muovono esclusivamente verso l'estremità positiva del microtubulo, mentre altre si muovono verso l'estremità negativa. La maggioranza delle proteine di questa superfamiglia sembra essere coinvolta nei movimenti e nella separazione dei cromosomi. È interessante notare che, mentre le chinesine e le miosine condividono la stessa geometria generale, non c'è apparente omologia nelle sequenze amminoacidiche tra queste famiglie. È molto probabile che nell'evoluzione si siano differenziate molto presto da una protoproteina motrice, progenitrice comune di entrambe le famiglie.
Dineina
La dineina citoplasmatica è il più ubiquitario trasportatore nella direzione dell'estremità negativa. Questa proteina motrice è responsabile del trasporto assonale retrogrado, finalizzato al riciclo di vescicole che si accumulano all'estremità dell'assone per azione della chinesina. Le proteine appartenenti alla famiglia delle proteine motrici del tipo della dineina hanno anche un ruolo importante in quei dispositivi di propulsione che sono le ciglia e i flagelli (questi ultimi non hanno nulla in comune con i flagelli batterici precedentemente trattati). Queste due strutture sono lunghe e sottili protrusioni (ca. 200 nm di diametro) della superficie cellulare che s'incurvano a onda, come una frusta, con un movimento ritmico detto 'battito' (beating). Le ciglia sono relativamente brevi (ca. 10 μm) e si trovano numerose, una vicino all'altra, sulla superficie delle cellule cigliate. Esse battono all'unisono rimanendo distese durante l'oscillazione in una direzione e curvandosi flaccidamente durante il colpo di ritorno, spostando così uno strato di fluido o di particelle microscopiche in una determinata direzione rispetto alla cellula. Le ciglia hanno ruoli diversi, quali, per esempio, il movimento natatorio dell'organismo unicellulare Paramecium, il trasporto all'esterno di contaminanti intrappolati nelle mucose delle vie respiratorie e il trasporto dell'ovulo lungo le trombe di Falloppio. A differenza delle ciglia, i flagelli sono assai lunghi (fino a 500 μm), sono in genere presenti singolarmente e sono specializzati per produrre elevate velocità di propulsione di una singola cellula. Il ruolo più noto è quello della propulsione degli spermatozoi che nuotano verso un ovulo, con un movimento basato sulla propagazione di un'onda sinusoidale, come avviene per la locomozione di un serpente.
Ciglia e flagelli possiedono lo stesso tipo di struttura interna, l'assonema, un complesso altamente organizzato di microtubuli associato a diversi tipi di proteine comprendenti anche una dineina ciliare. Al suo centro vi sono due microtubuli circondati da nove doppiette dell'assonema, ciascuna delle quali è composta da una coppia di microtubuli attaccati fra loro. In ciascuna doppietta un microtubulo è completo, mentre l'altro contiene solo dieci protofilamenti. Lungo ogni tubulo A si trovano due file di dineine motrici; le teste globulari possono legarsi reversibilmente al tubulo B della doppietta adiacente e, quando ciò accade, viene esercitata una forza longitudinale che promuove lo scorrimento reciproco delle due doppiette. Poiché le doppiette sono saldamente ancorate alla loro base, questo scorrimento porta a una curvatura dell'assonema. Tutte le dineine sono complessi molecolari enormi se paragonate alla miosina e alla chinesina. Esse contengono due, in certi casi tre, catene pesanti piuttosto grandi (400÷500 kDa) composte da una testa globulare ad attività meccano-enzimatica connessa a una coda, a forma di stelo allungato, e a una gamma altamente variabile di almeno sette catene intermedie e leggere (8÷70 kDa). Nonostante la differente dimensione, la geometria generale della dineina ricorda quella della miosina e della chinesina, con più teste globulari ad attività enzimatica che fuoriescono da un comune corpo allungato.
Meccanismo di generazione della forza
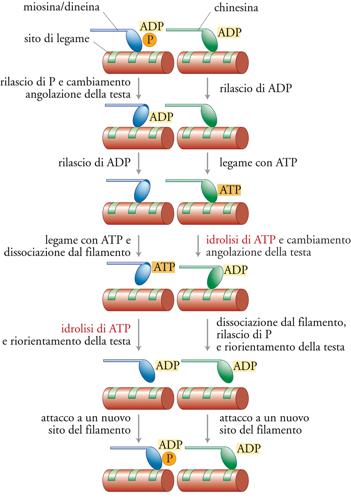
Il ruolo fondamentale dei motori molecolari è produrre lavoro, un compito che richiede generazione di forza e movimento e che necessita ovviamente di una fonte di energia. La miosina, la chinesina e la dineina ottengono questa energia catalizzando l'idrolisi del legame γ-fosforico dell'ATP. Il lavoro prodotto da una singola molecola è ottenuto cumulativamente tramite un ciclo meccano-chimico ripetuto, in cui viene idrolizzata almeno una molecola di ATP: questo provoca un microscopico evento motorio discreto lungo il filamento. L'ipotesi del ponte trasversale rotante, postulata in origine per spiegare il meccanismo della contrazione muscolare, prevede che la testa motrice della miosina, chinesina o dineina si attacchi a un sito specifico sul filamento e vada incontro a una modificazione della propria angolazione rispetto alla coda, il cosiddetto 'colpo di trazione', che trascina la coda in avanti e poi si dissocia dal filamento. Il ciclo poi ricomincia con un nuovo legame in un sito differente del filamento (fig. 3). La differenza principale è che miosina e dineina idrolizzano l'ATP dopo la dissociazione dal filamento, mentre la chinesina lo idrolizza quando è legata al filamento.
La frazione di tempo del ciclo in cui viene mantenuto il legame col filamento, detta duty ratio (rapporto di utilizzazione), è specifica per la particolare funzione di ciascun tipo di motore. Il duty ratio ha un profondo effetto sulla processività del motore, ossia la capacità di una singola molecola di percorrere grandi distanze lungo un filamento; in saggi in vitro la miosina non è risultata processiva poiché in genere si stacca dal filamento e se ne allontana dopo un singolo ciclo, mentre la chinesina, altamente processiva, va incontro a circa cento cicli prima di dissociarsi dal microtubulo. Il nucleo centrale del motore è il sito di legame per l'ATP, molto simile in tutti i motori; inoltre la sua conformazione generale assomiglia a quelle trovate nelle proteine G, deputate alla trasduzione dei segnali. Tra motori e proteine G vi sono anche notevoli somiglianze negli elementi di struttura secondaria (anse ed eliche) che servono a percepire lo stato in cui si trova il nucleotide (idrolizzato o non) e a trasmettere queste informazioni alla parte proteica deputata a mediare le interazioni con altre proteine, come quelle di un filamento. Sulla base di queste osservazioni sembra esistere un meccanismo generale fondato su un interruttore molecolare sensibile allo stato del nucleotide. Riguardo al meccanismo generale della rotazione o della curvatura su un perno intramolecolare, è utile paragonare la struttura della famiglia di motori del tipo della miosina con la famiglia delle chinesine. La struttura cristallina del nucleo delle teste motrici risulta inaspettatamente quasi identica e ciò suggerisce l'esistenza di una strategia comune per la produzione del movimento. Infine, sembra che la direzionalità sia determinata da differenze strutturali molto piccole dei motori, in quanto non sono state riscontrate importanti differenze tra le strutture cristalline della chinesina e della proteina NCD che hanno direzionalità opposte.
Polimerasi ed elicasi come motori proteici
Gli enzimi DNA- e RNA-polimerasi scorrono lungo un filamento stampo di DNA per replicarlo in un nuovo DNA o per trascriverlo in un RNA. Anche le elicasi scorrono lungo il DNA precedendo una RNA- o una DNA-polimerasi e permettendo loro di accedere a un filamento isolato che viene usato come stampo. Il loro moto lungo il DNA (500÷1000 basi al secondo) è rapido e processivo e utilizza l'energia derivata dall'idrolisi di ATP per destabilizzare i legami idrogeno che tengono unita la doppia elica del DNA. Sebbene la funzione biologica di questi enzimi non sia quella di produrre movimento e forza, di fatto essi si spostano vettorialmente lungo percorsi costituiti da filamenti di DNA in un modo simile al movimento delle chinesine, delle miosine e delle dineine. Come i motori, le polimerasi e le elicasi devono spostarsi rapidamente per svolgere le loro funzioni. Oltre alla velocità richiesta, le polimerasi devono produrre una forza sufficiente a opporsi a una forza contraria, dovuta al superavvolgimento del DNA e necessaria per spostare le proteine associate allo stampo. Nonostante questi ostacoli, l'apparato per la replicazione di Esterichia coli è in grado di spostarsi lungo il DNA in vivo alla velocità di circa 50.000 coppie di basi (17 μm) al minuto, un valore paragonabile a quello della chinesina lungo i microtubuli. Lo studio delle polimerasi e delle elicasi con tecniche biofisiche sviluppate per le ricerche sulla miosina e la chinesina è un approccio promettente per la comprensione dei meccanismi di questi enzimi. È stata misurata la forza generata da una polimerasi: nell'esperimento di Hong Yin la RNA-polimerasi di E. coli, adsorbita su una superficie di vetro, si spostava lungo uno stampo di DNA che era attaccato a un grano di polistirene, in una trappola ottica simile a quella usata per una singola molecola di miosina. L'RNA-polimerasi poteva esercitare una forza fino a 14 piconewton, il doppio dei valori massimi osservati per la chinesina e la miosina. Tuttavia l'efficienza termodinamica complessiva risultava più bassa.
Processi motori alimentati dalla polimerizzazione
I microtubuli e i filamenti di actina non sono dei semplici binari immobili su cui si muovono le proteine motrici. Al contrario, entrambi vanno incontro, nello spazio, a riarrangiamenti dinamici, nell'orientamento e nella concentrazione. Le forze necessarie per generare questi riarrangiamenti sono prodotte, in molti casi, dalle proteine motrici, e sarebbe interessante sapere se i filamenti stessi siano in grado di funzionare da generatori di forza. Actina e tubulina possono, almeno in teoria, produrre forza e movimento attraverso il processo di polimerizzazione. Una questione ancora aperta è se nella cellula si verifichino processi alimentati dalla polimerizzazione.
Spermatozoi dei Nematodi
Il miglior esempio di movimento indotto dalla polimerizzazione è quello degli spermatozoi del nematode Ascaris lumbricoides. Le cellule spermatiche di questo parassita dell'uomo strisciano invece di nuotare estendendo la membrana plasmatica in avanti rispetto al corpo cellulare; queste estroflessioni (pseudopodi) esercitano una forza di trazione che trascina il resto della cellula. Gli pseudopodi sono pieni di filamenti di una proteina specifica detta MSP (Major sperm protein); i monomeri di MSP polimerizzano in filamenti all'altezza del margine degli pseudopodi che si sposta in avanti e si depolimerizzano vicino al corpo cellulare. Una volta assemblati, i filamenti stessi rimangono stabili rispetto al substrato su cui si sposta la cellula, così che la velocità motoria del margine anteriore dello pseudopodio corrisponde al tasso di polimerizzazione (ca. 1 μm/s). L'idrolisi dell'ATP è accoppiata al ciclo polimerizzazione-depolimerizzazione della proteina MSP e ciò rende il processo esoergonico. Si può ipotizzare un meccanismo in cui sia l'aggiunta di monomeri di proteina MSP a dare la spinta. In altri eucarioti, che presentano lo stesso tipo di movimento, processi analoghi sono l'estensione del lamellipode, dipendente da actina, e i coni di crescita neuronali. In queste strutture, la polimerizzazione dell'actina si verifica sotto la membrana dell'estremità che avanza. Tuttavia, a differenza dei filamenti della proteina MSP presenti negli pseudopodi degli spermatozoi dei Nematodi, i filamenti di actina non sono stazionari rispetto al substrato, ma fluiscono all'indietro verso il corpo cellulare.
La motilità del batterio Listeria monocytogenes
Listeria monocytogenes è un batterio patogeno che invade il citoplasma della cellula ospite. Per spostarsi all'interno del citoplasma dell'ospite e per raggiungere cellule vicine, esso induce, a una sua estremità, la polimerizzazione dell'actina della cellula ospite e la formazione di legami casuali tra i filamenti generati. Il risultato è che il batterio viene spinto in avanti da una struttura a 'coda di cometa' di filamenti di actina e da proteine che incrociano trasversalmente i filamenti, analogamente a quanto descritto per gli spermatozoi dei Nematodi. Sembra che l'unica componente necessaria per il movimento di L. monocytogenes sia una proteina chiamata 'actA', poiché la sua espressione in batteri assai diversi è in grado di generare una motilità del tutto simile a quella di Listeria.
L'accoppiamento della depolimerizzazione al movimento
I precedenti esempi hanno evidenziato la presenza di forze che agiscono spingendo, grazie al processo di polimerizzazione dei filamenti, ma si possono generare forze che tirano mediante il processo di depolarizzazione. Questa ipotesi è stata spesso evocata per rendere conto del meccanismo di spostamento dei cromosomi durante la mitosi. È stato osservato che, nelle condizioni in cui è favorita la depolimerizzazione dei microtubuli, il cromosoma si mantiene attaccato all'estremità che depolimerizza e, di conseguenza, viene trascinato verso l'estremità opposta del microtubulo in fase di accorciamento. Tuttavia, rimane motivo di controversia se nelle cellule in vivo la depolimerizzazione sia, essa stessa, la causa del movimento cromosomico all'anafase.
Bibliografia
Desai, Mitchison 1995: Desai, Arshad - Mitchison, Timothy J., A new role for motor proteins as couplers to depolymerizing microtubules, "Journal of cell biology", 128, 1995, pp. 1-4.
Huxley, Hanson 1954: Huxley, Hugh E. - Hanson, Jean, Changes in the cross-striations of muscle during contraction and stretch and their structural interpretation, "Nature", 173, 1954, pp. 973-976.
Larsen 1974: Larsen, Steven H. e altri, Chemomechanical coupling without ATP: the source of energy for motility and chemotaxis in bacteria, "Proceedings of the National Academy of Sciences USA", 71, 1974, pp. 1239-1243.
Vale 1985: Vale, Ronald D. - Reese, Thomas S. - Sheetz, Michael P., Identification of a novel force-generating protein, kinesin, involved in microtubule-based motility, "Cell", 42, 1985, pp. 39-50.
Vale 1995: Vale, Ronald D. e altri, Direct observation of single kinesin molecules moving along microtubules, "Nature", 380, 1995, pp. 451-453.
Yin 1995: Yin, Hong e altri, Transcription against an applied force, "Science", 270, 1995, pp. 1653-1657.