ribosoma
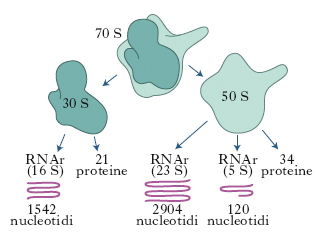
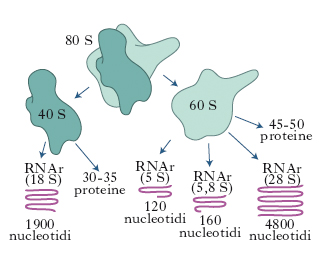
In biologia, particella del citoplasma cellulare, contenente RNA ribosomiale (RNAr) e diverse proteine. Messi per la prima volta in evidenza al microscopio elettronico nel 1953, da G.E. Palade, i r. svolgono un ruolo chiave nella sintesi proteica. Il loro numero varia da 20.000 a 50.000 a seconda di quanto la cellula è attivamente impegnata nella sintesi proteica e hanno un diametro di 250-350 nm. Nelle cellule eucariotiche i r. possono essere liberi nel citoplasma o attaccati al margine esterno delle membrane che costituiscono il reticolo endoplasmatico (➔ reticolo). I r. dei batteri hanno un coefficiente di sedimentazione pari a 70 unità Svedberg (70S). Sono composti da due subunità, una piccola (30S) e una grande (50S). La subunità 30S contiene una molecola di RNAr (16S) composta da 1542 nucleotidi e da 21 proteine differenti; la subunità 50S contiene due molecole di RNAr formate rispettivamente da 2904 nucleotidi (23S) e da 120 nucleotidi (5S) associate a 34 differenti proteine (fig. 1). Nelle cellule animali e in quelle vegetali i r. hanno un coefficiente di sedimentazione di 80S e sono costituiti da subunità 40S e 60S. La subunità piccola contiene un RNAr (18S) composto da 1900 nucleotidi e 30-35 proteine; la subunità grande contiene tre catene di RNA 5S, 5,8S e 28S formate rispettivamente da 120, 160 e 4800 nucleotidi (fig. 2). R. più piccoli e con un minor numero di RNA e proteine sono presenti anche nei cloroplasti e nei mitocondri.
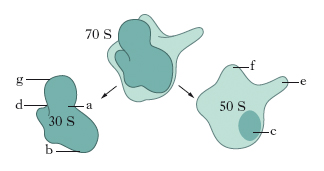
Le subunità maggiore e minore sono state analizzate al microscopio elettronico e hanno rivelato caratteristiche strutturali simili sia nei batteri sia nelle cellule animali e vegetali. La subunità minore presenta una testa e un corpo connessi da un collo più sottile; il collo presenta un avvallamento da una parte e una profonda fenditura dall’altra. Le subunità maggiori hanno la forma di una barca con l’estremità più appuntita segnata da una protuberanza centrale e da un peduncolo. La lieve curvatura della subunità minore determina la formazione di una regione pianeggiante sulla superficie, chiamata piattaforma (fig. 3). Questa superficie è localizzata in corrispondenza di una depressione sulla subunità maggiore. Si forma così un solco che presumibilmente accoglie l’RNAm durante la traduzione. Mediante la diffrazione ai raggi X è stato anche possibile osservare la presenza di un canale di circa 10 nm di lunghezza e 24 nm di diametro all’interno della subunità maggiore; probabilmente attraverso di esso passa la catena polipeptidica neosintetizzata che fuoriesce dal ribosoma.
Le singole proteine e gli RNA dei r. sono tenuti insieme da legami non covalenti, principalmente legami idrogeno. Ciò permette di disaggregare i r. con metodi relativamente blandi che, almeno per i procarioti, non determinano grosse alterazioni della struttura dell’RNA e delle proteine ribosomiali. È anche possibile ricostruire in vitro subunità ribosomiali perfettamente funzionali e identiche al tipo nativo, mescolando tra loro tutte le componenti in condizioni ambientali appropriate. Ciò significa che l’informazione necessaria all’assemblaggio di questo complesso è intrinseca alla struttura stessa delle sue parti.
Lo studio della organizzazione strutturale dei r. ha rivelato che le proteine ribosomiali interagiscono in modo cooperativo; non sono state individuate infatti reazioni condotte da una sola proteina. In alcune reazioni, per es. nel legame dell’amminoacil-tRNA e dei fattori di allungamento, le proteine di entrambe le subunità cooperano, costituendo i siti ribosomiali A e P (➔ proteine). Anche gli RNAr partecipano attivamente a molte reazioni della sintesi proteica mentre un tempo si pensava avessero solo una funzione strutturale nell’assemblaggio delle proteine ribosomiali. Numerose evidenze sperimentali infatti accreditano l’ipotesi che i r. siano degli enormi e molto complessi superenzimi ribonucleoproteici che presentano una grande quantità di siti attivi determinati dalla cooperazione fra le proteine ribosomiali e gli RNAr e che gli stessi RNAr possano avere un’attività catalitica.