nucleici, acidi
Molecole complesse di grandi dimensioni attraverso le quali avviene la trasmissione dell’informazione genetica. Sono presenti nelle cellule di tutti gli organismi (animali e vegetali), dai più semplici procarioti ai più complessi eucarioti, sotto forma di acido desossiribonucleico, DNA (deoxyribonucleic acid) e di acido ribonucleico, RNA (ribonucleic acid).
Il primo è il depositario dei caratteri ereditari contenuti nelle cellule e si trasmette alle cellule figlie per autoduplicazione; gli acidi ribonucleici hanno la funzione di tradurre morfologicamente l’informazione genetica contenuta nel DNA.
Struttura del DNA
Gli acidi n. sono formati da basi puriniche [adenina (A) e guanina (G)] e pirimidiniche [citosina (C), timina (T) e uracile (U)], da un carboidrato a 5 atomi di carbonio (ribosio o desossiribosio) e da acido ortofosforico. Il DNA contiene desossiribosio, mentre l’RNA contiene ribosio. Inoltre, nel DNA è presente la timina, mentre nell’RNA, al posto della timina, si trova l’uracile. In alcuni tipi di DNA e di RNA, oltre alle basi già citate, possono essere presenti derivati metilati. L’unità fondamentale degli acidi n. è il nucleotide, che è formato dall’unione di una base azotata con il pentosio, il quale a sua volta è esterificato dall’acido fosforico.
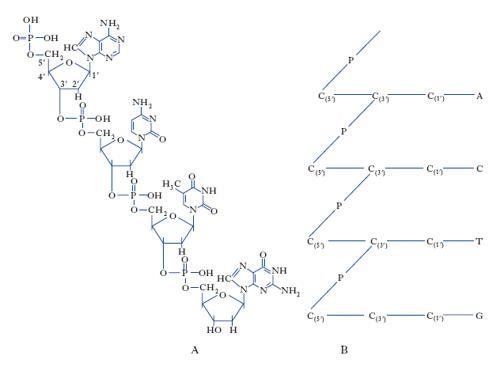
Gli acidi n. sono dei polinucleotidi: i vari nucleotidi sono attaccati l’uno all’altro mediante il radicale fosforico che si lega a una funzione alcolica del carboidrato del nucleotide adiacente con il legame tipico degli esteri. Le posizioni del pentosio coinvolte nel legame suddetto con il radicale fosforico sono quelle corrispondenti agli atomi di carbonio 3′ e 5′; l’atomo di carbonio in posizione 1′ è impegnato, invece, nel legame con la base azotata. Nella fig. 1 è riportata la struttura di una porzione di acido n. con a fianco la relativa rappresentazione schematica.
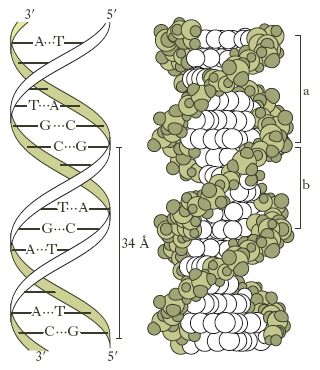
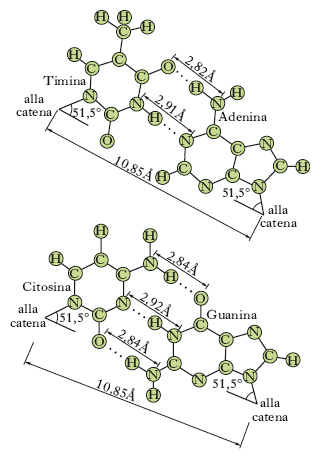
Mediante studi con la diffrazione dei raggi X, J.D. Watson e F.H.C. Crick hanno descritto nel 1953 la struttura tridimensionale del DNA, meritando per queste ricerche il premio Nobel per la fisiologia o la medicina nel 1962. A eccezione di alcuni virus nei quali il DNA è formato da un’unica catena, in tutti gli altri organismi esso è costituito da due catene di polinucleotidi che formano insieme una doppia elica. Queste catene hanno polarità opposta, sono cioè antiparallele; il loro scheletro è rappresentato da residui di desossiribosio alternati a residui fosforici, mentre le basi azotate, affacciate all’interno della catena perpendicolarmente all’asse, contribuiscono a mantenere rigida questa struttura mediante la formazione di legami idrofobici tra i nuclei delle purine e delle pirimidine e di legami idrogeno (fig. 2). Di questi ultimi, se ne stabiliscono due tra l’adenina e la timina e tre tra la citosina e la guanina, nel modo indicato nella fig. 3. Questo specifico accoppiamento di basi costituisce la caratteristica principale della struttura tridimensionale del DNA. La doppia elica è una struttura regolare: contiene 10 coppie di basi per ogni spira, per una lunghezza di 34 Å, in modo tale che tra due successive coppie di basi vi è una distanza di 3,4 Å. Lo scheletro formato da molecole di desossiribosio e da radicali fosforici dista dall’asse dell’elica 10 Å. Il peso molecolare del DNA si aggira intorno ai 6-12 milioni, valore corrispondente alla presenza di 20.000-40.000 nucleotidi.
La complementarità delle basi azotate fa sì che il rapporto A/T e G/C sia sempre uguale a uno. Quello che varia nelle diverse specie è il rapporto (A+T)/(G+C). Si è così osservato che in una stessa specie il rapporto tra le basi, e quindi la composizione del DNA, è costante, non dipende dal tessuto da cui il DNA viene estratto e non viene modificato dall’età, dalle condizioni di crescita e dai diversi fattori ambientali. Inoltre, le diverse specie di uno stesso genere hanno una composizione in basi molto simile. A eccezione di alcuni virus che contengono solo RNA, il DNA è presente in tutte le cellule ed è localizzato nel nucleo, dove, in associazione con RNA e proteine forma i cromosomi (➔ istoni). Nei batteri e in alcune alghe, in cui è assente un vero e proprio nucleo, il materiale cromosomico si trova nel citoplasma. Anche nei cloroplasti delle piante verdi e nei mitocondri è presente DNA (➔ mitocondrio).
Duplicazione del DNA
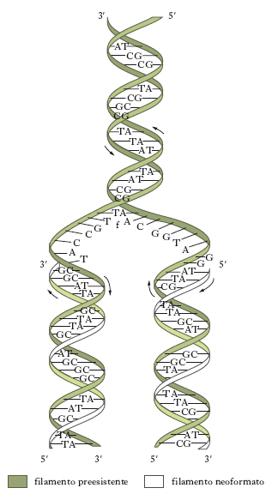
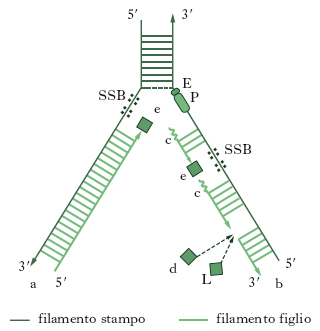
Per rottura dei legami idrogeno, le due eliche complementari si separano e ciascuna di esse funge da stampo per la formazione di un nuovo filamento (duplicazione semiconservativa, fig. 4). La replicazione del DNA inizia in punti specifici del cromosoma chiamati origini di replicazione e si estende in due opposte direzioni. I virus e i batteri hanno una o poche origini di replicazione mentre gli eucarioti ne hanno diverse in ogni cromosoma. I maggiori dettagli sulla sintesi di DNA sono stati studiati nelle cellule batteriche.
Nella prima tappa del ciclo di replicazione alcuni enzimi iniziatori aggregati in una struttura chiamata primosoma riconoscono la sequenza nucleotidica di origine; successivamente l’RNA polimerasi sintetizza corte sequenze di ribonucleotidi dette innesco o primer.
Nella seconda fase della duplicazione la DNA-polimerasi III, partendo dai desossiribonucleotidi trifosfati presenti nella cellula, sintetizza la nuova elica di DNA aggiungendo nucleotidi trifosfati all’estremità 3−OH del primer. Dato che la reazione chimica catalizzata dalla DNA-polimerasi permette a un nucleotide trifosfato di reagire solamente con l’estremità 3−OH libera di un frammento polinucleotidico preesistente, si dice che l’accrescimento della catena neosintetizzata avviene in direzione 5′-3′. L’elica figlia è sintetizzata in maniera continua in una direzione (filamento guida), mentre l’altra elica è sintetizzata in direzione opposta e in maniera discontinua (filamento lento).
Nella fase finale, il primer è degradato e una polimerasi I inserisce nuovi nucleotidi nei varchi fra i frammenti. L’enzima ligasi catalizza infine il legame fra le estremità 3′ e 5′ libere e chiude definitivamente il varco. Dalle cellule batteriche sono state isolate altre proteine coinvolte nella replicazione del DNA: le elicasi, le proteine SSB (single strand binding proteins, proteine che legano il singolo filamento) e le topoisomerasi. Le elicasi aumentano la velocità di separazione della doppia elica a livello della forcella di replicazione e rendono rapidamente accessibili nuove regioni a filamento singolo che sono subito riconosciute e coperte da una grande quantità di proteine SSB. Esse rendono il DNA a singolo filamento rigido, senza piegature o distorsioni, adatto quindi alla sua funzione di stampo per l’elica complementare (fig. 5). Lo svolgimento della doppia elica introduce però tensioni nella molecola; le topoisomerasi sono in grado di effettuare tagli transitori a monte della forcella di replicazione in modo da srotolare parzialmente la doppia elica e allentare la tensione. La riproduzione dell’informazione genetica contenuta nel DNA deve essere estremamente fedele, pena l’introduzione di mutazioni a ogni ciclo di duplicazione. Se viene aggiunta una base sbagliata alla estremità 3′ della catena nascente, essa ha un’alta probabilità di essere rimossa, prima che venga aggiunta la base successiva. Le polimerasi hanno infatti anche la capacità di tagliare, tornando indietro, la catena polinucleotidica nascente. Questa attività esonucleasica delle DNA polimerasi costituisce la ‘correzione di bozze’ che determina l’alta fedeltà dell’intero processo. Esistono inoltre enzimi che riparano le catene danneggiate, per esempio, da raggi ultravioletti o dai raggi X (➔ riparazione).
Acidi ribonucleici
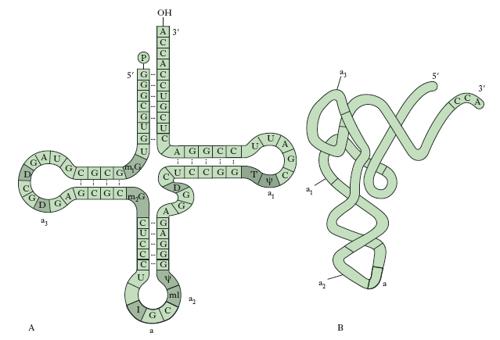
Vi sono tre tipi di RNA comuni a tutti gli organismi: RNAr (ribosomiale) che rappresenta il 45-50% del peso secco dei ribosomi dei Mammiferi; RNAm (messaggero) che contiene l’informazione per la sintesi delle proteine, RNAt (transfer) necessario per la traduzione nei ribosomi. L’RNAm è sintetizzato nel nucleo sullo stampo del DNA, ma esplica la sua funzione nel citoplasma. Ha una composizione in basi complementare a quella del DNA della stessa cellula, ma è formato da un’unica catena polinucleotidica. La funzione dell’RNAm è quella di raccogliere l’informazione per la struttura primaria di una proteina, contenuta in un tratto di DNA, e di trasferirla nel citoplasma sulla sede della sintesi proteica (➔ proteina). L’RNAt è distribuito principalmente nel citoplasma delle cellule e ha un peso molecolare di circa 30.000. Ogni molecola di RNAt ha una conformazione tridimensionale ripiegata con tratti a doppia elica e tratti a elica singola, determinati dalla presenza di basi insolite che non permettono la formazione di legami idrogeno. La molecola è stabilizzata dalle stesse interazioni chimiche deboli presenti nel DNA, ma nell’RNA le interazioni si verificano fra nucleotidi della stessa catena. L’avvolgimento della catena di RNAt è importante per la sua funzione di molecola adattatrice, capace di combinarsi con gli amminoacidi e di trasportarli ai ribosomi, dove per interazione con l’RNAm, avviene la sintesi delle catene polipeptidiche. Come mostrato in fig. 6, quattro segmenti della catena contengono una struttura a doppia elica ma, da un punto di vista funzionale, quelli che più contano sono i tre nucleotidi non accoppiati a ciascuna estremità della molecola: a una estremità è presente una tripletta di nucleotidi di sequenza variabile, l’anticodone, destinato a legarsi alla tripletta complementare di una molecola di RNAm, mentre la tripletta sita all’altra estremità (estremità 3′) presenta sempre la stessa sequenza di basi, citosina, citosina, adenina (CCA), e si lega covalentemente a uno specifico amminoacido. Questo legame avviene con l’apporto energetico di molecole di ATP e la sua specificità è assicurata dalle amminoaciltransferasi che uniscono ogni amminoacido al corrispondente RNAt.
Oltre alle molecole di RNA coinvolte nella sintesi delle proteine, ne sono state scoperte numerose altre che costituiscono una parte integrante di importanti funzioni cellulari. Molecole di RNA, per es., sono parte essenziale della telomerasi che funge da stampo per la replicazione dei telomeri e delle particelle SRP, necessarie al trasporto delle proteine nella cellula (➔ reticolo). Alcune molecole di RNA hanno funzione di catalizzatori (➔ ribozima); altre possono avere una funzione di metilazione di specifici siti dell’RNAt, altre ancora, con una funzione simile alle chaperonine, sono chiamate snoRNA (small nucleolar RNA). Alcune molecole, sotto forma di piccoli RNA nucleari (snRNA, small nuclear RNA), costituiscono le particelle ribonucleoproteiche dello spliceosoma, struttura nella quale avviene lo splicing. Molecole di RNA, dette RNAg, servono come sequenza guida per l’editing dell’RNAm dei tripanosomi, considerato un modo primitivo per modificare l’espressione dei geni. Infine, molecole di RNA sembrano avere un ruolo essenziale nei processi epigenetici (➔ epigenesi) di regolazione dei geni: l’RNA codificato dal gene XIST (X inactive specific transcript) ha infatti un ruolo nel processo di inattivazione di uno dei due cromosomi X durante lo sviluppo; gli studi svolti alla fine degli anni 1990 hanno inoltre dimostrato il ruolo essenziale delle molecole di RNA nei processi di silenziamento dei geni in vari organismi.