mappa
Nell’antichità classica, panno, generalmente di lino, usato sia come tovagliolo, sia come acconciatura femminile. Gli antichi agronomi chiamarono m. (perché spesso eseguite su tela) ogni rappresentazione grafica di una zona di terreno; di qui l’uso moderno della parola, che in topografia serve a indicare la rappresentazione grafica di una zona di terreno, in cui la scala di riduzione è superiore a 1:10.000, e per estensione è riferito a vari tipi di rappresentazione.
Biologia
In genetica, rappresentazione schematica dei geni su un cromosoma e delle loro distanze relative.
M. dei geni umani
La localizzazione dei geni sui cromosomi umani ha impegnato i laboratori scientifici di tutto il mondo e ha portato nel 2001 alla conoscenza delle sequenze complete del DNA del genoma umano (➔ genoma). Il paragone fra le m. genetiche o m. di associazione (linkage), le m. citologiche o citogenetiche e le m. fisiche, basate sull’analisi diretta delle molecole di DNA, costituisce un punto di partenza importante sia per la localizzazione dei geni ancora sconosciuti, compresi quelli di malattie ereditarie umane, sia per lo studio delle modalità di espressione dei geni, cioè per la genomica funzionale (➔ genoma).
M. di associazione
Si basano sull’analisi statistica (➔ lod score) della frequenza con la quale un marcatore genetico e un gene da identificare vengono ereditati insieme. Un marcatore genetico può essere un qualunque sito nel genoma, detto sito polimorfico, dove esistono variazioni nella sequenza del DNA che possono essere evidenziate con metodi quali il Southern blotting o la PCR (➔ blotting e PCR). Le variazioni si osservano soprattutto nelle sequenze di DNA non codificante, in quanto sono generalmente poco conservate nel corso dell’evoluzione. Tali variazioni possono essere dovute o alla sostituzione di una base nella sequenza del DNA o a un numero diverso di ripetizioni di tratti più o meno lunghi di DNA. I marcatori polimorfici di prima generazione sono i polimorfismi della lunghezza dei frammenti di restrizione (➔ RFLP), ma, con l’avvento della PCR, è stato possibile evidenziare altre sequenze polimorfe, costituite da brevi ripetizioni testa-coda di sequenze dinucleotidiche dette microsatelliti o STRP (short tandem repeat polymorphisms). I microsatelliti si sono dimostrati utili come marcatori per costruire m. dei cromosomi umani; nel 1996, è stata pubblicata una m. comprendente 5264 microsatelliti presenti nel genoma umano. Essi definiscono 2335 siti con una distanza media di 1,6∙106 paia di basi tra i marcatori adiacenti. La distanza notevole degli STRP pone tuttavia un limite al loro uso. In conseguenza dei progressi ottenuti con il sequenziamento del genoma viene contemplata una terza generazione di marcatori polimorfici, gli SNP (single nucleotide polymorphisms), che presentano variazioni di un solo nucleotide. Stime preliminari pongono un SNP ogni 1000 basi. Una volta identificati in grande numero, essi potrebbero rappresentare i marcatori migliori per gli studi futuri di mappatura genica.
Qualsiasi sia il marcatore usato, per costruire una m. di associazione si assume che, se il gene da localizzare e il marcatore sono sullo stesso cromosoma, vengono ereditati insieme; la probabilità di essere ereditati insieme è tanto maggiore quanto più gene e marcatore sono vicini sul cromosoma e quindi hanno scarsa possibilità di scambio (crossing-over) fra loro. Controllando gli alberi genealogici di grandi gruppi familiari per l’eredità di un gene che, se mutato, determina una malattia e un gran numero di marcatori, si possono identificare tutti i marcatori ereditati insieme al gene e la loro distanza. Una volta identificato, un marcatore può essere utilizzato come sonda per determinare il cromosoma sul quale è localizzato e per trovarne altri più strettamente associati al gene. Con varie tecniche si possono poi identificare, clonare e determinare la sequenza del gene, la sequenza amminoacidica della proteina da esso codificata e le mutazioni che causano la malattia ereditaria.
M. citogenetiche
Per mappare i geni umani vengono usate strategie diverse a seconda delle conoscenze che già si hanno sul gene che si vuole localizzare. La m. citologica o citogenetica permette di arrivare al risultato qualora a un determinato fenotipo mutato corrispondano sul cariotipo anomalie cromosomiche visibili al microscopio ottico, oppure si abbia a disposizione una sonda molecolare adeguata per effettuare sui cromosomi in metafase l’ibridazione in situ (➔ citogenetica e bandeggio). Si può avere a disposizione una sonda molecolare quando è nota la proteina codificata dal gene, in quanto dalla sequenza amminoacidica si può risalire, in base al codice genetico, alla sequenza del DNA corrispondente. Traslocazioni fra il cromosoma X e un autosoma hanno permesso, per es., di individuare la posizione del gene DMD responsabile della distrofia muscolare di Duchenne (➔ distrofia) e microdelezioni sul cromosoma 15 sono state utili per localizzare il gene della sindrome di Prader Willi (➔ imprinting). Sia le m. citogenetiche sia le m. di associazione individuano la zona dove il gene è localizzato, all’interno di 2-4 milioni di coppie (m. a bassa risoluzione). La m. fisica ha un alto potere di risoluzione, che va da 1 a 100.000 coppie di basi.
M. fisiche
La prima tappa della mappatura fisica richiede la separazione di singoli cromosomi o di grandi segmenti di essi. I cromosomi batterici o quelli di alcuni Eucarioti più semplici si possono separare mediante elettroforesi su gel in campo elettrico pulsante (PFGE, pulse-field gel electrophoresis), mentre i cromosomi di Eucarioti più complessi si separano attraverso apposite apparecchiature (➔ citometria). Il cromosoma così isolato si frammenta con un enzima di restrizione in molti segmenti e questi si clonano in vettori, generalmente BAC, YAC o cosmidi, per creare genoteche specifiche per ogni cromosoma, cioè una collezione di vettori, ciascuno dei quali reca un tratto di DNA di quel cromosoma. Bisogna poi determinare l’ordine con il quale i segmenti cromosomici clonati, si presentano in un cromosoma intatto. Tale collezione di cloni, detta contig (➔ contiguo), contiene un tratto continuo di cromosoma sotto forma di regioni sovrapponibili; per identificare i membri di un contig si possono identificare i membri adiacenti che contengono regioni sovrapponibili. Se un membro di un contig contiene un gene o un marcatore molecolare dei quali è già nota la posizione, esso può rappresentare il punto di partenza per l’allineamento dei vari frammenti. I marcatori usati sono sia RFLP sia brevi tratti di DNA (100-400 paia di basi) presenti una sola volta nell’ambito dell’intero genoma, chiamati siti di sequenze etichetta o STS (sequence tagged sites). Gli STS vengono identificati mediante PCR. I vari frammenti di un contig possono così essere sequenziati e in tal modo si può conoscere la sequenza nucleotidica, senza interruzioni, di vaste regioni cromosomiche.
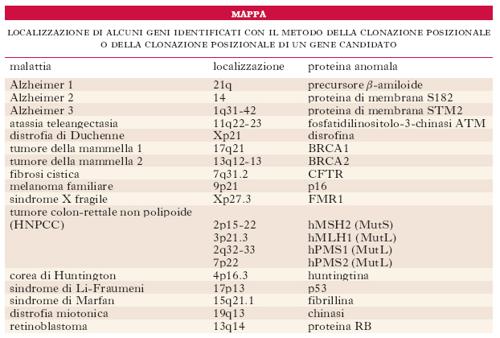
Clonazione dei geni-malattia umani
Le quattro strategie fondamentali per isolare i geni umani che, se mutati, determinano malattie sono la clonazione funzionale, la clonazione di un gene candidato, la clonazione posizionale e la clonazione posizionale di un gene candidato (v. tab.). Per scoprire i geni si può impiegare un’ampia varietà di strumenti (la PCR, i marcatori del genoma, l’ibridazione) e di risorse (le m. fisiche, le librerie di cDNA, le librerie genomiche e i database di sequenze geniche). La dimostrazione definitiva che un gene è coinvolto in una determinata malattia ereditaria si basa comunque sull’identificazione, negli individui affetti, di mutazioni assenti nei geni degli individui non affetti. Per questo scopo sono stati ideati numerosi metodi (➔ mutazione).
La clonazione funzionale può essere usata solo se la proteina codificata dal gene è nota. La sequenza amminoacidica della proteina viene usata per sintetizzare in vitro oligonucleotidi che serviranno per riconoscere frammenti di DNA di una genoteca di cDNA. Una volta isolato il primo frammento di DNA, esso può essere utilizzato a sua volta come ulteriore sonda per conoscere e isolare la completa sequenza del gene in una genoteca genomica. Un saggio di identificazione di mutazioni stabilisce poi se il gene sia responsabile di una malattia genetica. Con il metodo della clonazione funzionale è stato, per es., mappato il gene che codifica la β-globina, il quale, quando è mutato, può dare origine a diverse emoglobinopatie.
Nel caso della clonazione di un gene candidato, si parte da un gene del quale si conosce la sequenza ma non la funzione e che, a causa della sua posizione o di qualche altra proprietà, si ritiene implicato in una malattia genetica; un saggio di identificazione di mutazioni deve stabilire se questa ipotesi è corretta.
La clonazione posizionale viene attuata invece quando non si ha alcuna informazione sulla proteina che causa una malattia genetica. In questo caso per mappare un gene che, se mutato, determina la malattia, si possono usare m. citogenetiche o di associazione. Si stabilisce quali marcatori polimorfici sono associati al gene; si usano poi sonde molecolari che riconoscono questi siti polimorfici per clonare il DNA della zona di interesse. Se la zona è grande le tecniche sperimentali usate per identificare il gene sono complesse. Si usa, per es., il walking cromosomico (➔) per identificare il punto preciso dove è situato il gene o se un ricercatore ha già creato un contig nella regione del cromosoma nella quale è localizzato il gene di interesse, si possono analizzare i singoli cloni del contig. Per stabilire se in un determinato clone sono presenti sequenze del gene (gli esoni) di interesse si ricorre, per es., all’osservazione della presenza di eventuali isole CpG (➔ metilazione), all’ibridazione con il DNA di specie diverse o zooblotting (➔), all’analisi computerizzata delle molecole di DNA sequenziate per scoprire eventuali omologie con sequenze note.
A questo scopo ha notevolmente contribuito la scoperta di sequenze espresse di DNA particolari e riconoscibili definite sequenze etichetta espresse o EST (expressed sequence tags), che sono brevi sequenze uniche di cDNA generate da mRNA di specifici tessuti umani. Mediante la conoscenza della posizione sul genoma di EST note, che fungono da punti di riferimento, è possibile costruire una m. di molti o di tutti i geni di una specifica regione di un cromosoma. Queste informazioni permettono di effettuare la clonazione posizionale di un gene candidato, utilizzata per l’identificazione di geni responsabili di malattie monogeniche. Questo è un metodo composito che implica la mappatura di un gene-malattia con funzione sconosciuta in una posizione cromosomica, e poi l’esame di aggiornate m. genetiche (geni) e trascrizionali (EST) per determinare quali regioni codificanti siano state mappate nello stesso intervallo del gene-malattia.
Fisica
M. di campo
Rappresentazione grafica delle superfici equipotenziali (➔ potenziale) di un campo elettrico, magnetico, gravitazionale ecc.
Matematica
Nella teoria degli insiemi, sinonimo di applicazione, con riferimento a insiemi dotati di strutture algebriche, sinonimo di morfismo (➔ categoria).
In topologia è usato a proposito di applicazioni biunivoche e bicontinue f tra due spazi topologici S e S′: se f applica l’aperto A di S nell’aperto A′ di S′, A′ si dice m. di A nella f.
Medicina
In elettrocardiografia, m. epicardica, l’insieme dei tracciati che si ottengono dalla registrazione simultanea dell’elettrocardiogramma epicardico ottenuta dall’applicazione di numerosi elettrodi direttamente sulla superficie del cuore, in corso di intervento chirurgico.
In neurologia, m. cerebrali elettriche rappresentazione della distribuzione del potenziale cerebrale con riferimento sia all’attività elettroencefalografica di base sia ai potenziali correlati a eventi esterni ben definiti. Accanto alle m. temporali, nello studio dei potenziali cerebrali vengono utilizzate anche m. in frequenza, principalmente per l’analisi dell’EEG. In tal caso ogni m. è relativa a una frequenza, o intervallo di frequenza, e a ogni punto della m. stessa è associato il livello di ampiezza o di energia relativo a quella frequenza. Poiché la costruzione di tali m. avviene con l’ausilio di un elaboratore elettronico, è possibile ottenere m. derivanti da elaborazioni di vario tipo; fra queste, quelle di maggiore interesse diagnostico risultano le m. statistiche, che evidenziano le regioni sul cuoio capelluto nelle quali, confrontando due popolazioni di individui (o un individuo e una popolazione), emergono differenze significative. La tecnica delle m. elettriche cerebrali è stata utilizzata sia nell’indagine fisiologica sia a scopo diagnostico per individuare la presenza di alterazioni patologiche (di tipo neoplastico o di altra natura, in corso di dislessia, morbo di Parkinson). Le m. cerebrali magnetiche, ossia le m. relative ai campi magnetici cerebrali, utilizzano tecniche di elaborazione sostanzialmente analoghe a quelle per le m. elettriche; la differenza maggiore risiede nella strumentazione necessaria per la determinazione del campo magnetico, rappresentata da particolari magnetometri, gli SQUID (➔), talmente sensibili da poter determinare valori del campo magnetico cerebrale, che sono estremamente bassi. La determinazione dei campi magnetici cerebrali risulta particolarmente utile per localizzare le sorgenti profonde dei campi rilevati; infatti, poiché il cranio è ‘trasparente’ al campo magnetico, il campo rilevato non presenta quelle distorsioni che si verificano nel caso del campo elettrico.
Storia
Dal 1° sec. m. particolarmente ricche, colorate, purpuree, ricamate, si ponevano sulle tavole accanto al piatto; nei palazzi imperiali c’era uno speciale servizio a mappis per la confezione e custodia di esse.
Con mappae rosse i bestiari nel circo eccitavano gli animali e il segnale dell’inizio delle corse era dato dall’imperatore o dal magistrato gettando la m. nell’arena (mittere mappam): due statue oggi nel museo dei Conservatori a Roma raffigurano magistrati del 4° sec. d.C. in questo atto.
Con l’affermarsi del cristianesimo la m. entra nella liturgia o per avvolgere le mani di chi deve toccare arredi sacri, o per ricoprire l’altare.
Nel Basso Impero si chiamano mappae i libri lintei e gli editti scritti su rotoli di tela.