mutazione
Biologia
Variazione ereditaria della struttura del materiale genetico, spontanea o indotta da agenti mutageni fisici o chimici. Il termine fu usato da H. De Vries (1901), l’ideatore della teoria delle m. o mutazionismo. Egli riteneva che le m. fossero sempre rappresentate da deviazioni notevoli rispetto alla norma (forme nane e forme giganti, foglie strette e foglie larghe ecc.), così da cadere fuori della curva normale di variabilità. Secondo gli studi successivi sono considerate m. tutte le variazioni ereditarie del genotipo, qualunque sia l’ampiezza delle variazioni che esse determinano nel fenotipo.
Caratteri generali
Le m. di solito portano a un difetto funzionale: un gene mutato spesso codifica una proteina anormale o, talora, nessuna proteina. Non sempre però questo si verifica: talvolta la m. di un gene ha un effetto minimo o nullo sulla proteina codificata da quel gene o addirittura costituisce un vantaggio per l’organismo mutato. In quest’ultimo caso la frequenza delle m. utili può poi aumentare nella popolazione a causa della selezione naturale. In Drosophila melanogaster una m. che determina l’atrofia delle ali, di per sé deleteria, non risulta tuttavia dannosa per la popolazione di moscerini che vive sulle coste ventose della Bretagna in quanto, incapaci di volare, essi rimangono attaccati alla scogliera e non vengono portati via dal vento; nell’uomo, la m. che determina l’anemia falciforme è deleteria in omozigosi, ma, in eterozigosi, essa conferisce un vantaggio in quanto protegge dalla malaria. Gli individui portatori di tale m., in alcune zone dove la malaria è endemica, hanno una maggiore idoneità biologica rispetto agli individui normali (➔ fitness). Tutti i geni sono originati da una successione di eventi mutazionali. Le m. determinano variabilità genetica e costituiscono pertanto gli eventi fondamentali per l’evoluzione delle specie. Se il carico mutazionale però è eccessivo si può arrivare alla morte dell’individuo o all’estinzione della specie.
La frequenza di m. di un gene si calcola in base alla frequenza di neo-mutazioni per gamete nella popolazione in esame. Le m. sono classificate secondo il tipo di cambiamenti strutturali che esse comportano nel DNA e presentano tutte le dimensioni possibili senza soluzione di continuità. Esse insorgono spontaneamente o possono essere determinate da agenti mutageni fisici o chimici. Le m. consistono in cambiamenti di una singola coppia di basi (m. geniche o puntiformi) o di più basi che coinvolgono due o più geni (m. pluribase). Si chiamano m. anche le modificazioni o gli spostamenti nel genoma di frammenti di cromosomi (m. cromosomiche) o la perdita e l’aggiunta di interi cromosomi (m. genomiche).
M. puntiformi
Le m. puntiformi si classificano come sostituzioni, inserzioni o delezioni di basi. Le m. per sostituzione di basi sono dette transizioni quando una purina è sostituita da un’altra purina o una pirimidina è sostituita da un’altra pirimidina; sono dette trasversioni quando una purina è sostituita da una pirimidina o viceversa. Le sostituzioni sono causate da errati appaiamenti per cambiamenti tautomerici (➔ tautomeria) o per deamminazione delle basi. Il cambiamento di una base in una tripletta del DNA può determinare la sostituzione di un amminoacido con un altro nella proteina codificata dal gene mutato (m. di senso); per es., la sostituzione del sesto amminoacido (acido glutammico) con la valina nella β globina degli individui affetti da anemia falciforme. Se la sostituzione della base determina invece la formazione di una tripletta di terminazione del messaggio genetico (m. non senso) si ha l’interruzione prematura della catena polipeptidica perché la tripletta non codifica nessun amminoacido; per es., interruzione della sintesi della β globina negli individui affetti da una delle varie forme di talassemia o anemia mediterranea. Il cambiamento di una singola coppia di basi non comporta invece la sostituzione di un amminoacido nella catena polipeptidica se la tripletta modificata codifica lo stesso amminoacido (➔ codice). In questo caso il cambiamento può essere evidenziato solo facendo l’analisi della sequenza delle basi e non ha effetti sul fenotipo.
Le m. per inserzione o delezione di una coppia di basi non solo alterano la lettura del codone cui appartengono, ma producono uno scivolamento del modulo di lettura del codice genetico (➔ modulo). Come conseguenza, un gene mutato per delezione o inserzione codifica sempre una proteina anomala perché tutti gli amminoacidi codificati a valle della m. risultano cambiati.
È detta carico mutazionale (genetic load) la serie di m. geniche, specialmente di tipo letale o detrimentale-recessivo, accumulate nel tempo da una popolazione, che si ripresentano a bassa frequenza in tutte le generazioni.
M. pluribase
Le m. in cui sono alterate due o più coppie di basi si possono classificare in duplicazioni, delezioni, traslocazioni, inversioni.
Le duplicazioni comportano la ripetizione di un frammento di DNA una o più volte, di solito a coppie (tandem). Il genoma degli eucarioti contiene numerose serie di coppie di geni, per es.: i geni per gli istoni e i geni per le catene α e β dell’emoglobina. Le duplicazioni di geni hanno svolto un ruolo importante nell’evoluzione e talvolta rappresentano un vantaggio.
Le delezioni e le inserzioni pluribase comportano la perdita o l’aggiunta di poche basi o di centinaia o migliaia di basi. Per es., una piccola delezione di tre basi successive determina la fibrosi cistica nell’uomo; delezioni di grandezza variabile nel gene che codifica la distrofina, proteina normalmente presente nelle fibre muscolari, determinano la distrofia muscolare di Duchenne.
Nelle inversioni un frammento di DNA è ruotato di 180°, così che esso si trova orientato in direzione opposta. Questi cambiamenti non avvengono con una frequenza elevata eccetto che in particolari posizioni: normalmente segmenti ripetuti invertiti si trovano negli elementi mobili trasponibili negli eucarioti e nei procarioti (➔ elemento; trasposone).
La traslocazione consiste nel trasferimento di un segmento del genoma in un’altra posizione nello stesso cromosoma o in un altro.
M. cromosomiche o aberrazioni cromosomiche di struttura
I termini usati per descrivere le aberrazioni da riarrangiamento dei cromosomi sono gli stessi che vengono usati per descrivere le corrispondenti m. pluribase. La caratterizzazione delle m. cromosomiche viene effettuata osservando la conformazione dei cromosomi al microscopio ottico durante la mitosi e la meiosi. Le prime osservazioni, tuttavia, sono state fatte sui cromosomi giganti delle ghiandole salivari delle larve di Drosophila melanogaster.
La prima aberrazione cromosomica studiata fu una delezione del cromosoma X di Drosophila chiamata Notch, mutazione letale recessiva scoperta da C.B. Bridges nel 1917. Le femmine portatrici della delezione Notch hanno ali frastagliate (notched). La prima sindrome nell’uomo dovuta a una delezione cromosomica fu osservata, nel 1963, da J. Lejeune e da suoi collaboratori e chiamata cri du chat.
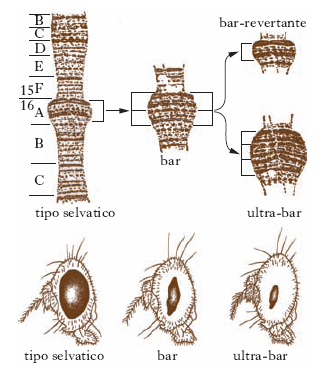
I cromosomi delle ghiandole salivari di Drosophila sono stati anche utili per caratterizzare le duplicazioni. Una riduzione anomala degli ommatidi dell’occhio di Drosophila è stata messa in relazione con una duplicazione in serie di un tratto di un cromosoma X (mutazione occhio Bar: fig. 1).
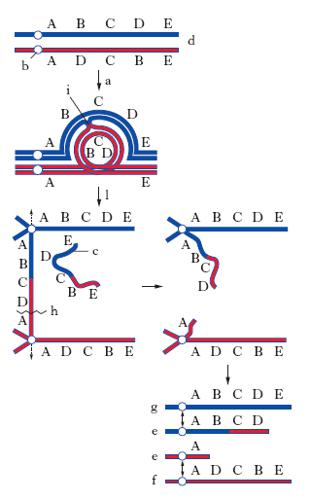
Le inversioni consistono invece nella presenza di un segmento cromosomico orientato in direzione opposta. Le inversioni pericentriche contengono il centromero nel segmento invertito; le inversioni paracentriche non lo contengono. L’appaiamento dei cromosomi omologhi alla meiosi negli individui eterozigoti per l’inversione deve avvenire in modo tale per cui il contatto tra le regioni omologhe dei cromosomi appaiati sia massimo: per questo motivo nell’appaiamento fra un cromosoma normale e uno invertito si formano figure ad ansa. Le inversioni determinano una drastica riduzione delle frequenze dei ricombinati se il crossing-over fra i due cromosomi omologhi appaiati si verifica nella regione dell’inversione: i gameti che contengono i cromosomi ricombinanti presentano duplicazioni e delezioni di geni (fig. 2).
La traslocazione consiste in uno scambio di segmenti fra cromosomi non omologhi: a speciali fattori genetici noti come elementi genetici trasponibili sono stati addebitati molti casi di traslocazione. Anche la traslocazione porta ad appaiamenti anomali alla meiosi. Nella specie umana le duplicazioni e le delezioni di tratti di cromosomi sono spesso incompatibili con la vita. Invece gli individui portatori di traslocazioni o di inversioni possono essere assolutamente normali ma, a causa dell’appaiamento anomalo e della conseguente segregazione errata dei cromosomi omologhi, alla meiosi possono produrre gameti con un contenuto genico sbilanciato e quindi risultare sterili, avere un’elevata frequenza di aborti spontanei o dare origine a individui con gravi anomalie. Le traslocazioni inoltre sono presenti nelle cellule somatiche degli individui affetti da forme di leucemie e tumori.
M. genomiche o aberrazioni cromosomiche di numero
Consistono nella perdita o nell’acquisizione di interi cromosomi. Quando si tratta di singoli cromosomi l’anomalia è nota come aneuploidia, quando è coinvolta l’intera serie dei cromosomi l’anomalia prende il nome di poliploidia.
L’aneuploidia deriva da non disgiunzione alla prima o alla seconda divisione meiotica. La non disgiunzione dà origine a gameti con un particolare cromosoma in quantità doppia o assente. Quando questi gameti anomali si uniscono a un normale gamete, nel corso della fecondazione, il risultato sarà una trisomia o una monosomia. Gravi difetti e persino la morte sono le conseguenze più frequenti di un’anomalia nel numero dei cromosomi: la forma più comune della sindrome di Down è prodotta dalla non disgiunzione e dalla risultante condizione trisomica per il cromosoma 21. Non disgiunzioni nei cromosomi sessuali alla meiosi determinano la nascita di individui affetti da sindrome di Turner (XO), Klinefelter (XXY), Triplo X, XYY ecc.
Il termine poliploidia viene comunemente usato per indicare un corredo cromosomico formato da un numero di serie cromosomiche superiore a quello diploide consueto (triploidia, tetraploidia ecc.). La poliploidia è più comune nelle piante che negli animali (in alcune felci il numero cromosomico supera il migliaio). In alcune piante la triploidia è la regola anziché l’eccezione. La poliploidia può insorgere nel tessuto germinale e nel tessuto somatico. Le cause più comuni della formazione di embrioni triploidi anomali sono la mancata divisione riduzionale dei cromosomi durante la gametogenesi (➔ meiosi) o la fecondazione plurima di un uovo da parte di più spermatozoi. La poliploidia nelle cellule somatiche si origina dalla non disgiunzione dei cromosomi durante la mitosi. Per es., nel fegato di mammifero è normale trovare cellule tetraploidi o persino ottoploidi: le cellule poliploidi si formano a causa di cicli incompleti di divisione perché la duplicazione dei cromosomi non è stata accompagnata dalla divisione cellulare (➔ mitosi). Alterazioni del numero e della struttura dei cromosomi sono una causa frequente di aborti spontanei.
Identificazione di m. in geni umani
Per identificare le m. si sono sviluppati negli anni 1990 numerosi saggi, fra i quali, il polimorfismo della conformazione a singolo filamento o SSCP (single-strand conformational polymorphism), l’elettroforesi su gel in gradiente denaturante o DGGE (denaturing gradient gel electrophoresis), il taglio chimico degli appaiamenti errati o CMC (chemical mismatch cleavage) e il test delle proteine troncate o PTT (protein truncation test). Tutti questi saggi si basano sull’amplificazione mediante PCR di campioni di DNA provenienti da individui affetti e da alcuni individui non affetti.
Nei primi tre citati le variazioni nucleotidiche si possono poi studiare in base alla differenza di migrazione dei campioni variamente trattati in una corsa elettroforetica. Per es., nel caso dell’analisi della conformazione a singolo filamento, che è uno dei procedimenti più usati, i prodotti della PCR vengono denaturati, raffreddati e sottoposti a elettroforesi. Ciascuna molecola di DNA denaturata a singolo filamento assume una conformazione tridimensionale che dipende dalla sua sequenza nucleotidica ed è determinata dalla formazione di legami fra le basi all’interno del filamento. Dato che le differenti conformazioni hanno una velocità di migrazione diversa, confrontando, nell’elettroforesi, il DNA proveniente dagli individui affetti e quello proveniente dagli individui non affetti si possono identificare le differenze nella migrazione delle molecole di DNA.
Il saggio SSCP localizza un’alterazione nucleotidica su una specifica regione o su uno specifico esone di un gene, ma non rivela la natura della m.; nei prodotti della PCR lunghi 200 paia di basi è in grado tuttavia di identificare circa il 90% delle m. di singole coppie di basi. Nel test delle proteine troncate invece i prodotti della PCR vengono trascritti e tradotti in vitro e la proteina sintetizzata viene marcata e identificata mediante diversi sistemi.
Il PTT è in grado di identificare m. non senso, cioè quelle che determinano nella sequenza nucleotidica una tripletta di stop e quindi interrompono prematuramente la sintesi di proteine, oppure delezioni o inserzioni più lunghe di 25 paia di basi. Se il gene non ha questo tipo di m. codificherà una proteina di lunghezza normale; se invece sono presenti delezioni maggiori di 25 paia di basi, m. non senso o m. del sito di splicing che determinano l’omissione di un esone, verrà prodotta una proteina accorciata. Questo saggio, come gli altri saggi di identificazione di m., delimita per lo più la posizione di una m. a un esone o a una regione vicina. L’effettiva alterazione nucleotidica viene poi sempre determinata mediante sequenziamento del DNA.
Nella genetica dei microrganismi, sono detti mutanti nutrizionali (o mutanti biochimici) quei ceppi che abbiano perduto, per m., la capacità di metabolizzare una sostanza indispensabile alla loro crescita e moltiplicazione. Le ricerche fondamentali sui mutanti nutrizionali negli Ascomiceti (Neurospora crassa) e sui Batteri (Escherichia coli) sono legate rispettivamente ai nomi di G.W. Beadle e E.L. Tautum e a quello di J. Lederberg.
Linguistica
Termine con cui è di solito tradotto, in alternanza con rotazione, il tedesco Lautverschiebung, per indicare i mutamenti subiti dal consonantismo delle lingue germaniche sia rispetto alle altre lingue sia rispetto al germanico più antico.